+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of the human CTF18-RFC-PCNA-DNA ternary complex with narrow PCNA opening state II (state 6) | |||||||||
 マップデータ マップデータ | EM sharpen map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA clamp loader complex / REPLICATION-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Elg1 RFC-like complex / DNA replication factor C complex / Ctf18 RFC-like complex / positive regulation of deoxyribonuclease activity / dinucleotide insertion or deletion binding / PCNA-p21 complex / mitotic telomere maintenance via semi-conservative replication / DNA clamp loader activity / purine-specific mismatch base pair DNA N-glycosylase activity / nuclear lamina ...Elg1 RFC-like complex / DNA replication factor C complex / Ctf18 RFC-like complex / positive regulation of deoxyribonuclease activity / dinucleotide insertion or deletion binding / PCNA-p21 complex / mitotic telomere maintenance via semi-conservative replication / DNA clamp loader activity / purine-specific mismatch base pair DNA N-glycosylase activity / nuclear lamina / positive regulation of DNA-directed DNA polymerase activity / Polymerase switching / Telomere C-strand (Lagging Strand) Synthesis / MutLalpha complex binding / Processive synthesis on the lagging strand / PCNA complex / Removal of the Flap Intermediate / Processive synthesis on the C-strand of the telomere / Polymerase switching on the C-strand of the telomere / Removal of the Flap Intermediate from the C-strand / Mismatch repair (MMR) directed by MSH2:MSH3 (MutSbeta) / Mismatch repair (MMR) directed by MSH2:MSH6 (MutSalpha) / Transcription of E2F targets under negative control by DREAM complex / replisome / HDR through Single Strand Annealing (SSA) / response to L-glutamate / Impaired BRCA2 binding to RAD51 / DNA strand elongation involved in DNA replication / histone acetyltransferase binding / DNA synthesis involved in DNA repair / DNA polymerase processivity factor activity / leading strand elongation / G1/S-Specific Transcription / response to dexamethasone / replication fork processing / nuclear replication fork / Presynaptic phase of homologous DNA pairing and strand exchange / SUMOylation of DNA replication proteins / PCNA-Dependent Long Patch Base Excision Repair / ATP-dependent activity, acting on DNA / mismatch repair / translesion synthesis / Activation of ATR in response to replication stress / response to cadmium ion / cyclin-dependent protein kinase holoenzyme complex / estrous cycle / base-excision repair, gap-filling / DNA polymerase binding / epithelial cell differentiation / positive regulation of DNA repair / male germ cell nucleus / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / Translesion synthesis by REV1 / Translesion synthesis by POLK / liver regeneration / Translesion synthesis by POLI / replication fork / Gap-filling DNA repair synthesis and ligation in GG-NER / positive regulation of DNA replication / nuclear estrogen receptor binding / Termination of translesion DNA synthesis / Recognition of DNA damage by PCNA-containing replication complex / Translesion Synthesis by POLH / G2/M DNA damage checkpoint / receptor tyrosine kinase binding / HDR through Homologous Recombination (HRR) / Dual Incision in GG-NER / cellular response to hydrogen peroxide / DNA-templated DNA replication / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / cellular response to UV / cellular response to xenobiotic stimulus / E3 ubiquitin ligases ubiquitinate target proteins / response to estradiol / heart development / Processing of DNA double-strand break ends / Regulation of TP53 Activity through Phosphorylation / damaged DNA binding / chromosome, telomeric region / DNA replication / nuclear body / DNA repair / centrosome / chromatin binding / protein-containing complex binding / chromatin / enzyme binding / negative regulation of transcription by RNA polymerase II / ATP hydrolysis activity / DNA binding / extracellular exosome / nucleoplasm / ATP binding / identical protein binding / nucleus / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
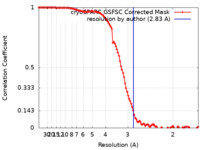
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.83 Å | |||||||||
 データ登録者 データ登録者 | Wang F / He Q / Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Cryo-EM reveals a nearly complete PCNA loading process and unique features of the human alternative clamp loader CTF18-RFC. 著者: Qing He / Feng Wang / Michael E O'Donnell / Huilin Li /  要旨: The DNA sliding clamp PCNA is a multipurpose platform for DNA polymerases and many other proteins involved in DNA metabolism. The topologically closed PCNA ring needs to be cracked open and loaded ...The DNA sliding clamp PCNA is a multipurpose platform for DNA polymerases and many other proteins involved in DNA metabolism. The topologically closed PCNA ring needs to be cracked open and loaded onto DNA by a clamp loader, e.g., the well-studied pentameric ATPase complex RFC (RFC1-5). The CTF18-RFC complex is an alternative clamp loader found recently to bind the leading strand DNA polymerase ε and load PCNA onto leading strand DNA, but its structure and the loading mechanism have been unknown. By cryo-EM analysis of in vitro assembled human CTF18-RFC-DNA-PCNA complex, we have captured seven loading intermediates, revealing a detailed PCNA loading mechanism onto a 3'-ss/dsDNA junction by CTF18-RFC. Interestingly, the alternative loader has evolved a highly mobile CTF18 AAA+ module likely to lower the loading activity, perhaps to avoid competition with the RFC and to limit its role to leading strand clamp loading. To compensate for the lost stability due to the mobile AAA+ module, CTF18 has evolved a unique β-hairpin motif that reaches across RFC2 to interact with RFC5, thereby stabilizing the pentameric complex. Further, we found that CTF18 also contains a separation pin to locally melt DNA from the 3'-end of the primer; this ensures its ability to load PCNA to any 3'-ss/dsDNA junction, facilitated by the binding energy of the E-plug to the major groove. Our study reveals unique structural features of the human CTF18-RFC and contributes to a broader understanding of PCNA loading by the alternative clamp loaders. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42388.map.gz emd_42388.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42388-v30.xml emd-42388-v30.xml emd-42388.xml emd-42388.xml | 31.8 KB 31.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42388_fsc.xml emd_42388_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42388.png emd_42388.png | 61.6 KB | ||
| Filedesc metadata |  emd-42388.cif.gz emd-42388.cif.gz | 8.8 KB | ||
| その他 |  emd_42388_additional_1.map.gz emd_42388_additional_1.map.gz emd_42388_additional_2.map.gz emd_42388_additional_2.map.gz emd_42388_half_map_1.map.gz emd_42388_half_map_1.map.gz emd_42388_half_map_2.map.gz emd_42388_half_map_2.map.gz | 110.8 MB 62.6 MB 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42388 http://ftp.pdbj.org/pub/emdb/structures/EMD-42388 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42388 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42388 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42388_validation.pdf.gz emd_42388_validation.pdf.gz | 954.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42388_full_validation.pdf.gz emd_42388_full_validation.pdf.gz | 954 KB | 表示 | |
| XML形式データ |  emd_42388_validation.xml.gz emd_42388_validation.xml.gz | 18.9 KB | 表示 | |
| CIF形式データ |  emd_42388_validation.cif.gz emd_42388_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42388 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42388 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42388 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42388 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8umyMC  8umtC  8umuC  8umvC  8umwC  8un0C  8unjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42388.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42388.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM sharpen map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: EM sharpen map by DeepEMhancer
| ファイル | emd_42388_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM sharpen map by DeepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Original unsharpen EM map
| ファイル | emd_42388_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Original unsharpen EM map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EM half map A
| ファイル | emd_42388_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EM half map B
| ファイル | emd_42388_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : the human clamp-clamp loader complex PCNA-CTF18
+超分子 #1: the human clamp-clamp loader complex PCNA-CTF18
+分子 #1: Chromosome transmission fidelity protein 18 homolog
+分子 #2: Replication factor C subunit 2
+分子 #3: Replication factor C subunit 5
+分子 #4: Replication factor C subunit 4
+分子 #5: Replication factor C subunit 3
+分子 #6: Proliferating cell nuclear antigen
+分子 #7: DNA (40-MER)
+分子 #8: DNA (20-MER)
+分子 #9: MAGNESIUM ION
+分子 #10: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
+分子 #11: ADENOSINE-5'-DIPHOSPHATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 280 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 123 |
|---|---|
| 得られたモデル |  PDB-8umy: |
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)