+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 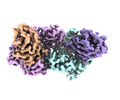 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Deinococcus aerius TR0125 C-glucosyl deglycosidase (CGD), cryo-EM | ||||||||||||||||||
 マップデータ マップデータ | Refinement map. | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | bacterial C-glucosyl deglycosidase / C-C bond cleavage / C-glucosyl aromatic polyketides / C-glucosyl flavonoids / Deinococcus aerius / soil bacterium / N-terminal DUF6379 beta-sandwich domain / C-terminal TIM-barrel domain / alpha2beta2 heterotetramer / LYASE | ||||||||||||||||||
| 機能・相同性 | Domain of unknown function DUF6379 / Domain of unknown function (DUF6379) / Xylose isomerase-like, TIM barrel domain / Xylose isomerase-like TIM barrel / Xylose isomerase-like superfamily / Xylose isomerase-like TIM barrel domain-containing protein / DUF6379 domain-containing protein 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |  Deinococcus aerius (バクテリア) Deinococcus aerius (バクテリア) | ||||||||||||||||||
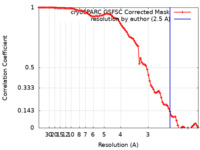
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Furlanetto V / Kalyani DC / Kostelac A / Haltrich D / Hallberg BM / Divne C | ||||||||||||||||||
| 資金援助 |  スウェーデン, スウェーデン,  オーストリア, 5件 オーストリア, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2024 ジャーナル: J Mol Biol / 年: 2024タイトル: Structural and Functional Characterization of a Gene Cluster Responsible for Deglycosylation of C-glucosyl Flavonoids and Xanthonoids by Deinococcus aerius. 著者: Valentina Furlanetto / Dayanand C Kalyani / Anja Kostelac / Jolanta Puc / Dietmar Haltrich / B Martin Hällberg / Christina Divne /   要旨: Plant C-glycosylated aromatic polyketides are important for plant and animal health. These are specialized metabolites that perform functions both within the plant, and in interaction with soil or ...Plant C-glycosylated aromatic polyketides are important for plant and animal health. These are specialized metabolites that perform functions both within the plant, and in interaction with soil or intestinal microbes. Despite the importance of these plant compounds, there is still limited knowledge of how they are metabolized. The Gram-positive aerobic soil bacterium Deinococcus aerius strain TR0125 and other Deinococcus species thrive in a wide range of harsh environments. In this work, we identified a C-glycoside deglycosylation gene cluster in the genome of D. aerius. The cluster includes three genes coding for a GMC-type oxidoreductase (DaCGO1) that oxidizes the glucosyl C3 position in aromatic C-glucosyl compounds, which in turn provides the substrate for the C-glycoside deglycosidase (DaCGD; composed of α+β subunits) that cleaves the glucosyl-aglycone C-C bond. Our results from size-exclusion chromatography, single particle cryo-electron microscopy and X-ray crystallography show that DaCGD is an αβ heterotetramer, which represents a novel oligomeric state among bacterial CGDs. Importantly, the high-resolution X-ray structure of DaCGD provides valuable insights into the activation of the catalytic hydroxide ion by Lys261. DaCGO1 is specific for the 6-C-glucosyl flavones isovitexin, isoorientin and the 2-C-glucosyl xanthonoid mangiferin, and the subsequent C-C-bond cleavage by DaCGD generated apigenin, luteolin and norathyriol, respectively. Of the substrates tested, isovitexin was the preferred substrate (DaCGO1, K 0.047 mM, k 51 min; DaCGO1/DaCGD, K 0.083 mM, k 0.42 min). | ||||||||||||||||||
| 履歴 |
|
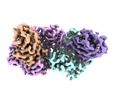
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42375.map.gz emd_42375.map.gz | 55.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42375-v30.xml emd-42375-v30.xml emd-42375.xml emd-42375.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42375_fsc.xml emd_42375_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42375.png emd_42375.png | 96.2 KB | ||
| マスクデータ |  emd_42375_msk_1.map emd_42375_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42375.cif.gz emd-42375.cif.gz | 6.4 KB | ||
| その他 |  emd_42375_half_map_1.map.gz emd_42375_half_map_1.map.gz emd_42375_half_map_2.map.gz emd_42375_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42375 http://ftp.pdbj.org/pub/emdb/structures/EMD-42375 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42375 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42375 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42375_validation.pdf.gz emd_42375_validation.pdf.gz | 760.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42375_full_validation.pdf.gz emd_42375_full_validation.pdf.gz | 759.7 KB | 表示 | |
| XML形式データ |  emd_42375_validation.xml.gz emd_42375_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_42375_validation.cif.gz emd_42375_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42375 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42375 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42375 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42375 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42375.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42375.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refinement map. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.01 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42375_msk_1.map emd_42375_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
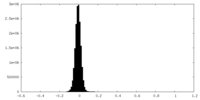
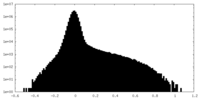
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfmap B
| ファイル | emd_42375_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfmap A
| ファイル | emd_42375_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C-glucosyl deglycosidase, alpha subunit
| 全体 | 名称: C-glucosyl deglycosidase, alpha subunit |
|---|---|
| 要素 |
|
-超分子 #1: C-glucosyl deglycosidase, alpha subunit
| 超分子 | 名称: C-glucosyl deglycosidase, alpha subunit / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: The complex contains two alpha subunits and two beta subunits that form an alpha2beta2 heteroteramer |
|---|---|
| 由来(天然) | 生物種:  Deinococcus aerius (バクテリア) / 株: TR0125 Deinococcus aerius (バクテリア) / 株: TR0125 |
-分子 #1: Xylose isomerase-like TIM barrel domain-containing protein
| 分子 | 名称: Xylose isomerase-like TIM barrel domain-containing protein タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Deinococcus aerius (バクテリア) / 株: TR0125 Deinococcus aerius (バクテリア) / 株: TR0125 |
| 分子量 | 理論値: 40.554328 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTQPQIKRGV SLYSFQEEFF LRKMTLEDCV AACASMGAYG IESLAEQMMP GFPNLDDAFY DGWHAMMAKY GTVSVCHDMF LDTKKFRGR LMTLDEQVES FVRDIRHASR LGCTVIRVLN FVSPELMEKV LPHAEQSNMR LGLEIHAPMH FEHPWVLRHI E FMDRLGSP ...文字列: MTQPQIKRGV SLYSFQEEFF LRKMTLEDCV AACASMGAYG IESLAEQMMP GFPNLDDAFY DGWHAMMAKY GTVSVCHDMF LDTKKFRGR LMTLDEQVES FVRDIRHASR LGCTVIRVLN FVSPELMEKV LPHAEQSNMR LGLEIHAPMH FEHPWVLRHI E FMDRLGSP LLGFIPDMGI FTKHFPPVMA ERLIRQGATP HIIEYIREQY DRRVLAEYVV GDVRNMGGNP VDIRAAEMLR HN NWSNPRR LLEHMDRIFH VHAKFYEMDE QDRETSLGYE EVIPVLKEGG YSGYLASEYE GNRHIQDAFE VDSVEQVRRH QRM LARLIG EREVAHVAEN LYFQSHHHHH H UniProtKB: Xylose isomerase-like TIM barrel domain-containing protein |
-分子 #2: DUF6379 domain-containing protein
| 分子 | 名称: DUF6379 domain-containing protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Deinococcus aerius (バクテリア) / 株: TR0125 Deinococcus aerius (バクテリア) / 株: TR0125 |
| 分子量 | 理論値: 13.755647 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFDKYIVVED SLKRVPGGVQ FGVRLPYYRG LGLSMVETMD VTVDGERVPE ENLTVTLGDR TVPFARRDDE TDTIWNFGEI ATVTARLPH ELGPGEHQVG VNFGLRISYF PVPMVGQDAK TLKL UniProtKB: DUF6379 domain-containing protein |
-分子 #3: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 詳細 | Data collected at the Karolinska Institutet 3D-EM facility |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 2153 / 平均露光時間: 1.5 sec. / 平均電子線量: 56.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 20.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X