+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Prefusion structure of the PRD-0038 spike glycoprotein ectodomain trimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Sarbecoviruses / Spike glycoprotein / fusion protein / neutralizing antibodies / Structural Genomics / Seattle Structural Genomics Center for Infectious Disease / SSGCID / viral protein | |||||||||
| 生物種 |  Sarbecovirus sp. (ウイルス) Sarbecovirus sp. (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Lee J / Park YJ / Veesler D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2023 ジャーナル: Cell Host Microbe / 年: 2023タイトル: Broad receptor tropism and immunogenicity of a clade 3 sarbecovirus. 著者: Jimin Lee / Samantha K Zepeda / Young-Jun Park / Ashley L Taylor / Joel Quispe / Cameron Stewart / Elizabeth M Leaf / Catherine Treichel / Davide Corti / Neil P King / Tyler N Starr / David Veesler /   要旨: Although Rhinolophus bats harbor diverse clade 3 sarbecoviruses, the structural determinants of receptor tropism along with the antigenicity of their spike (S) glycoproteins remain uncharacterized. ...Although Rhinolophus bats harbor diverse clade 3 sarbecoviruses, the structural determinants of receptor tropism along with the antigenicity of their spike (S) glycoproteins remain uncharacterized. Here, we show that the African Rhinolophus bat clade 3 sarbecovirus PRD-0038 S has a broad angiotensin-converting enzyme 2 (ACE2) usage and that receptor-binding domain (RBD) mutations further expand receptor promiscuity and enable human ACE2 utilization. We determine a cryo-EM structure of the PRD-0038 RBD bound to Rhinolophus alcyone ACE2, explaining receptor tropism and highlighting differences with SARS-CoV-1 and SARS-CoV-2. Characterization of PRD-0038 S using cryo-EM and monoclonal antibody reactivity reveals its distinct antigenicity relative to SARS-CoV-2 and identifies PRD-0038 cross-neutralizing antibodies for pandemic preparedness. PRD-0038 S vaccination elicits greater titers of antibodies cross-reacting with vaccine-mismatched clade 2 and clade 1a sarbecoviruses compared with SARS-CoV-2 S due to broader antigenic targeting, motivating the inclusion of clade 3 antigens in next-generation vaccines for enhanced resilience to viral evolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41842.map.gz emd_41842.map.gz | 484.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41842-v30.xml emd-41842-v30.xml emd-41842.xml emd-41842.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41842.png emd_41842.png | 107.7 KB | ||
| Filedesc metadata |  emd-41842.cif.gz emd-41842.cif.gz | 6.5 KB | ||
| その他 |  emd_41842_additional_1.map.gz emd_41842_additional_1.map.gz emd_41842_half_map_1.map.gz emd_41842_half_map_1.map.gz emd_41842_half_map_2.map.gz emd_41842_half_map_2.map.gz | 257.7 MB 474.6 MB 474.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41842 http://ftp.pdbj.org/pub/emdb/structures/EMD-41842 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41842 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41842 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41842_validation.pdf.gz emd_41842_validation.pdf.gz | 896.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41842_full_validation.pdf.gz emd_41842_full_validation.pdf.gz | 896.3 KB | 表示 | |
| XML形式データ |  emd_41842_validation.xml.gz emd_41842_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_41842_validation.cif.gz emd_41842_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41842 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41842 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41842 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41842 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41842.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41842.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_41842_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
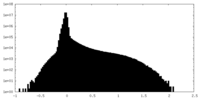
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41842_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
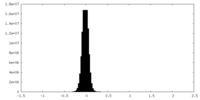
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41842_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PRD-0038 Spike glycoprotein
| 全体 | 名称: PRD-0038 Spike glycoprotein |
|---|---|
| 要素 |
|
-超分子 #1: PRD-0038 Spike glycoprotein
| 超分子 | 名称: PRD-0038 Spike glycoprotein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Sarbecovirus sp. (ウイルス) Sarbecovirus sp. (ウイルス) |
-分子 #1: PRD-0038 Spike glycoprotein
| 分子 | 名称: PRD-0038 Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sarbecovirus sp. (ウイルス) Sarbecovirus sp. (ウイルス) |
| 分子量 | 理論値: 141.350484 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GTQEGCGILS NKSKPALTQY SSSRRGFYYF DDTFRSSVRV LTTGYFLPFN SNLTGYSSR NAVTGRLIQF DNPNIPFKDG LYFAATERSN VIRGWIFGST LDNTTQSAVL FNNGTHIVIN VCNFYFCQDP M LTVANGSH ...文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GTQEGCGILS NKSKPALTQY SSSRRGFYYF DDTFRSSVRV LTTGYFLPFN SNLTGYSSR NAVTGRLIQF DNPNIPFKDG LYFAATERSN VIRGWIFGST LDNTTQSAVL FNNGTHIVIN VCNFYFCQDP M LTVANGSH YKSWVFLNAT NCTYNRVHAF EIDPSLNTGA FIHLREHVFR NVDGFLYVYH NYERANVYDN FPSGFSVLKP IL KLPFGLN ITQFKVIMTL FSPTTSSFNA DASVYFVGHL KPLTMLAEFD ENGTITDAVD CSQDPLSELK CTTKSLTVEK GIY QTSNFR VSPSTEVVRF PNITNLCPFG QVFNASKFPS VYAWERLRIS DCVADYSVLY NSSSSFSTFK CYGVSPTKLN DLCF SSVYA DYFVVKGDDV RQIAPAQTGV IADYNYKLPD DFTGCVLAWN TNSVDSKQGN NFYYRLFRHG KIKPYERDIS NVLYN SAGG TCSSTSQLGC YEPLKSYGFT PTVGVGYQPY RVVVLSFELL NAPATVCGPK KSTELVKNKC VNFNFNGLTG TGVLTS STK KFQPFQQFGR DVSDFTDSVR DPKTLEILDI SPCSYGGVSV ITPGTNTSKA VAVLYQDVNC TDVPTMLHVE QVSTDWR VY ALSADGNMFQ TQAGCLVGAT YENSTYECDI PIGAGICAKF GSNKLRLESI VAYTMSIGED QSIAYSNNTI AIPTNFSI S VTTEVLPVSM TKTSVDCNMY ICGDSTECSN LLLQYGSFCT QLNRALSGIA VEQDRNTRDV FAQTKAIYKT PNIKDFGGF NFSQILPDPK KPSYRSPIED LLYNKVTLSD PGFMKQYGDC LGGINARDLI CAQKFNGLTV LPPLLTDDMI AAYTAALISG TATAGYTFG AGAALQIPFP MQMAYRFNGI GVTQNVLYEN QKQIANQFNN AISKIQDSLT TTPAALGKLQ DVINQNAVAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQAAP HGVVFLHVTY VPSQQQNFTT APAICHNGKA YFPREGVFVM NGTHWFITQR NFYSPQVITT DNT FESGSC DVVIGIVNNT VYDPLQPELE SFKQELDKYF KNHTSPDVDL GDISGINASV VDIKKEIAHL NEIAKNLNES LIDL QELGK YEQYVKSGRE NLYFQGGGGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGHH HHHHHHGGSS GLNDIFEAQK IEWHE |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 33 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 103347 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)