+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Polyclonal immune complex of Fab binding the H1 HA from serum of subject 2-2 at week 16 | |||||||||
 マップデータ マップデータ | Negative stain map of global refinement of particle stack used to sort polyclonal responses to H1 HA from human subject 2-2 at week 16 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | influenza / hemagglutinin / H2 / polyclonal complex / Fab complex / viral fusion protein / VIRAL PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Yang YR / Han J / Richey ST / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2023 タイトル: Immune memory shapes human polyclonal antibody responses to H2N2 vaccination. 著者: Yuhe R Yang / Julianna Han / Hailee R Perrett / Sara T Richey / Abigail M Jackson / Alesandra J Rodriguez / Rebecca A Gillespie / Sarah O'Connell / Julie E Raab / Lauren Y Cominsky / Ankita ...著者: Yuhe R Yang / Julianna Han / Hailee R Perrett / Sara T Richey / Abigail M Jackson / Alesandra J Rodriguez / Rebecca A Gillespie / Sarah O'Connell / Julie E Raab / Lauren Y Cominsky / Ankita Chopde / Masaru Kanekiyo / Katherine V Houser / Grace L Chen / Adrian B McDermott / Sarah F Andrews / Andrew B Ward /   要旨: Influenza A virus subtype H2N2, which caused the 1957 influenza pandemic, remains a global threat. A recent phase I clinical trial investigating a ferritin nanoparticle displaying H2 hemagglutinin in ...Influenza A virus subtype H2N2, which caused the 1957 influenza pandemic, remains a global threat. A recent phase I clinical trial investigating a ferritin nanoparticle displaying H2 hemagglutinin in H2-naïve and H2-exposed adults. Therefore, we could perform comprehensive structural and biochemical characterization of immune memory on the breadth and diversity of the polyclonal serum antibody response elicited after H2 vaccination. We temporally map the epitopes targeted by serum antibodies after first and second vaccinations and show previous H2 exposure results in higher responses to the variable head domain of hemagglutinin while initial responses in H2-naïve participants are dominated by antibodies targeting conserved epitopes. We use cryo-EM and monoclonal B cell isolation to describe the molecular details of cross-reactive antibodies targeting conserved epitopes on the hemagglutinin head including the receptor binding site and a new site of vulnerability deemed the medial junction. Our findings accentuate the impact of pre-existing influenza exposure on serum antibody responses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
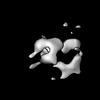
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41563.map.gz emd_41563.map.gz | 17 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41563-v30.xml emd-41563-v30.xml emd-41563.xml emd-41563.xml | 29 KB 29 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41563.png emd_41563.png | 44 KB | ||
| Filedesc metadata |  emd-41563.cif.gz emd-41563.cif.gz | 4.7 KB | ||
| その他 |  emd_41563_additional_1.map.gz emd_41563_additional_1.map.gz emd_41563_additional_2.map.gz emd_41563_additional_2.map.gz emd_41563_additional_3.map.gz emd_41563_additional_3.map.gz emd_41563_additional_4.map.gz emd_41563_additional_4.map.gz emd_41563_additional_5.map.gz emd_41563_additional_5.map.gz emd_41563_additional_6.map.gz emd_41563_additional_6.map.gz emd_41563_half_map_1.map.gz emd_41563_half_map_1.map.gz emd_41563_half_map_2.map.gz emd_41563_half_map_2.map.gz | 12.6 MB 17 MB 17 MB 12.6 MB 12.6 MB 12.6 MB 17 MB 17 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41563 http://ftp.pdbj.org/pub/emdb/structures/EMD-41563 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41563 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41563 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41563_validation.pdf.gz emd_41563_validation.pdf.gz | 575.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41563_full_validation.pdf.gz emd_41563_full_validation.pdf.gz | 574.8 KB | 表示 | |
| XML形式データ |  emd_41563_validation.xml.gz emd_41563_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_41563_validation.cif.gz emd_41563_validation.cif.gz | 12 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41563 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41563 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41563 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41563 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41563.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41563.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of global refinement of particle stack used to sort polyclonal responses to H1 HA from human subject 2-2 at week 16 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.77 Å | ||||||||||||||||||||||||||||||||||||
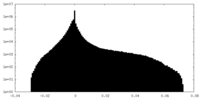
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Negative stain map of polyclonal Fab binding the...
| ファイル | emd_41563_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of polyclonal Fab binding the H1 HA top head 1 epitope from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Negative stain map of polyclonal Fab binding the...
| ファイル | emd_41563_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of polyclonal Fab binding the H1 HA central stem epitope from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Negative stain map of polyclonal Fab binding the...
| ファイル | emd_41563_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of polyclonal Fab binding the H1 HA RBS epitope from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Negative stain map of polyclonal Fab binding the...
| ファイル | emd_41563_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of polyclonal Fab binding the H1 HA medial junction epitope from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Negative stain map of polyclonal Fab binding the...
| ファイル | emd_41563_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of polyclonal Fab binding the H1 HA top head 2 epitope from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Negative stain map of polyclonal Fab binding the...
| ファイル | emd_41563_additional_6.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of polyclonal Fab binding the H1 HA top head 3 epitope from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Negative stain map of half map A for...
| ファイル | emd_41563_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of half map A for global refinement of particle stack used to sort polyclonal responses to H1 HA from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Negative stain map of half map B for...
| ファイル | emd_41563_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain map of half map B for global refinement of particle stack used to sort polyclonal responses to H1 HA from human subject 2-2 at week 16 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Polyclonal immune complex of Fab binding the H1 HA from serum of ...
| 全体 | 名称: Polyclonal immune complex of Fab binding the H1 HA from serum of subject 2-2 at week 16 |
|---|---|
| 要素 |
|
-超分子 #1: Polyclonal immune complex of Fab binding the H1 HA from serum of ...
| 超分子 | 名称: Polyclonal immune complex of Fab binding the H1 HA from serum of subject 2-2 at week 16 タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 280 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.015 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 染色 | タイプ: NEGATIVE / 材質: 2% w/v uranyl formate |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD / 最大 デフォーカス(公称値): -2.0 µm / 最小 デフォーカス(公称値): -2.0 µm |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















































































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)