+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Human ACKR3 phosphorylated by GRK5 in complex with Arrestin3 | |||||||||||||||||||||||||||||||||||||||
 Map data Map data | ||||||||||||||||||||||||||||||||||||||||
 Sample Sample |
| |||||||||||||||||||||||||||||||||||||||
 Keywords Keywords | complex / GPCR / arrestin / signaling / SIGNALING PROTEIN | |||||||||||||||||||||||||||||||||||||||
| Biological species |  | |||||||||||||||||||||||||||||||||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.9 Å | |||||||||||||||||||||||||||||||||||||||
 Authors Authors | Chen Q / Tesmer JJG | |||||||||||||||||||||||||||||||||||||||
| Funding support |  United States, United States,  Denmark, 12 items Denmark, 12 items
| |||||||||||||||||||||||||||||||||||||||
 Citation Citation |  Journal: Nature / Year: 2025 Journal: Nature / Year: 2025Title: Effect of phosphorylation barcodes on arrestin binding to a chemokine receptor. Authors: Qiuyan Chen / Christopher T Schafer / Somnath Mukherjee / Kai Wang / Martin Gustavsson / James R Fuller / Katelyn Tepper / Thomas D Lamme / Yasmin Aydin / Parth Agrawal / Genki Terashi / Xin- ...Authors: Qiuyan Chen / Christopher T Schafer / Somnath Mukherjee / Kai Wang / Martin Gustavsson / James R Fuller / Katelyn Tepper / Thomas D Lamme / Yasmin Aydin / Parth Agrawal / Genki Terashi / Xin-Qiu Yao / Daisuke Kihara / Anthony A Kossiakoff / Tracy M Handel / John J G Tesmer /    Abstract: Unique phosphorylation 'barcodes' installed in different regions of an active seven-transmembrane receptor by different G-protein-coupled receptor (GPCR) kinases (GRKs) have been proposed to promote ...Unique phosphorylation 'barcodes' installed in different regions of an active seven-transmembrane receptor by different G-protein-coupled receptor (GPCR) kinases (GRKs) have been proposed to promote distinct cellular outcomes, but it is unclear whether or how arrestins differentially engage these barcodes. Here, to address this, we developed an antigen-binding fragment (Fab7) that recognizes both active arrestin2 (β-arrestin1) and arrestin3 (β-arrestin2) without interacting with bound receptor polypeptides. We used Fab7 to determine the structures of both arrestins in complex with atypical chemokine receptor 3 (ACKR3) phosphorylated in different regions of its C-terminal tail by either GRK2 or GRK5 (ref. ). The GRK2-phosphorylated ACKR3 resulted in more heterogeneous 'tail-mode' assemblies, whereas phosphorylation by GRK5 resulted in more rigid 'ACKR3-adjacent' assemblies. Unexpectedly, the finger loops of both arrestins engaged the micelle surface rather than the receptor intracellular pocket, with arrestin3 being more dynamic, partly because of its lack of a membrane-anchoring motif. Thus, both the region of the barcode and the arrestin isoform involved can alter the structure and dynamics of GPCR-arrestin complexes, providing a possible mechanistic basis for unique downstream cellular effects, such as the efficiency of chemokine scavenging and the robustness of arrestin binding in ACKR3. | |||||||||||||||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_41290.map.gz emd_41290.map.gz | 168.1 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-41290-v30.xml emd-41290-v30.xml emd-41290.xml emd-41290.xml | 18 KB 18 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_41290_fsc.xml emd_41290_fsc.xml | 13.4 KB | Display |  FSC data file FSC data file |
| Images |  emd_41290.png emd_41290.png | 43.2 KB | ||
| Filedesc metadata |  emd-41290.cif.gz emd-41290.cif.gz | 4.3 KB | ||
| Others |  emd_41290_half_map_1.map.gz emd_41290_half_map_1.map.gz emd_41290_half_map_2.map.gz emd_41290_half_map_2.map.gz | 165.4 MB 165.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41290 http://ftp.pdbj.org/pub/emdb/structures/EMD-41290 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41290 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41290 | HTTPS FTP |
-Related structure data
| Related structure data |  8tiiC  8tilC  8tinC  8tioC  8vj9C  9e82C  41287  41288  41293  41294 C: citing same article ( |
|---|
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_41290.map.gz / Format: CCP4 / Size: 178 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_41290.map.gz / Format: CCP4 / Size: 178 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.054 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #1
| File | emd_41290_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
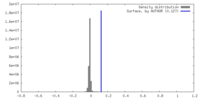
| Density Histograms |
-Half map: #2
| File | emd_41290_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
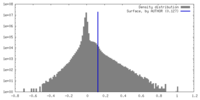
| Density Histograms |
- Sample components
Sample components
-Entire : human ACKR3 phosphorylated by GRK5 in complex with Arrestin3
| Entire | Name: human ACKR3 phosphorylated by GRK5 in complex with Arrestin3 |
|---|---|
| Components |
|
-Supramolecule #1: human ACKR3 phosphorylated by GRK5 in complex with Arrestin3
| Supramolecule | Name: human ACKR3 phosphorylated by GRK5 in complex with Arrestin3 type: complex / ID: 1 / Parent: 0 |
|---|---|
| Source (natural) | Organism:  |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.7 mg/mL |
|---|---|
| Buffer | pH: 8 |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Average electron dose: 56.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.5 µm / Nominal defocus min: 0.6 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)