+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Main central pair structure | |||||||||
 マップデータ マップデータ | Main CP map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cilia / Axoneme / central pair / microtubules / STRUCTURAL PROTEIN | |||||||||
| 生物種 |  | |||||||||
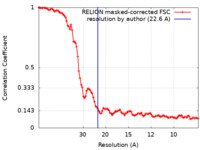
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 22.6 Å | |||||||||
 データ登録者 データ登録者 | Legal T / Bui KH | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2023 ジャーナル: J Cell Biol / 年: 2023タイトル: CEP104/FAP256 and associated cap complex maintain stability of the ciliary tip. 著者: Thibault Legal / Mireya Parra / Maxwell Tong / Corbin S Black / Ewa Joachimiak / Melissa Valente-Paterno / Karl Lechtreck / Jacek Gaertig / Khanh Huy Bui /    要旨: Cilia are essential organelles that protrude from the cell body. Cilia are made of a microtubule-based structure called the axoneme. In most types of cilia, the ciliary tip is distinct from the rest ...Cilia are essential organelles that protrude from the cell body. Cilia are made of a microtubule-based structure called the axoneme. In most types of cilia, the ciliary tip is distinct from the rest of the cilium. Here, we used cryo-electron tomography and subtomogram averaging to obtain the structure of the ciliary tip of the ciliate Tetrahymena thermophila. We show that the microtubules at the tip are highly crosslinked with each other and stabilized by luminal proteins, plugs, and cap proteins at the plus ends. In the tip region, the central pair lacks typical projections and twists significantly. By analyzing cells lacking a ciliary tip-enriched protein CEP104/FAP256 by cryo-electron tomography and proteomics, we discovered candidates for the central pair cap complex and explained the potential functions of CEP104/FAP256. These data provide new insights into the function of the ciliary tip and the mechanisms of ciliary assembly and length regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40891.map.gz emd_40891.map.gz | 83.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40891-v30.xml emd-40891-v30.xml emd-40891.xml emd-40891.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40891_fsc.xml emd_40891_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40891.png emd_40891.png | 110.5 KB | ||
| Filedesc metadata |  emd-40891.cif.gz emd-40891.cif.gz | 3.8 KB | ||
| その他 |  emd_40891_half_map_1.map.gz emd_40891_half_map_1.map.gz emd_40891_half_map_2.map.gz emd_40891_half_map_2.map.gz | 71.7 MB 71.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40891 http://ftp.pdbj.org/pub/emdb/structures/EMD-40891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40891 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40891_validation.pdf.gz emd_40891_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40891_full_validation.pdf.gz emd_40891_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_40891_validation.xml.gz emd_40891_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_40891_validation.cif.gz emd_40891_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40891 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40891 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40891 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40891 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40891.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40891.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main CP map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.24 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half-map2
| ファイル | emd_40891_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map1
| ファイル | emd_40891_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 32-nm repeat of the main ciliary central pair
| 全体 | 名称: 32-nm repeat of the main ciliary central pair |
|---|---|
| 要素 |
|
-超分子 #1: 32-nm repeat of the main ciliary central pair
| 超分子 | 名称: 32-nm repeat of the main ciliary central pair / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 詳細: 32-nm repeat of the main ciliary central pair from Tetrahymena thermophila CU428 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 4.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)