+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of immature PhiKZ Major Capsid Protein from tubular arrays | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | major capsid protein / HK97-fold / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 | viral capsid / Major capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Phikzvirus phiKZ (ウイルス) Phikzvirus phiKZ (ウイルス) | |||||||||||||||
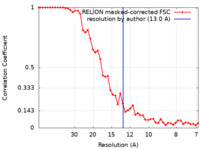
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 13.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Laughlin TG / Villa E | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: An intron endonuclease facilitates interference competition between coinfecting viruses. 著者: Erica A Birkholz / Chase J Morgan / Thomas G Laughlin / Rebecca K Lau / Amy Prichard / Sahana Rangarajan / Gabrielle N Meza / Jina Lee / Emily Armbruster / Sergey Suslov / Kit Pogliano / ...著者: Erica A Birkholz / Chase J Morgan / Thomas G Laughlin / Rebecca K Lau / Amy Prichard / Sahana Rangarajan / Gabrielle N Meza / Jina Lee / Emily Armbruster / Sergey Suslov / Kit Pogliano / Justin R Meyer / Elizabeth Villa / Kevin D Corbett / Joe Pogliano /  要旨: Introns containing homing endonucleases are widespread in nature and have long been assumed to be selfish elements that provide no benefit to the host organism. These genetic elements are common in ...Introns containing homing endonucleases are widespread in nature and have long been assumed to be selfish elements that provide no benefit to the host organism. These genetic elements are common in viruses, but whether they confer a selective advantage is unclear. In this work, we studied intron-encoded homing endonuclease gp210 in bacteriophage ΦPA3 and found that it contributes to viral competition by interfering with the replication of a coinfecting phage, ΦKZ. We show that gp210 targets a specific sequence in ΦKZ, which prevents the assembly of progeny viruses. This work demonstrates how a homing endonuclease can be deployed in interference competition among viruses and provide a relative fitness advantage. Given the ubiquity of homing endonucleases, this selective advantage likely has widespread evolutionary implications in diverse plasmid and viral competition as well as virus-host interactions. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40674.map.gz emd_40674.map.gz | 5.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40674-v30.xml emd-40674-v30.xml emd-40674.xml emd-40674.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40674_fsc.xml emd_40674_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40674.png emd_40674.png | 55.7 KB | ||
| マスクデータ |  emd_40674_msk_1.map emd_40674_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40674.cif.gz emd-40674.cif.gz | 5.6 KB | ||
| その他 |  emd_40674_half_map_1.map.gz emd_40674_half_map_1.map.gz emd_40674_half_map_2.map.gz emd_40674_half_map_2.map.gz | 5.4 MB 5.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40674 http://ftp.pdbj.org/pub/emdb/structures/EMD-40674 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40674 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40674 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40674_validation.pdf.gz emd_40674_validation.pdf.gz | 896.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40674_full_validation.pdf.gz emd_40674_full_validation.pdf.gz | 895.9 KB | 表示 | |
| XML形式データ |  emd_40674_validation.xml.gz emd_40674_validation.xml.gz | 10.5 KB | 表示 | |
| CIF形式データ |  emd_40674_validation.cif.gz emd_40674_validation.cif.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40674 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40674 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40674 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40674 | HTTPS FTP |
-関連構造データ
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40674.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40674.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.457 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40674_msk_1.map emd_40674_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40674_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40674_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Immature PhiKZ major capsid protein in a tubular array
| 全体 | 名称: Immature PhiKZ major capsid protein in a tubular array |
|---|---|
| 要素 |
|
-超分子 #1: Immature PhiKZ major capsid protein in a tubular array
| 超分子 | 名称: Immature PhiKZ major capsid protein in a tubular array タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Phikzvirus phiKZ (ウイルス) Phikzvirus phiKZ (ウイルス) |
-分子 #1: PhiKZ major capsid protein
| 分子 | 名称: PhiKZ major capsid protein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Phikzvirus phiKZ (ウイルス) Phikzvirus phiKZ (ウイルス) |
| 配列 | 文字列: MSVHALRELF KHKGEKNYEV FSMEDFIGRL ESEIGLNDSV VSQGRSLISS ISHENFGTVQ ATDIQDAAAI YNKMQMLVND YGFERVSSSD PQVRAREERV RENQITAATM AAIACADETK YIRALRGITK AKASNEDHVK VVQHQFNGPA GGIQVFENGV GLENYNEKSQ ...文字列: MSVHALRELF KHKGEKNYEV FSMEDFIGRL ESEIGLNDSV VSQGRSLISS ISHENFGTVQ ATDIQDAAAI YNKMQMLVND YGFERVSSSD PQVRAREERV RENQITAATM AAIACADETK YIRALRGITK AKASNEDHVK VVQHQFNGPA GGIQVFENGV GLENYNEKSQ RDFRVVTIGY NLAASRQDEF AERIYPTTVI NPIEGGVVQV LPYIAVMKDV YHEVSGVKMD NEEVNMVEAY RDPSILDDES IALIPALDPA GSNADFFVDP ALVPPYTIKN EQNLTITTAP LKANVRLDLM GNSNANLLIQ RGMLEVSDTI DPAGRLKNLF VLLGGKVVKF KVDRLPRAVF QPDLVGDTRN AVIRFDSDDL VVSGDTTFID GSADGVINDL KTAKLSLRLS VGFGGTISLS KGDSKFGATD TYVDKVLNED GQVMDNADPA VKAILDQLTD LAVIGFELDT RFTNTNRRQR GHLLQTRALQ FRHPIPMHAP VTLPMDTMTD EGPGEVVKAL TVNTNIRNSN NAVKRMLNYL AQLREVVHNG YNRPKFGIIE GALSAVMRPT YRYKELDLEK VIDTIKSKDR WDDVCAAILN CVKAELFPAH RDSNIEAAFR VISGNQDETP MYLFCSDKEI ANYLMTKGDD RTLGAYLKYD IVSTNNQLFD GKLVVIPTRA VQQENDILSW GQFFYVSTVI ADLPITRGGH QVTREIAAIP FNLHVNNIPF ALEFKITGFQ KVMGETQFNG KLADLKP UniProtKB: Major capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: HOMEMADE PLUNGER |
| 詳細 | P. aeruginosa cells expressing PhiPA3 gp210-GFPmut1 infected with PhiKZ |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 1 / 平均電子線量: 2.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 7.0 µm / 最小 デフォーカス(公称値): 4.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)