+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pendrin in complex with iodide | |||||||||
 マップデータ マップデータ | Pendrin in complex with iodide | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | chloride transport / iodide transport / bicarbonate transport / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報iodide transmembrane transporter activity / secondary active sulfate transmembrane transporter activity / regulation of pH / chloride:bicarbonate antiporter activity / brush border membrane / regulation of protein localization / apical plasma membrane / extracellular exosome 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Wang L / Hoang A / Zhou M | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Mechanism of anion exchange and small-molecule inhibition of pendrin. 著者: Lie Wang / Anthony Hoang / Eva Gil-Iturbe / Arthur Laganowsky / Matthias Quick / Ming Zhou /  要旨: Pendrin (SLC26A4) is an anion exchanger that mediates bicarbonate (HCO) exchange for chloride (Cl) and is crucial for maintaining pH and salt homeostasis in the kidney, lung, and cochlea. Pendrin ...Pendrin (SLC26A4) is an anion exchanger that mediates bicarbonate (HCO) exchange for chloride (Cl) and is crucial for maintaining pH and salt homeostasis in the kidney, lung, and cochlea. Pendrin also exports iodide (I) in the thyroid gland. Pendrin mutations in humans lead to Pendred syndrome, causing hearing loss and goiter. Inhibition of pendrin is a validated approach for attenuating airway hyperresponsiveness in asthma and for treating hypertension. However, the mechanism of anion exchange and its inhibition by drugs remains poorly understood. We applied cryo-electron microscopy to determine structures of pendrin from Sus scrofa in the presence of either Cl, I, HCO or in the apo-state. The structures reveal two anion-binding sites in each protomer, and functional analyses show both sites are involved in anion exchange. The structures also show interactions between the Sulfate Transporter and Anti-Sigma factor antagonist (STAS) and transmembrane domains, and mutational studies suggest a regulatory role. We also determine the structure of pendrin in a complex with niflumic acid (NFA), which uncovers a mechanism of inhibition by competing with anion binding and impeding the structural changes necessary for anion exchange. These results reveal directions for understanding the mechanisms of anion selectivity and exchange and their regulations by the STAS domain. This work also establishes a foundation for analyzing the pathophysiology of mutations associated with Pendred syndrome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
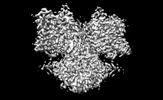
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40479.map.gz emd_40479.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40479-v30.xml emd-40479-v30.xml emd-40479.xml emd-40479.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40479.png emd_40479.png | 72.8 KB | ||
| Filedesc metadata |  emd-40479.cif.gz emd-40479.cif.gz | 6.8 KB | ||
| その他 |  emd_40479_half_map_1.map.gz emd_40479_half_map_1.map.gz emd_40479_half_map_2.map.gz emd_40479_half_map_2.map.gz | 95.4 MB 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40479 http://ftp.pdbj.org/pub/emdb/structures/EMD-40479 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40479 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40479 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40479_validation.pdf.gz emd_40479_validation.pdf.gz | 859.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40479_full_validation.pdf.gz emd_40479_full_validation.pdf.gz | 859.3 KB | 表示 | |
| XML形式データ |  emd_40479_validation.xml.gz emd_40479_validation.xml.gz | 13.3 KB | 表示 | |
| CIF形式データ |  emd_40479_validation.cif.gz emd_40479_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40479 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40479 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40479 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40479 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40479.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40479.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pendrin in complex with iodide | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.826 Å | ||||||||||||||||||||||||||||||||||||
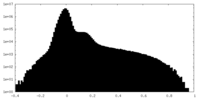
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_40479_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
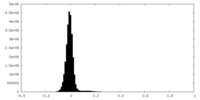
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_40479_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
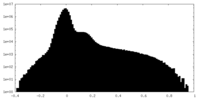
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : homodimer of Pendrin
| 全体 | 名称: homodimer of Pendrin |
|---|---|
| 要素 |
|
-超分子 #1: homodimer of Pendrin
| 超分子 | 名称: homodimer of Pendrin / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 75 KDa |
-分子 #1: Pendrin
| 分子 | 名称: Pendrin / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 86.113273 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAPGSRLEP PPLPEYSCSY VVSRPVYSEL AFQQQYERRL QERKTLRESL AKRCSCSRKR TLGVLKTLLP VLDWLPKYRI KEWLLSDII SGVSTGLVGT LQGMAYALLA AVPVGYGLYS AFFPILTYFI FGTSRHISVG PFPVVSLMVG SVVLSMAPDE H FIISSSNG ...文字列: MAAPGSRLEP PPLPEYSCSY VVSRPVYSEL AFQQQYERRL QERKTLRESL AKRCSCSRKR TLGVLKTLLP VLDWLPKYRI KEWLLSDII SGVSTGLVGT LQGMAYALLA AVPVGYGLYS AFFPILTYFI FGTSRHISVG PFPVVSLMVG SVVLSMAPDE H FIISSSNG TALNTTVIDF AARDAARVLI ASTLTLLVGI IQLIFGGLQI GFIVRYLADP LVGGFTTAAA FQVLVSQLKI VL NVSTKNY NGILSIIYTL IEIFQNIGNT NLADFIAGLL TIIICMAVKE LNDRFKHKIP VPIPIEVIVT IIATAISYAV NLE KNYNAG IVKSIPRGFL PPEIPPISLF SEMLTASFSI AVVAYAIAVS VGKVYAIKYD YTIDGNQEFI AFGISNIFSG FFSC FVATT ALSRTAVQES TGGKTQIAGI ISAAVVMIAI VALGKLLEPL QKSVLAAVVI ANLKGMFMQV CDVPRLWRQN KTDAV IWVF TCIASIILGL DLGLLAGLMF GFLTVVVRVQ FPSWNSLGSI PNTDIYRSTK DYKNIEEPEG VKILRFSSPI FYGNVD GLK KCIKSTVGFD AIRVYNKRLK ALRKIQKLIK KGQLRATKNG IISDAGSSNN AFEPDEDIED PEELDIPTKE IEIQVDW NS ELPVKVNVPK VPIHSLVLDC GAVSFLDVVG VRSLRMIVKE FQRIDVHVYF ASLQDHVIEK LEQCGFFNDS IRKDIFFL T VHDAILHLRS QVKSQEVQDS ILETITLIQD CKDPLELMEA ELIEEELDVQ DEAMRRLAS UniProtKB: STAS domain-containing protein |
-分子 #2: Lauryl Maltose Neopentyl Glycol
| 分子 | 名称: Lauryl Maltose Neopentyl Glycol / タイプ: ligand / ID: 2 / コピー数: 4 / 式: AV0 |
|---|---|
| 分子量 | 理論値: 1.005188 KDa |
| Chemical component information |  ChemComp-AV0: |
-分子 #3: IODIDE ION
| 分子 | 名称: IODIDE ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: IOD |
|---|---|
| 分子量 | 理論値: 126.904 Da |
| Chemical component information |  ChemComp-IOD: |
-分子 #4: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 4 / コピー数: 12 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #5: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 5 / コピー数: 18 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)