+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM map of a de novo designed T=4 octahedral nanocage hierarchically built from pseudosymmetric trimers; design Oct(T=4)-3 | |||||||||
 マップデータ マップデータ | sharpened map using DeepEMhancer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | icosahedral nanocage / T=4 / T=4 icosahedra / nanomaterial / computational design / de novo / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.87 Å | |||||||||
 データ登録者 データ登録者 | Philomin A / Borst AJ / Kibler RD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Design of four component T=4 tetrahedral, octahedral, and icosahedral protein nanocages through programmed symmetry breaking 著者: Lee S / Kibler RD / Hsia Y / Borst AJ / Philomin A / Kennedy MA / Stoddard B / Baker D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40268.map.gz emd_40268.map.gz | 92 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40268-v30.xml emd-40268-v30.xml emd-40268.xml emd-40268.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40268_fsc.xml emd_40268_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40268.png emd_40268.png | 134.6 KB | ||
| Filedesc metadata |  emd-40268.cif.gz emd-40268.cif.gz | 4.8 KB | ||
| その他 |  emd_40268_additional_1.map.gz emd_40268_additional_1.map.gz emd_40268_half_map_1.map.gz emd_40268_half_map_1.map.gz emd_40268_half_map_2.map.gz emd_40268_half_map_2.map.gz | 50.9 MB 95.4 MB 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40268 http://ftp.pdbj.org/pub/emdb/structures/EMD-40268 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40268 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40268 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40268_validation.pdf.gz emd_40268_validation.pdf.gz | 775.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40268_full_validation.pdf.gz emd_40268_full_validation.pdf.gz | 774.8 KB | 表示 | |
| XML形式データ |  emd_40268_validation.xml.gz emd_40268_validation.xml.gz | 18.1 KB | 表示 | |
| CIF形式データ |  emd_40268_validation.cif.gz emd_40268_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40268 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40268 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40268 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40268 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40268.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40268.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map using DeepEMhancer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.36 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: unsharpened map from final Non-Uniform Refinement
| ファイル | emd_40268_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map from final Non-Uniform Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
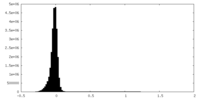
| 密度ヒストグラム |
-ハーフマップ: half map A from final Non-Uniform Refinement
| ファイル | emd_40268_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A from final Non-Uniform Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
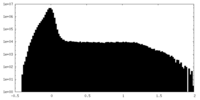
| 密度ヒストグラム |
-ハーフマップ: half map B from final Non-Uniform Refinement
| ファイル | emd_40268_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B from final Non-Uniform Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
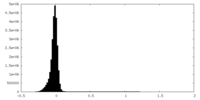
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Oct(T=4)-3
| 全体 | 名称: Oct(T=4)-3 |
|---|---|
| 要素 |
|
-超分子 #1: Oct(T=4)-3
| 超分子 | 名称: Oct(T=4)-3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: A hierarchically designed T=4 octahedral nanocage |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Oct(T=4)-3 chain A
| 分子 | 名称: Oct(T=4)-3 chain A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSLELALKA LQILVNAAYV LAEIARDRGN EELLEKAARL AEEAARQAER IARQARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEL LEYAARLAEE AARQAIEIWA QAMEEGNQQL RTKAAHIILR AAEVLLEIAR DRGNQELLEK AASLVDAVAA LQQAAAAILE ...文字列: MGSLELALKA LQILVNAAYV LAEIARDRGN EELLEKAARL AEEAARQAER IARQARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEL LEYAARLAEE AARQAIEIWA QAMEEGNQQL RTKAAHIILR AAEVLLEIAR DRGNQELLEK AASLVDAVAA LQQAAAAILE GDVEKAVRAA QEAVKAAKEA GDNDMLRAVA IAALRIAKEA EKQGNVEVAV KAARVAVEAA KQAGDQDVLL KVATQAARIA LEAIKQGNLE VKLEALKVAQ EALKQTGGSG GSHHHHHH |
-分子 #2: Oct(T=4)-3 chain B
| 分子 | 名称: Oct(T=4)-3 chain B / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: MGSPRLVLRA LENMVRAAHT LAEIARDNGN EEWLERAARL AEEVARRAER LAREARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEE LEYAARLAEE AARQAIEIAA QAMEEGNLEL ALKALQIIVN AAYVLAEIAR DRGNEELLEK AASLAEAAAA LAEAIAAILE ...文字列: MGSPRLVLRA LENMVRAAHT LAEIARDNGN EEWLERAARL AEEVARRAER LAREARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEE LEYAARLAEE AARQAIEIAA QAMEEGNLEL ALKALQIIVN AAYVLAEIAR DRGNEELLEK AASLAEAAAA LAEAIAAILE GDVEKAVRAA QEAVKAAKEA GDNDMLRAVA IAALRIAKEA EKQGNVEVAV KAARVAVEAA KQAGDQDVLK KVAAQAARIM LEAIKQGNTE VALEALKVAQ EALKQTGGSG GSHHHHHH |
-分子 #3: Oct(T=4)-3 chain C
| 分子 | 名称: Oct(T=4)-3 chain C / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSPELFLQD LRSLVEAARI LARLARQRGD EHALERAARW AEQAARQAER LARQARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEL LEYAARLAEE AARQAIEIAA QAMEEGNFEL ALEALEIINE AARVLARIAH HRGNQELLEK AASLTHASAA LSRAIAAILE ...文字列: MGSPELFLQD LRSLVEAARI LARLARQRGD EHALERAARW AEQAARQAER LARQARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEL LEYAARLAEE AARQAIEIAA QAMEEGNFEL ALEALEIINE AARVLARIAH HRGNQELLEK AASLTHASAA LSRAIAAILE GDVEKAVRAA QEAVKAAKEA GDNDMLRAVA IAALRIAKEA EKQGNVEVAV KAARVAVEAA KQAGDNDVLR KVAEQALRIA KEAEKQGNVI VAMKAIDVAV EAAGQAGDID VIKKVADQTQ RIVDEWLKQG |
-分子 #4: Oct(T=4)-3 chain ho
| 分子 | 名称: Oct(T=4)-3 chain ho / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSPELFLQD LRSLVEAARI LARLARQRGD EHALERAARW AEQAARQAER LARQARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEL LEYAARLAEE AARQAIEIWA QAMEEGNQQL RTKAAHIILR AAEVLLEIAR DRGNQELLEK AASLVDAVAA LQQAAAAILE ...文字列: MGSPELFLQD LRSLVEAARI LARLARQRGD EHALERAARW AEQAARQAER LARQARKEGN LELALKALQI LVNAAYVLAE IARDRGNEEL LEYAARLAEE AARQAIEIWA QAMEEGNQQL RTKAAHIILR AAEVLLEIAR DRGNQELLEK AASLVDAVAA LQQAAAAILE GDVEKAVRAA QEAVKAAKEA GDNDMLRAVA IAALRIAKEA EKQGNVEVAV KAARVAVEAA KQAGDNDVLR KVAEQALRIA KEAEKQGNVY VAAKAVQVAA EAAKQAGDID VLKKVIGQTQ RIRDEWVKQG GSGGSHHHHH H |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 25mM Tris, 300mM NaCl |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)