+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Homo-octamer of PbuCsx28 protein | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-associated protein / ANTIVIRAL PROTEIN | |||||||||
| 機能・相同性 | membrane / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Prevotella buccae (バクテリア) Prevotella buccae (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.65 Å | |||||||||
 データ登録者 データ登録者 | Park JU / Kellogg EH | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Csx28 is a membrane pore that enhances CRISPR-Cas13b-dependent antiphage defense. 著者: Arica R VanderWal / Jung-Un Park / Bogdan Polevoda / Julia K Nicosia / Adrian M Molina Vargas / Elizabeth H Kellogg / Mitchell R O'Connell /  要旨: Type VI CRISPR-Cas systems use RNA-guided ribonuclease (RNase) Cas13 to defend bacteria against viruses, and some of these systems encode putative membrane proteins that have unclear roles in Cas13- ...Type VI CRISPR-Cas systems use RNA-guided ribonuclease (RNase) Cas13 to defend bacteria against viruses, and some of these systems encode putative membrane proteins that have unclear roles in Cas13-mediated defense. We show that Csx28, of type VI-B2 systems, is a transmembrane protein that assists to slow cellular metabolism upon viral infection, increasing antiviral defense. High-resolution cryo-electron microscopy reveals that Csx28 forms an octameric pore-like structure. These Csx28 pores localize to the inner membrane in vivo. Csx28's antiviral activity in vivo requires sequence-specific cleavage of viral messenger RNAs by Cas13b, which subsequently results in membrane depolarization, slowed metabolism, and inhibition of sustained viral infection. Our work suggests a mechanism by which Csx28 acts as a downstream, Cas13b-dependent effector protein that uses membrane perturbation as an antiviral defense strategy. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40059.map.gz emd_40059.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40059-v30.xml emd-40059-v30.xml emd-40059.xml emd-40059.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40059_fsc.xml emd_40059_fsc.xml | 7.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40059.png emd_40059.png | 176.5 KB | ||
| Filedesc metadata |  emd-40059.cif.gz emd-40059.cif.gz | 5.9 KB | ||
| その他 |  emd_40059_half_map_1.map.gz emd_40059_half_map_1.map.gz emd_40059_half_map_2.map.gz emd_40059_half_map_2.map.gz | 27.9 MB 27.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40059 http://ftp.pdbj.org/pub/emdb/structures/EMD-40059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40059 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40059 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40059_validation.pdf.gz emd_40059_validation.pdf.gz | 615 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40059_full_validation.pdf.gz emd_40059_full_validation.pdf.gz | 614.6 KB | 表示 | |
| XML形式データ |  emd_40059_validation.xml.gz emd_40059_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_40059_validation.cif.gz emd_40059_validation.cif.gz | 19.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40059 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40059 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40059 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40059 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gi1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40059.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40059.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.33 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: halfmap 1
| ファイル | emd_40059_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
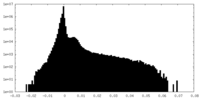
| 密度ヒストグラム |
-ハーフマップ: halfmap 2
| ファイル | emd_40059_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
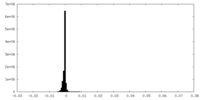
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : octameric assembly of csx28
| 全体 | 名称: octameric assembly of csx28 |
|---|---|
| 要素 |
|
-超分子 #1: octameric assembly of csx28
| 超分子 | 名称: octameric assembly of csx28 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Prevotella buccae (バクテリア) Prevotella buccae (バクテリア) |
| 分子量 | 理論値: 300 KDa |
-分子 #1: Accessory protein Csx28
| 分子 | 名称: Accessory protein Csx28 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Prevotella buccae (バクテリア) Prevotella buccae (バクテリア) |
| 分子量 | 理論値: 21.910193 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDYMELAKEA FSIICTFIAA YVAYYYAIKQ LHQKSVENIE YAKYQAVLQA HKSLYKLLRF TTNTENEDSI LIWEKTKDGK QEATYYFRK ENIRKFIKEL SKEIYNEGCG IFMSKEALSL ISEYRNIVYG FMLSAQNNPQ ETIRITNRES VERMKKIHQN L SIEIRQAI NLKKRDLRFE NLYFQ UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.35 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GRAPHENE OXIDE / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 47.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8gi1: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X