+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of LYCHOS | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Lysosome / GPCR-like / Transporter / Cholesterol / Membrane protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to cholesterol / cholesterol binding / negative regulation of BMP signaling pathway / positive regulation of TORC1 signaling / cellular response to amino acid starvation / transmembrane transport / cognition / intracellular signal transduction / lysosomal membrane / extracellular exosome 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.11 Å | |||||||||
 データ登録者 データ登録者 | Xiong Q / Zhu Z / Li T / Zhou Z / Chao Y / Qu Q / Li D | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2025 ジャーナル: Nat Struct Mol Biol / 年: 2025タイトル: Molecular architecture of human LYCHOS involved in lysosomal cholesterol signaling. 著者: Qi Xiong / Zhini Zhu / Tingting Li / Xiaotian Li / Zixuan Zhou / Yulin Chao / Chuanhui Yang / Suihan Feng / Qianhui Qu / Dianfan Li /  要旨: Lysosomal membrane protein LYCHOS (lysosomal cholesterol signaling) translates cholesterol abundance to mammalian target of rapamycin activation. Here we report the 2.11-Å structure of human LYCHOS, ...Lysosomal membrane protein LYCHOS (lysosomal cholesterol signaling) translates cholesterol abundance to mammalian target of rapamycin activation. Here we report the 2.11-Å structure of human LYCHOS, revealing a unique fusion architecture comprising a G-protein-coupled receptor (GPCR)-like domain and a transporter domain that mediates homodimer assembly. The NhaA-fold transporter harbors a previously uncharacterized intramembrane Na pocket. The GPCR-like domain is stabilized, by analogy to canonical GPCRs, in an inactive state through 'tethered antagonism' by a lumenal loop and strong interactions at the cytosol side preventing the hallmark swing of the sixth transmembrane helix seen in active GPCRs. A cholesterol molecule and an associated docosahexaenoic acid (DHA)-phospholipid are entrapped between the transporter and GPCR-like domains, with the DHA-phospholipid occupying a pocket previously implicated in cholesterol sensing, indicating inter-domain coupling via dynamic lipid-protein interactions. Our work provides a high-resolution framework for functional investigations of the understudied LYCHOS protein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39851.map.gz emd_39851.map.gz | 168.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39851-v30.xml emd-39851-v30.xml emd-39851.xml emd-39851.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39851_fsc.xml emd_39851_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39851.png emd_39851.png | 109.8 KB | ||
| Filedesc metadata |  emd-39851.cif.gz emd-39851.cif.gz | 7.9 KB | ||
| その他 |  emd_39851_additional_1.map.gz emd_39851_additional_1.map.gz emd_39851_half_map_1.map.gz emd_39851_half_map_1.map.gz emd_39851_half_map_2.map.gz emd_39851_half_map_2.map.gz | 89.7 MB 165.1 MB 165.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39851 http://ftp.pdbj.org/pub/emdb/structures/EMD-39851 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39851 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39851 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39851_validation.pdf.gz emd_39851_validation.pdf.gz | 968.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39851_full_validation.pdf.gz emd_39851_full_validation.pdf.gz | 968.2 KB | 表示 | |
| XML形式データ |  emd_39851_validation.xml.gz emd_39851_validation.xml.gz | 20.2 KB | 表示 | |
| CIF形式データ |  emd_39851_validation.cif.gz emd_39851_validation.cif.gz | 26.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39851 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39851 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39851 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39851 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8z8zMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39851.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39851.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.932 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_39851_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_39851_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_39851_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LYCHOS
| 全体 | 名称: LYCHOS |
|---|---|
| 要素 |
|
-超分子 #1: LYCHOS
| 超分子 | 名称: LYCHOS / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 127 KDa |
-分子 #1: Lysosomal cholesterol signaling protein,Fluorescent Protein
| 分子 | 名称: Lysosomal cholesterol signaling protein,Fluorescent Protein タイプ: protein_or_peptide / ID: 1 詳細: The protein is a fusion protein with expression tag. Residues 1-3 is the initial methionine and GS linker. Residues 4-873 is LYCHOS (uniport ID Q7Z3F1). Residues 874-889 is the linker with a ...詳細: The protein is a fusion protein with expression tag. Residues 1-3 is the initial methionine and GS linker. Residues 4-873 is LYCHOS (uniport ID Q7Z3F1). Residues 874-889 is the linker with a 3 C protease digestion site. Residues 890-1114 is a thermostable green fluorescent protein fusion (PDB entry 4TZA, residue 5-229). Residues 1115-1144 is the linker with an expression tag コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 127.213109 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGSMNSNLPA ENLTIAVNMT KTLPTAVTHG FNSTNDPPSM SITRLFPALL ECFGIVLCGY IAGRANVITS TQAKGLGNFV SRFALPALL FKNMVVLNFS NVDWSFLYSI LIAKASVFFI VCVLTLLVAS PDSRFSKAGL FPIFATQSND FALGYPIVEA L YQTTYPEY ...文字列: MGSMNSNLPA ENLTIAVNMT KTLPTAVTHG FNSTNDPPSM SITRLFPALL ECFGIVLCGY IAGRANVITS TQAKGLGNFV SRFALPALL FKNMVVLNFS NVDWSFLYSI LIAKASVFFI VCVLTLLVAS PDSRFSKAGL FPIFATQSND FALGYPIVEA L YQTTYPEY LQYIYLVAPI SLMMLNPIGF IFCEIQKWKD TQNASQNKIK IVGLGLLRVL QNPIVFMVFI GIAFNFILDR KV PVYVENF LDGLGNSFSG SALFYLGLTM VGKIKRLKKS AFVVLILLIT AKLLVLPLLC REMVELLDKG DSVVNHTSLS NYA FLYGVF PVAPGVAIFA TQFNMEVEII TSGMVISTFV SAPIMYVSAW LLTFPTMDPK PLAYAIQNVS FDISIVSLIS LIWS LAILL LSKKYKQLPH MLTTNLLIAQ SIVCAGMMIW NFVKEKNFVG QILVFVLLYS SLYSTYLWTG LLAISLFLLK KRERV QIPV GIIIISGWGI PALLVGVLLI TGKHNGDSID SAFFYGKEQM ITTAVTLFCS ILIAGISLMC MNQTAQAGSY EGFDQS QSH KVVEPGNTAF EESPAPVNEP ELFTSSIPET SCCSCSMGNG ELHCPSIEPI ANTSTSEPVI PSFEKNNHCV SRCNSQS CI LAQEEEQYLQ SGDQQLTRHV LLCLLLIIGL FANLSSCLWW LFNQEPGRLY VELQFFCAVF NFGQGFISFG IFGLDKHL I ILPFKRRLEF LWNNKDTAEN RDSPVSEEIK MTCQQFIHYH RDLCIRNIVK ERRCGAKTSA GTFCGCDLVS WLIEVGLAS DRGEAVIYGD RLVQGGVIQH ITNEYEFRDE YLFYRFLQKS PEQSPPAINA NTLQQERYKE IEHSSPPSHS PKTGTLEVLF QGPGGSGGS ASVIKPEMKI KLRMEGAVNG HKFVIEGEGI GKPYEGTQTL DLTVEEGAPL PFSYDILTPA FQYGNRAFTK Y PEDIPDYF KQAFPEGYSW ERSMTYEDQG ICIATSDITM EGDCFFYEIR FDGTNFPPNG PVMQKKTLKW EPSTEKMYVE DG VLKGDVE MALLLEGGGH YRCDFKTTYK AKKDVRLPDA HEVDHRIEIL SHDKDYNKVR LYEHAEARYS GGGSGGGSAW SHP QFEKGG GSGGGSGGSA WSHPQFEK UniProtKB: Lysosomal cholesterol signaling protein |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 3 / コピー数: 6 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 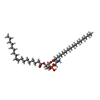 ChemComp-6OU: |
-分子 #4: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 4 / コピー数: 2 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #5: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (4~{Z},7~{Z},10~{Z},13~{Z},16~{Z},19~{Z})-docosa-4,7,10,13,16,19-hexaenoate タイプ: ligand / ID: 5 / コピー数: 2 / 式: A1L1C |
|---|---|
| 分子量 | 理論値: 764.023 Da |
-分子 #6: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 6 / コピー数: 2 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #7: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 7 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 2 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: 0.001 % LMNG, 150 mM NaCl, 0.2 mM TECP, 50 mM Tris HCl pH 8.0 | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 50 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.026000000000000002 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-8z8z: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































