[English] 日本語
 Yorodumi
Yorodumi- EMDB-39671: Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 comp... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex from a marine photoheterotrophic bacterium, purified with magnesium-free solutions. | |||||||||
 Map data Map data | Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex from a marine photoheterotrophic bacterium | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Photosynthesis / Reaction center / Energy transfer / Photosynthetic bacteria | |||||||||
| Function / homology |  Function and homology information Function and homology informationorganelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / electron transfer activity / iron ion binding / heme binding / metal ion binding ...organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / electron transfer activity / iron ion binding / heme binding / metal ion binding / membrane / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Dinoroseobacter shibae DFL 12 = DSM 16493 (bacteria) Dinoroseobacter shibae DFL 12 = DSM 16493 (bacteria) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.7 Å | |||||||||
 Authors Authors | Chen JH / Zheng Q / Zhang X | |||||||||
| Funding support |  China, 1 items China, 1 items
| |||||||||
 Citation Citation |  Journal: Adv Sci (Weinh) / Year: 2025 Journal: Adv Sci (Weinh) / Year: 2025Title: Cryo-EM Analysis of a Tri-Heme Cytochrome-Associated RC-LH1 Complex from the Marine Photoheterotrophic Bacterium Dinoroseobacter Shibae. Authors: Weiwei Wang / Yanting Liu / Jiayi Gu / Shaoya An / Cheng Ma / Haichun Gao / Nianzhi Jiao / Jian-Ren Shen / John Thomas Beatty / Michal Koblížek / Xing Zhang / Qiang Zheng / Jing-Hua Chen /    Abstract: The reaction center-light harvesting 1 (RC-LH1) complex converts solar energy into electrical energy, driving the initiation of photosynthesis. The authors present a cryo-electron microscopy ...The reaction center-light harvesting 1 (RC-LH1) complex converts solar energy into electrical energy, driving the initiation of photosynthesis. The authors present a cryo-electron microscopy structure of the RC-LH1 isolated from a marine photoheterotrophic bacterium Dinoroseobacter shibae. The RC comprises four subunits, including a three-heme cytochrome (Cyt) c protein, and is surrounded by a closed LH ring composed of 17 pairs of antenna subunits. Notably, a novel subunit with an N-terminal "helix-turn-helix" motif embedded in the gap between the RC and the LH ring is identified. The purified RC-LH1 complex exhibits high stability in solutions containing Mg or Ca. The periplasmic Cyt c is predicted to bind at the junction between the Cyt subunit and the membrane plane, enabling electron transfer from Cyt c to the proximal heme of the tri-heme Cyt, and subsequently to the special pair of bacteriochlorophylls. These findings provide structural insights into the efficient energy and electron transfer processes within a distinct type of RC-LH1, and shed light on evolutionary adaptations of photosynthesis. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_39671.map.gz emd_39671.map.gz | 32.3 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-39671-v30.xml emd-39671-v30.xml emd-39671.xml emd-39671.xml | 28 KB 28 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_39671.png emd_39671.png | 66.2 KB | ||
| Filedesc metadata |  emd-39671.cif.gz emd-39671.cif.gz | 7.8 KB | ||
| Others |  emd_39671_half_map_1.map.gz emd_39671_half_map_1.map.gz emd_39671_half_map_2.map.gz emd_39671_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39671 http://ftp.pdbj.org/pub/emdb/structures/EMD-39671 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39671 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39671 | HTTPS FTP |
-Validation report
| Summary document |  emd_39671_validation.pdf.gz emd_39671_validation.pdf.gz | 1.2 MB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_39671_full_validation.pdf.gz emd_39671_full_validation.pdf.gz | 1.2 MB | Display | |
| Data in XML |  emd_39671_validation.xml.gz emd_39671_validation.xml.gz | 12.5 KB | Display | |
| Data in CIF |  emd_39671_validation.cif.gz emd_39671_validation.cif.gz | 14.7 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39671 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39671 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39671 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39671 | HTTPS FTP |
-Related structure data
| Related structure data |  8yy9MC  8yz2C  9km0C M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_39671.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_39671.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex from a marine photoheterotrophic bacterium | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex...
| File | emd_39671_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex from a marine photoheterotrophic bacterium | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex...
| File | emd_39671_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Cryo-EM structure of a tri-heme cytochrome-associated RC-LH1 complex from a marine photoheterotrophic bacterium half-mapA | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
+Entire : tri-heme cytochrome-associated RC-LH1 complex
+Supramolecule #1: tri-heme cytochrome-associated RC-LH1 complex
+Macromolecule #1: Antenna pigment protein alpha chain
+Macromolecule #2: Reaction center protein O chain
+Macromolecule #3: Antenna pigment protein beta chain
+Macromolecule #4: Reaction center protein M chain
+Macromolecule #5: Reaction center protein L chain
+Macromolecule #6: Reaction center protein H chain
+Macromolecule #7: Photosynthetic reaction center cytochrome c subunit
+Macromolecule #8: BACTERIOCHLOROPHYLL A
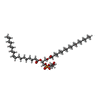
+Macromolecule #9: (4~{E},16~{E},26~{E})-2-methoxy-2,6,10,14,19,23,27,31-octamethyl-...
+Macromolecule #10: DODECYL-BETA-D-MALTOSIDE
+Macromolecule #11: (21R,24R,27S)-24,27,28-trihydroxy-18,24-dioxo-19,23,25-trioxa-24l...
+Macromolecule #12: FE (III) ION
+Macromolecule #13: BACTERIOPHEOPHYTIN A
+Macromolecule #14: UBIQUINONE-10
+Macromolecule #15: CARDIOLIPIN
+Macromolecule #16: HEME C
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 8 |
|---|---|
| Grid | Model: Quantifoil R1.2/1.3 / Material: GOLD / Mesh: 300 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 60 sec. |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 279 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.5 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)