+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of the trimeric HerA | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | antiphage system / IMMUNE SYSTEM | |||||||||
| Function / homology | SIR2-like domain / : / SIR2 family protein Function and homology information Function and homology information | |||||||||
| Biological species |  | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.81 Å | |||||||||
 Authors Authors | Zhen X / Zhou B / Xiong X | |||||||||
| Funding support |  China, 1 items China, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2024 Journal: Nat Commun / Year: 2024Title: Mechanistic basis for the allosteric activation of NADase activity in the Sir2-HerA antiphage defense system. Authors: Xiangkai Zhen / Biao Zhou / Zihe Liu / Xurong Wang / Heyu Zhao / Shuxian Wu / Zekai Li / Jiamin Liang / Wanyue Zhang / Qingjian Zhu / Jun He / Xiaoli Xiong / Songying Ouyang /  Abstract: Sir2-HerA is a widely distributed antiphage system composed of a RecA-like ATPase (HerA) and an effector with potential NADase activity (Sir2). Sir2-HerA is believed to provide defense against phage ...Sir2-HerA is a widely distributed antiphage system composed of a RecA-like ATPase (HerA) and an effector with potential NADase activity (Sir2). Sir2-HerA is believed to provide defense against phage infection in Sir2-dependent NAD depletion to arrest the growth of infected cells. However, the detailed mechanism underlying its antiphage activity remains largely unknown. Here, we report functional investigations of Sir2-HerA from Staphylococcus aureus (SaSir2-HerA), unveiling that the NADase function of SaSir2 can be allosterically activated by the binding of SaHerA, which then assembles into a supramolecular complex with NADase activity. By combining the cryo-EM structure of SaSir2-HerA in complex with the NAD cleavage product, it is surprisingly observed that Sir2 protomers that interact with HerA are in the activated state, which is due to the opening of the α15-helix covering the active site, allowing NAD to access the catalytic pocket for hydrolysis. In brief, our study provides a comprehensive view of an allosteric activation mechanism for Sir2 NADase activity in the Sir2-HerA immune system. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_39302.map.gz emd_39302.map.gz | 256.3 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-39302-v30.xml emd-39302-v30.xml emd-39302.xml emd-39302.xml | 15.6 KB 15.6 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_39302.png emd_39302.png | 42 KB | ||
| Masks |  emd_39302_msk_1.map emd_39302_msk_1.map | 512 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-39302.cif.gz emd-39302.cif.gz | 6 KB | ||
| Others |  emd_39302_half_map_1.map.gz emd_39302_half_map_1.map.gz emd_39302_half_map_2.map.gz emd_39302_half_map_2.map.gz | 475.1 MB 475.1 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39302 http://ftp.pdbj.org/pub/emdb/structures/EMD-39302 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39302 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39302 | HTTPS FTP |
-Related structure data
| Related structure data |  8yhxMC  8yhoC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_39302.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_39302.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.71 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_39302_msk_1.map emd_39302_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_39302_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_39302_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Cryo-Em structure of the trimeric HerA
| Entire | Name: Cryo-Em structure of the trimeric HerA |
|---|---|
| Components |
|
-Supramolecule #1: Cryo-Em structure of the trimeric HerA
| Supramolecule | Name: Cryo-Em structure of the trimeric HerA / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#2 |
|---|---|
| Source (natural) | Organism:  |
-Macromolecule #1: DUF87 domain-containing protein
| Macromolecule | Name: DUF87 domain-containing protein / type: protein_or_peptide / ID: 1 / Number of copies: 6 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 65.255168 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MHSIGKVTSV TFEKLIFEVS DFEKLNYNLL GQIYIAKGVI DYVTIKNEYS EKFIYQVVKV EDKEIPLSSE EHSKFKYHGR FECVPVGMI KHGKIEFNLK KYPFLQDKVY LTSQEEMEMV FSHFHNGNDI TIGLIDDQYP AYFNTAKLLT NHTAIIGNTG S GKSTTVRQ ...String: MHSIGKVTSV TFEKLIFEVS DFEKLNYNLL GQIYIAKGVI DYVTIKNEYS EKFIYQVVKV EDKEIPLSSE EHSKFKYHGR FECVPVGMI KHGKIEFNLK KYPFLQDKVY LTSQEEMEMV FSHFHNGNDI TIGLIDDQYP AYFNTAKLLT NHTAIIGNTG S GKSTTVRQ IISKINNLNT QNLHFHIFDV HDEYKDINGV KIVDVINDFK INIKNLEMQD WINLIKPSEL VQLPILQMGL KY ANAIENK IIEEEWLKCY IALSLYRNQQ TDAVTKRTKI LSILDGTNID TEKYDSKYGN MDSNTEKKFI ESLKNVVDNG GNI FTLSEV IEKAKYNVSS FNKLLEGLNY VFLLEESKGN NQARSYSATL ETRIKNVQTR FSNLFGNNDT ELEDKSIVYS VSEL DDDLL LFFTTFILKK EFEKNKKMKL EDRSVNVFIF EEAHRYISKF KESSQFNEVE AFKKIAREGR KFGCFLMLSS QRPSE LSST VLSQCNNYIV HRVKNNVDLE YLLNSIPYIN KFQLNRFSYL PTGTAYIVGE LFPIPVEIEI FEEFSKNSTI TPEIVY RS UniProtKB: UNIPROTKB: A0A844QRL0 |
-Macromolecule #2: SIR2 family protein
| Macromolecule | Name: SIR2 family protein / type: protein_or_peptide / ID: 2 / Number of copies: 12 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 50.710613 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MGIYHLNKDK DVLTDLKSNE KQEQVATFIN KHLSANNLTI FIGSGCSTGA VPLMSTTMKN ILEENESVLN YVKKFLNSKG IKEFIKYVE EQEQEKIQEK ERKALHTIMD QLEAENFKNL EEYSGWLDMQ DSEYKEEILN FLDCYYLNYS NIEELLNWIQ N GLHYDNNN ...String: MGIYHLNKDK DVLTDLKSNE KQEQVATFIN KHLSANNLTI FIGSGCSTGA VPLMSTTMKN ILEENESVLN YVKKFLNSKG IKEFIKYVE EQEQEKIQEK ERKALHTIMD QLEAENFKNL EEYSGWLDMQ DSEYKEEILN FLDCYYLNYS NIEELLNWIQ N GLHYDNNN GDLKDVFTTL KSEFIKTIPK VGDKEYSTET YEIYKDFYRY VFDKRTEQKS KVSIFTTNYD LFNEYALENN NI IYSTGIQ NTILKKFDIN QFKYRVVDDT NRYKEKWQPV SKEANLYKIH GSINWKSNEE GELQQIDFND EDDQVVIYPT MLK HKETAQ APYSELFREF SNCLQIKDTT LIIIGYGFPD EHINNIIAQN LKNQDFNLII FGDVKEENVK NFYDNFKNFN LHLI GGNSS KAEQKAHYFQ FIVENFLKNQ RRR UniProtKB: SIR2 family protein |
-Macromolecule #3: ADENOSINE-5-DIPHOSPHORIBOSE
| Macromolecule | Name: ADENOSINE-5-DIPHOSPHORIBOSE / type: ligand / ID: 3 / Number of copies: 6 / Formula: APR |
|---|---|
| Molecular weight | Theoretical: 559.316 Da |
| Chemical component information | 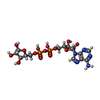 ChemComp-APR: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.4 µm / Nominal defocus min: 0.8 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| Startup model | Type of model: NONE |
|---|---|
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 2.81 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 79232 |
| Initial angle assignment | Type: MAXIMUM LIKELIHOOD |
| Final angle assignment | Type: MAXIMUM LIKELIHOOD |
 Movie
Movie Controller
Controller





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)












































