+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SARS-CoV-2 spike ectodomain (HexaPro, Omicron BA.5 variant) in complex with CeSPIACE, class 2 | ||||||||||||||||||
 マップデータ マップデータ | post-processed map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Spike protein / VIRAL PROTEIN-INHIBITOR COMPLEX | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.2 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Suzuki H / Nakamura S / Fujiyoshi Y | ||||||||||||||||||
| 資金援助 |  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Development of a short-peptide inhibiting any SARS-CoV-2 variants based on structural biology 著者: Nakamura S / Tanimura Y / Nomura R / Suzuki H / Nishikawa K / Kamegawa A / Numoto N / Tanaka A / Kawabata S / Sakaguchi S / Emi A / Suzuki Y / Fujiyoshi Y | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39186.map.gz emd_39186.map.gz | 140.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39186-v30.xml emd-39186-v30.xml emd-39186.xml emd-39186.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39186_fsc.xml emd_39186_fsc.xml | 12.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39186.png emd_39186.png | 67.4 KB | ||
| マスクデータ |  emd_39186_msk_1.map emd_39186_msk_1.map | 149.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-39186.cif.gz emd-39186.cif.gz | 7.4 KB | ||
| その他 |  emd_39186_half_map_1.map.gz emd_39186_half_map_1.map.gz emd_39186_half_map_2.map.gz emd_39186_half_map_2.map.gz | 118.3 MB 118.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39186 http://ftp.pdbj.org/pub/emdb/structures/EMD-39186 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39186 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39186 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39186_validation.pdf.gz emd_39186_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39186_full_validation.pdf.gz emd_39186_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_39186_validation.xml.gz emd_39186_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_39186_validation.cif.gz emd_39186_validation.cif.gz | 25.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39186 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39186 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39186 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39186 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ydzMC  8ydpC  8ydqC  8ydrC  8ydsC  8ydtC  8yduC  8ydvC  8ydwC  8ydxC  8ydyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39186.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39186.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post-processed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.44 Å | ||||||||||||||||||||||||||||||||||||
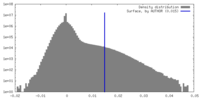
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_39186_msk_1.map emd_39186_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
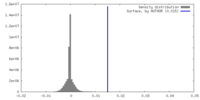
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_39186_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_39186_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike ectodomain (HexaPro, Omicron BA.5 variant) in co...
| 全体 | 名称: SARS-CoV-2 spike ectodomain (HexaPro, Omicron BA.5 variant) in complex with CeSPIACE |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike ectodomain (HexaPro, Omicron BA.5 variant) in co...
| 超分子 | 名称: SARS-CoV-2 spike ectodomain (HexaPro, Omicron BA.5 variant) in complex with CeSPIACE タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 詳細: the N-terminal sequence (from -18 to 12) is an mammalian secretion signal sequence, and the C-terminal sequence includes "T4 fibritin trimerization mottif" (Gly1211-Leu1237), "HRV 3C protease ...詳細: the N-terminal sequence (from -18 to 12) is an mammalian secretion signal sequence, and the C-terminal sequence includes "T4 fibritin trimerization mottif" (Gly1211-Leu1237), "HRV 3C protease cleavage site" (Leu1241-Pro1248), "8x His tag" (His1250-His1257) and "Twin-strep tag" (Trp1260-Lys1288) コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 144.193672 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GSQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF L GVYYHKNN ...文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GSQCVNLTTR TQLPPAYTNS FTRGVYYPDK VFRSSVLHST QDLFLPFFSN VTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF L GVYYHKNN KSWMESEFRV YSSANNCTFE YVSQPFLMDL EGKQGNFKNL REFVFKNIDG YFKIYSKHTP INLVRDLPQG FS ALEPLVD LPIGINITRF QTLLALHRSY LTPGDSSSGW TAGAAAYYVG YLQPRTFLLK YNENGTITDA VDCALDPLSE TKC TLKSFT VEKGIYQTSN FRVQPTESIV RFPNITNLCP FDEVFNATRF ASVYAWNRKR ISNCVADYSV LYNFAPFSAF KCYG VSPTK LNDLCFTNVY ADSFVIRGNE VSQIAPGQTG NIADYNYKLP DDFTGCVIAW NSNKLDSKVG GNYNYRYRLF RKSNL KPFE RDISTEIYQA GNKPCNGVAG VNCYFPLQSY GFRPTYGVGH QPYRVVVLSF ELLHAPATVC GPKKSTNLVK NKCVNF NFN GLTGTGVLTE SNKKFLPFQQ FGRDIADTTD AVRDPQTLEI LDITPCSFGG VSVITPGTNT SNQVAVLYQD VNCTEVP VA IHADQLTPTW RVYSTGSNVF QTRAGCLIGA EHVNNSYECD IPIGAGICAS YQTQTNSPGS ASSVASQSII AYTMSLGA E NSVAYSNNSI AIPTNFTISV TTEILPVSMT KTSVDCTMYI CGDSTECSNL LLQYGSFCTQ LNRALTGIAV EQDKNTQEV FAQVKQIYKT PPIKDFGGFN FSQILPDPSK PSKRSPIEDL LFNKVTLADA GFIKQYGDCL GDIAARDLIC AQKFNGLTVL PPLLTDEMI AQYTSALLAG TITSGWTFGA GPALQIPFPM QMAYRFNGIG VTQNVLYENQ KLIANQFNSA IGKIQDSLSS T PSALGKLQ DVVNQNAQAL NTLVKQLSSN FGAISSVLND ILSRLDPPEA EVQIDRLITG RLQSLQTYVT QQLIRAAEIR AS ANLAATK MSECVLGQSK RVDFCGKGYH LMSFPQSAPH GVVFLHVTYV PAQEKNFTTA PAICHDGKAH FPREGVFVSN GTH WFVTQR NFYEPQIITT DNTFVSGNCD VVIGIVNNTV YDPLQPELDS FKEELDKYFK NHTSPDVDLG DISGINASVV NIQK EIDRL NEVAKNLNES LIDLQELGKY EQGSGYIPEA PRDGQAYVRK DGEWVLLSTF LGRSLEVLFQ GPGHHHHHHH HSAWS HPQF EKGGGSGGGG SGGSAWSHPQ FEK UniProtKB: Spike glycoprotein |
-分子 #2: CeSPIACE
| 分子 | 名称: CeSPIACE / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.721475 KDa |
| 配列 | 文字列: DKLWILQKIY EIMVRLDEEG HGEASLMVSD LIYEFMKRD |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.42 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 69.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 3.4 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)