+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3897 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of S.aureus ClpC in complex with MecA | |||||||||
 マップデータ マップデータ | Low-resolution map of S.aureus ClpC in complex with MecA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Chaperone / AAA+ protein / unfoldase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報stress response to cadmium ion / stress response to copper ion / peptidase activity / cellular response to heat / protein-macromolecule adaptor activity / ATP hydrolysis activity / proteolysis / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Staphylococcus aureus (strain bovine RF122 / ET3-1) (黄色ブドウ球菌) Staphylococcus aureus (strain bovine RF122 / ET3-1) (黄色ブドウ球菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.0 Å | |||||||||
 データ登録者 データ登録者 | Carroni M / Mogk A | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: Regulatory coiled-coil domains promote head-to-head assemblies of AAA+ chaperones essential for tunable activity control. 著者: Marta Carroni / Kamila B Franke / Michael Maurer / Jasmin Jäger / Ingo Hantke / Felix Gloge / Daniela Linder / Sebastian Gremer / Kürşad Turgay / Bernd Bukau / Axel Mogk /   要旨: Ring-forming AAA+ chaperones exert ATP-fueled substrate unfolding by threading through a central pore. This activity is potentially harmful requiring mechanisms for tight repression and substrate- ...Ring-forming AAA+ chaperones exert ATP-fueled substrate unfolding by threading through a central pore. This activity is potentially harmful requiring mechanisms for tight repression and substrate-specific activation. The AAA+ chaperone ClpC with the peptidase ClpP forms a bacterial protease essential to virulence and stress resistance. The adaptor MecA activates ClpC by targeting substrates and stimulating ClpC ATPase activity. We show how ClpC is repressed in its ground state by determining ClpC cryo-EM structures with and without MecA. ClpC forms large two-helical assemblies that associate via head-to-head contacts between coiled-coil middle domains (MDs). MecA converts this resting state to an active planar ring structure by binding to MD interaction sites. Loss of ClpC repression in MD mutants causes constitutive activation and severe cellular toxicity. These findings unravel an unexpected regulatory concept executed by coiled-coil MDs to tightly control AAA+ chaperone activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3897.map.gz emd_3897.map.gz | 5.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3897-v30.xml emd-3897-v30.xml emd-3897.xml emd-3897.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3897_fsc.xml emd_3897_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3897.png emd_3897.png | 118.4 KB | ||
| マスクデータ |  emd_3897_msk_1.map emd_3897_msk_1.map | 67 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-3897.cif.gz emd-3897.cif.gz | 6.3 KB | ||
| その他 |  emd_3897_half_map_1.map.gz emd_3897_half_map_1.map.gz emd_3897_half_map_2.map.gz emd_3897_half_map_2.map.gz | 52.1 MB 52.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3897 http://ftp.pdbj.org/pub/emdb/structures/EMD-3897 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3897 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3897 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3897.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3897.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Low-resolution map of S.aureus ClpC in complex with MecA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
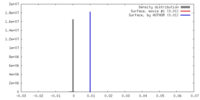
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_3897_msk_1.map emd_3897_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
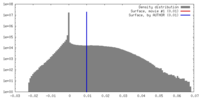
| 密度ヒストグラム |
-ハーフマップ: Low-resolution map of S.aureus ClpC in complex with MecA, half mapA
| ファイル | emd_3897_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Low-resolution map of S.aureus ClpC in complex with MecA, half mapA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Low-resolution map of S.aureus ClpC in complex with MecA, half mapB
| ファイル | emd_3897_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Low-resolution map of S.aureus ClpC in complex with MecA, half mapB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ClpC in complex with MecA from S. aureus
| 全体 | 名称: ClpC in complex with MecA from S. aureus |
|---|---|
| 要素 |
|
-超分子 #1: ClpC in complex with MecA from S. aureus
| 超分子 | 名称: ClpC in complex with MecA from S. aureus / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: ATP-dependent Clp protease ATP-binding subunit
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.325732 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KLTKEELKEI VTMMVNKLTN RLSEQNINII VTDKAKDKIA EEGYDPEYGA RPLIRAIQKT IEDNLSELIL DGNQIEGKKV TV UniProtKB: ATP-dependent Clp protease ATP-binding subunit |
-分子 #2: ATP-dependent Clp protease ATP-binding subunit
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.18359 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LTKINETESE KLLSLEDTLH ERVIGQKDAV NSISKAVRRA RAGLKDPKRP IGSFIFLGPT GVGKTELARA LAESMFGDDD AMIRVDMSE FMEKHAVSRL VGAPPGYVGH DDGGQLTEKV RRKPYSVILF DEIEKAHPDV FNILLQVLDD GHLTDTKGRT V DFRNTIII ...文字列: LTKINETESE KLLSLEDTLH ERVIGQKDAV NSISKAVRRA RAGLKDPKRP IGSFIFLGPT GVGKTELARA LAESMFGDDD AMIRVDMSE FMEKHAVSRL VGAPPGYVGH DDGGQLTEKV RRKPYSVILF DEIEKAHPDV FNILLQVLDD GHLTDTKGRT V DFRNTIII MTSNVGAQEL QDQRFAGFGG SSDGQDYETI RKTMLKELKN SFRPEFLNRV DDIIVFH UniProtKB: UNIPROTKB: A0A4P9AXU9 |
-分子 #3: Class III stress response-related ATPase, AAA+ superfamily
| 分子 | 名称: Class III stress response-related ATPase, AAA+ superfamily タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 6.539123 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NNLKEIEQEI EKVKNEKDAA VHAQEFENAA NLRDKQTKLE KQYEEAKNEW KNAQN UniProtKB: ATP-dependent Clp protease ATP-binding subunit ClpC |
-分子 #4: ATP-dependent Clp protease ATP-binding subunit ClpC
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit ClpC / タイプ: protein_or_peptide / ID: 4 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.42625 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SVVDTVAILK GLRDRYEAHH RINISDEAIE AAVKLSNRYV SDRFLPDKAI DLIDEASSKV RLKSHTTPNN LKEIEQEIEK VKNEKDAAV HAQEFENAAN LRDKQTKLEK QYEEAKNEWK NAQNGMSTSL SEEDIAEVIA GWTGIP UniProtKB: ATP-dependent Clp protease ATP-binding subunit ClpC |
-分子 #5: ATP-dependent Clp protease ATP-binding subunit ClpC
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit ClpC / タイプ: protein_or_peptide / ID: 5 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus aureus (strain bovine RF122 / ET3-1) (黄色ブドウ球菌) Staphylococcus aureus (strain bovine RF122 / ET3-1) (黄色ブドウ球菌)株: bovine RF122 / ET3-1 |
| 分子量 | 理論値: 19.699514 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TLDSLARDLT VIAKDGTLDP VIGRDKEITR VIEVLSRRTK NNPVLIGEPG VGKTAIAEGL AQAIVNNEVP ETLKDKRVMS LDMGTVVAG TKYRGEFEER LKKVMEEIQQ AGNVILFIDE LHTLVGAGGA EGAIDASNIL KPALARGELQ CIGATTLDEY R KNIEKDAA LERRFQPVQV DEP UniProtKB: ATP-dependent Clp protease ATP-binding subunit ClpC |
-分子 #6: ATP-dependent Clp protease ATP-binding subunit ClpC
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit ClpC / タイプ: protein_or_peptide / ID: 6 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus aureus (strain bovine RF122 / ET3-1) (黄色ブドウ球菌) Staphylococcus aureus (strain bovine RF122 / ET3-1) (黄色ブドウ球菌)株: bovine RF122 / ET3-1 |
| 分子量 | 理論値: 17.446889 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: RLTERAQRVL AHAQEEAIRL NHSNIGTEHL LLGLMKEPEG IAAKVLESFN ITEDKVIEEV EKLIGHGQDH VGTLHYTPRA KKVIELSMD EARKLHHNFV GTEHILLGLI RENEGVAARV FANLDLNITK ARAQVVKALG NPEMSNKNAQ ASKSNNTP UniProtKB: ATP-dependent Clp protease ATP-binding subunit ClpC |
-分子 #7: Adapter protein MecA
| 分子 | 名称: Adapter protein MecA / タイプ: protein_or_peptide / ID: 7 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 10.758939 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRIERVDDTT VKLFITYSDI EARGFSREDL WTNRKRGEEF FWSMMDEINE EEDFVVEGPL WIQVHAFEKG VEVTISKSKN EDMMNMSDD D UniProtKB: Adapter protein MecA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6emw: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)