+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The Cryo-EM structure of MPXV E5 C-terminal in complex with DNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | helicase / REPLICATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helicase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / host cell cytoplasm / hydrolase activity / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) | |||||||||
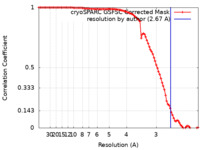
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.67 Å | |||||||||
 データ登録者 データ登録者 | Zhang W / Liu Y / Gao H / Gan J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Discov / 年: 2024 ジャーナル: Cell Discov / 年: 2024タイトル: Structural and functional insights into the helicase protein E5 of Mpox virus. 著者: Weizhen Zhang / Yusong Liu / Mengquan Yang / Jie Yang / Zhiwei Shao / Yanqing Gao / Xinran Jiang / Ruixue Cui / Yixi Zhang / Xin Zhao / Qiyuan Shao / Chulei Cao / Huili Li / Linxi Li / Hehua ...著者: Weizhen Zhang / Yusong Liu / Mengquan Yang / Jie Yang / Zhiwei Shao / Yanqing Gao / Xinran Jiang / Ruixue Cui / Yixi Zhang / Xin Zhao / Qiyuan Shao / Chulei Cao / Huili Li / Linxi Li / Hehua Liu / Haishan Gao / Jianhua Gan /  要旨: Mpox virus (MPXV) can cause mpox in humans. Due to its quick and wide spread in the past two years, mpox has turned into a significant public health concern. Helicase E5 is a multi-domain protein; ...Mpox virus (MPXV) can cause mpox in humans. Due to its quick and wide spread in the past two years, mpox has turned into a significant public health concern. Helicase E5 is a multi-domain protein; its primer synthesis and DNA unwinding activity are required for genome uncoating and DNA replication of MPXV. However, the in vitro DNA unwinding activity has never been demonstrated. Here, we report the structural and biochemical studies of MPXV E5, showing that the full-length protein adopts an auto-inhibited conformation. Truncation of the N-terminus can recover the in vitro unwinding activity of E5 towards the forked DNA. Further structural analysis reveals that MPXV E5 shares a conserved mechanism in DNA unwinding and primer synthesis with the homologous proteins. These findings not only advance our understanding on the function of MPXV E5, but also provide a solid basis for the development of anti-poxvirus drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38396.map.gz emd_38396.map.gz | 79 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38396-v30.xml emd-38396-v30.xml emd-38396.xml emd-38396.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_38396_fsc.xml emd_38396_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_38396.png emd_38396.png | 26.3 KB | ||
| Filedesc metadata |  emd-38396.cif.gz emd-38396.cif.gz | 5.8 KB | ||
| その他 |  emd_38396_half_map_1.map.gz emd_38396_half_map_1.map.gz emd_38396_half_map_2.map.gz emd_38396_half_map_2.map.gz | 77.6 MB 77.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38396 http://ftp.pdbj.org/pub/emdb/structures/EMD-38396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38396 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38396_validation.pdf.gz emd_38396_validation.pdf.gz | 919 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38396_full_validation.pdf.gz emd_38396_full_validation.pdf.gz | 918.6 KB | 表示 | |
| XML形式データ |  emd_38396_validation.xml.gz emd_38396_validation.xml.gz | 17.5 KB | 表示 | |
| CIF形式データ |  emd_38396_validation.cif.gz emd_38396_validation.cif.gz | 22.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38396 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38396 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38396 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38396 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38396.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38396.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.081 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_38396_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_38396_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Monkeypox virus
| 全体 | 名称:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Monkeypox virus
| 超分子 | 名称: Monkeypox virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / NCBI-ID: 10244 / 生物種: Monkeypox virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|
-分子 #1: DNA (70-MER)
| 分子 | 名称: DNA (70-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 21.440668 KDa |
| 配列 | 文字列: (DA)(DA)(DC)(DG)(DA)(DG)(DT)(DC)(DA)(DA) (DG)(DC)(DG)(DC)(DA)(DT)(DC)(DC)(DC)(DG) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT)(DT)(DT) ...文字列: (DA)(DA)(DC)(DG)(DA)(DG)(DT)(DC)(DA)(DA) (DG)(DC)(DG)(DC)(DA)(DT)(DC)(DC)(DC)(DG) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DC)(DG)(DG)(DG)(DA)(DT)(DG)(DC) (DG)(DC) (DT)(DT)(DG)(DA)(DC)(DT)(DC) (DG)(DT)(DT) |
-分子 #2: Monkeypox virus E5
| 分子 | 名称: Monkeypox virus E5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 53.282012 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GNKLFNIAQR ILDTNSVLLT ERGDHIVWIN NSWKFNSEEP LITKLILSIR HQLPKEYSSE LLCPRKRKTV EANIRDMLVD SVETDTYPD KLPFKNGVLD LVDGMFYSGD DAKKYTCTVS TGFKFDDTKF VEDSPEMEEL MNIINDIQPL TDENKKNREL Y EKTLSSCL ...文字列: GNKLFNIAQR ILDTNSVLLT ERGDHIVWIN NSWKFNSEEP LITKLILSIR HQLPKEYSSE LLCPRKRKTV EANIRDMLVD SVETDTYPD KLPFKNGVLD LVDGMFYSGD DAKKYTCTVS TGFKFDDTKF VEDSPEMEEL MNIINDIQPL TDENKKNREL Y EKTLSSCL CGATKGCLTF FFGETATGKS TTKRLLKSAI GDLFVETGQT ILTDVLDKGP NPFIANMHLK RSVFCSELPD FA CSGSKKI RSDNIKKLTE PCVIGRPCFS NKINNRNHAT IIIDTNYKPV FDRIDNALMR RIAVVRFRTH FSQPSGREAA ENN DAYDKV KLLDEGLDGK IQNNRYRFAF LYLLVKWYKK YHIPIMKLYP TPEEIPDFAF YLKIGTLLVS SSVKHIPLMT DLSK KGYIL YDNVVTLPLT TFQQKISKYF NSRLFGHDIE SFINRHKKFA NVSDEYLQYI FIEDISSP UniProtKB: Uncoating factor OPG117 |
-分子 #3: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 3 / コピー数: 3 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)