+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
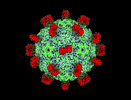
| タイトル | Cryo-EM structure of coxsackievirus A16 mature virion in complex with Fab h1A6.2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cryo-EM / virus / coxsackievirus A16 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont genome entry into host cell via pore formation in plasma membrane / viral capsid / host cell cytoplasm / symbiont-mediated suppression of host gene expression / virion attachment to host cell / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |    Coxsackievirus A16 (コクサッキーウイルス) Coxsackievirus A16 (コクサッキーウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.69 Å | |||||||||
 データ登録者 データ登録者 | Jiang Y / Huang Y / Zhu R / Zheng Q / Li S / Xia N | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2024 ジャーナル: Nat Microbiol / 年: 2024タイトル: Broadly therapeutic antibody provides cross-serotype protection against enteroviruses via Fc effector functions and by mimicking SCARB2. 著者: Rui Zhu / Yuanyuan Wu / Yang Huang / Yanan Jiang / Yichao Jiang / Dongqing Zhang / Hui Sun / Zhenhong Zhou / Lizhi Zhou / Shihan Weng / Hao Chen / Xiaoqing Chen / Wenjing Ning / Yuxiang Zou / ...著者: Rui Zhu / Yuanyuan Wu / Yang Huang / Yanan Jiang / Yichao Jiang / Dongqing Zhang / Hui Sun / Zhenhong Zhou / Lizhi Zhou / Shihan Weng / Hao Chen / Xiaoqing Chen / Wenjing Ning / Yuxiang Zou / Maozhou He / Hongwei Yang / Weixi Deng / Yu Li / Zhenqin Chen / Xiangzhong Ye / Jinle Han / Zhichao Yin / Huan Zhao / Che Liu / Yuqiong Que / Mujin Fang / Hai Yu / Jun Zhang / Wenxin Luo / Shaowei Li / Qingbing Zheng / Longfa Xu / Ningshao Xia / Tong Cheng /  要旨: Enteroviruses contain multiple serotypes and can cause severe neurological complications. The intricate life cycle of enteroviruses involving dynamic virus-receptor interaction hampers the ...Enteroviruses contain multiple serotypes and can cause severe neurological complications. The intricate life cycle of enteroviruses involving dynamic virus-receptor interaction hampers the development of broad therapeutics and vaccines. Here, using function-based screening, we identify a broadly therapeutic antibody h1A6.2 that potently protects mice in lethal models of infection with both enterovirus A71 and coxsackievirus A16 through multiple mechanisms, including inhibition of the virion-SCARB2 interactions and monocyte/macrophage-dependent Fc effector functions. h1A6.2 mitigates inflammation and improves intramuscular mechanics, which are associated with diminished innate immune signalling and preserved tissue repair. Moreover, cryogenic electron microscopy structures delineate an adaptive binding of h1A6.2 to the flexible and dynamic nature of the VP2 EF loop with a binding angle mimicking the SCARB2 receptor. The coordinated binding mode results in efficient binding of h1A6.2 to all viral particle types and facilitates broad neutralization of enterovirus, therefore informing a promising target for the structure-guided design of pan-enterovirus vaccine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38168.map.gz emd_38168.map.gz | 632.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38168-v30.xml emd-38168-v30.xml emd-38168.xml emd-38168.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_38168.png emd_38168.png | 94.6 KB | ||
| Filedesc metadata |  emd-38168.cif.gz emd-38168.cif.gz | 6.5 KB | ||
| その他 |  emd_38168_half_map_1.map.gz emd_38168_half_map_1.map.gz emd_38168_half_map_2.map.gz emd_38168_half_map_2.map.gz | 621.9 MB 621.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38168 http://ftp.pdbj.org/pub/emdb/structures/EMD-38168 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38168 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38168 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_38168_validation.pdf.gz emd_38168_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_38168_full_validation.pdf.gz emd_38168_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_38168_validation.xml.gz emd_38168_validation.xml.gz | 20.3 KB | 表示 | |
| CIF形式データ |  emd_38168_validation.cif.gz emd_38168_validation.cif.gz | 24.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38168 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38168 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38168 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-38168 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8x98MC  8x95C  8x96C  8x97C  8x99C  8x9aC  8x9bC  8ytbC  8ytjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38168.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38168.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_38168_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
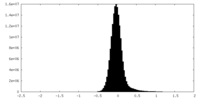
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_38168_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
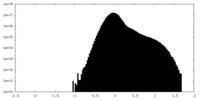
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Coxsackievirus A16 mature virion in complex with Fab h1A6.2
| 全体 | 名称: Coxsackievirus A16 mature virion in complex with Fab h1A6.2 |
|---|---|
| 要素 |
|
-超分子 #1: Coxsackievirus A16 mature virion in complex with Fab h1A6.2
| 超分子 | 名称: Coxsackievirus A16 mature virion in complex with Fab h1A6.2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #3: The Fab of h1A6.2
| 超分子 | 名称: The Fab of h1A6.2 / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: Coxsackievirus A16
| 超分子 | 名称: Coxsackievirus A16 / タイプ: virus / ID: 2 / 親要素: 1 / 含まれる分子: all / NCBI-ID: 31704 / 生物種: Coxsackievirus A16 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus A16 (コクサッキーウイルス) Coxsackievirus A16 (コクサッキーウイルス) |
| 分子量 | 理論値: 7.551226 KDa |
| 配列 | 文字列: MGSQVSTQRS GSHENSNSAS EGSTINYTTI NYYKDAYAAS AGRQDMSQDP KKFTDPVMDV IHEMAPPLK UniProtKB: Genome polyprotein |
-分子 #2: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus A16 (コクサッキーウイルス) Coxsackievirus A16 (コクサッキーウイルス) |
| 分子量 | 理論値: 33.106352 KDa |
| 配列 | 文字列: GDPIADMIDQ TVNNQVNRSL TALQVLPTAA NTEASSHRLG TGVVPALQAA ETGASSNASD KNLIETRCVL NHHSTQETAI GNFFSRAGL VSIITMPTTD TQNTDGYVNW DIDLMGYAQL RRKCELFTYM RFDAEFTFVV AKPNGVLVPQ LLQYMYVPPG A PKPTSRDS ...文字列: GDPIADMIDQ TVNNQVNRSL TALQVLPTAA NTEASSHRLG TGVVPALQAA ETGASSNASD KNLIETRCVL NHHSTQETAI GNFFSRAGL VSIITMPTTD TQNTDGYVNW DIDLMGYAQL RRKCELFTYM RFDAEFTFVV AKPNGVLVPQ LLQYMYVPPG A PKPTSRDS FAWQTATNPS VFVKMTDPPA QVSVPFMSPA SAYQWFYDGY PTFGEHLQAN DLDYGQCPNN MMGTFSIRTV GT EKSPHSI TLRVYMRIKH VRAWIPRPLR NQPYLFKTNP NYKGNDIKCT STSRDKITTL UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus A16 (コクサッキーウイルス) Coxsackievirus A16 (コクサッキーウイルス) |
| 分子量 | 理論値: 27.557104 KDa |
| 配列 | 文字列: SPSAEACGYS DRVAQLTIGN STITTQEAAN IVIAYGEWPE YCPDTDATAV DKPTRPDVSV NRFFTLDTKS WAKDSKGWYW KFPDVLTEV GVFGQNAQFH YLYRSGFCVH VQCNASKFHQ GALLVAVLPE YVLGTIAGGT GNENSHPPYA TTQPGQVGAV L THPYVLDA ...文字列: SPSAEACGYS DRVAQLTIGN STITTQEAAN IVIAYGEWPE YCPDTDATAV DKPTRPDVSV NRFFTLDTKS WAKDSKGWYW KFPDVLTEV GVFGQNAQFH YLYRSGFCVH VQCNASKFHQ GALLVAVLPE YVLGTIAGGT GNENSHPPYA TTQPGQVGAV L THPYVLDA GIPLSQLTVC PHQWINLRTN NCATIIVPYM NTVPFDSALN HCNFGLLVIP VVPLDFNAGA TSEIPITVTI AP MCAEFAG LRQAVKQ UniProtKB: Genome polyprotein |
-分子 #4: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Coxsackievirus A16 (コクサッキーウイルス) Coxsackievirus A16 (コクサッキーウイルス) |
| 分子量 | 理論値: 26.654295 KDa |
| 配列 | 文字列: GIPTELKPGT NQFLTTDDGV SAPILPGFHP TPPIHIPGEV HNLLEICRVE TILEVNNLKT NETTPMQRLC FPVSVQSKTG ELCAAFRAD PGRDGPWQST ILGQLCRYYT QWSGSLEVTF MFAGSFMATG KMLIAYTPPG GNVPADRITA MLGTHVIWDF G LQSSVTLV ...文字列: GIPTELKPGT NQFLTTDDGV SAPILPGFHP TPPIHIPGEV HNLLEICRVE TILEVNNLKT NETTPMQRLC FPVSVQSKTG ELCAAFRAD PGRDGPWQST ILGQLCRYYT QWSGSLEVTF MFAGSFMATG KMLIAYTPPG GNVPADRITA MLGTHVIWDF G LQSSVTLV VPWISNTHYR AHARAGYFDY YTTGIITIWY QTNYVVPIGA PTTAYIVALA AAQDNFTMKL CKDTEDIEQT AN IQ UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源: OTHER |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)