[日本語] English
 万見
万見- EMDB-37910: Structure of the SARS-CoV-2 BA.2.86 spike glycoprotein (closed state) -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the SARS-CoV-2 BA.2.86 spike glycoprotein (closed state) | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | spike protein / glycoprotein / VIRUS / VIRAL PROTEIN | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  | |||||||||||||||||||||
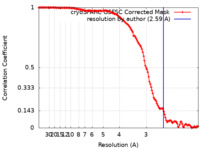
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.59 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Yajima H / Anraku Y / Kita S / Kimura K / Maenaka K / Hashiguchi T | |||||||||||||||||||||
| 資金援助 |  日本, 6件 日本, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for receptor-binding domain mobility of the spike in SARS-CoV-2 BA.2.86 and JN.1. 著者: Hisano Yajima / Yuki Anraku / Yu Kaku / Kanako Terakado Kimura / Arnon Plianchaisuk / Kaho Okumura / Yoshiko Nakada-Nakura / Yusuke Atarashi / Takuya Hemmi / Daisuke Kuroda / Yoshimasa ...著者: Hisano Yajima / Yuki Anraku / Yu Kaku / Kanako Terakado Kimura / Arnon Plianchaisuk / Kaho Okumura / Yoshiko Nakada-Nakura / Yusuke Atarashi / Takuya Hemmi / Daisuke Kuroda / Yoshimasa Takahashi / Shunsuke Kita / Jiei Sasaki / Hiromi Sumita / / Jumpei Ito / Katsumi Maenaka / Kei Sato / Takao Hashiguchi /   要旨: Since 2019, SARS-CoV-2 has undergone mutations, resulting in pandemic and epidemic waves. The SARS-CoV-2 spike protein, crucial for cellular entry, binds to the ACE2 receptor exclusively when its ...Since 2019, SARS-CoV-2 has undergone mutations, resulting in pandemic and epidemic waves. The SARS-CoV-2 spike protein, crucial for cellular entry, binds to the ACE2 receptor exclusively when its receptor-binding domain (RBD) adopts the up-conformation. However, whether ACE2 also interacts with the RBD in the down-conformation to facilitate the conformational shift to RBD-up remains unclear. Herein, we present the structures of the BA.2.86 and the JN.1 spike proteins bound to ACE2. Notably, we successfully observed the ACE2-bound down-RBD, indicating an intermediate structure before the RBD-up conformation. The wider and mobile angle of RBDs in the up-state provides space for ACE2 to interact with the down-RBD, facilitating the transition to the RBD-up state. The K356T, but not N354-linked glycan, contributes to both of infectivity and neutralizing-antibody evasion in BA.2.86. These structural insights the spike-protein dynamics would help understand the mechanisms underlying SARS-CoV-2 infection and its neutralization. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37910.map.gz emd_37910.map.gz | 108.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37910-v30.xml emd-37910-v30.xml emd-37910.xml emd-37910.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37910_fsc.xml emd_37910_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37910.png emd_37910.png | 102.3 KB | ||
| マスクデータ |  emd_37910_msk_1.map emd_37910_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-37910.cif.gz emd-37910.cif.gz | 7.6 KB | ||
| その他 |  emd_37910_additional_1.map.gz emd_37910_additional_1.map.gz emd_37910_half_map_1.map.gz emd_37910_half_map_1.map.gz emd_37910_half_map_2.map.gz emd_37910_half_map_2.map.gz | 108.1 MB 200.7 MB 200.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37910 http://ftp.pdbj.org/pub/emdb/structures/EMD-37910 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37910 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37910 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37910_validation.pdf.gz emd_37910_validation.pdf.gz | 891.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37910_full_validation.pdf.gz emd_37910_full_validation.pdf.gz | 891.3 KB | 表示 | |
| XML形式データ |  emd_37910_validation.xml.gz emd_37910_validation.xml.gz | 21.6 KB | 表示 | |
| CIF形式データ |  emd_37910_validation.cif.gz emd_37910_validation.cif.gz | 28.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37910 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37910 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37910 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37910 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8wxlMC  8xuxC  8xuyC  8xuzC  8xv0C  8xv1C  8xvmC  9iu1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37910.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37910.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.005 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
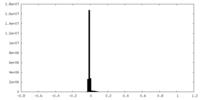
-マスク #1
| ファイル |  emd_37910_msk_1.map emd_37910_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
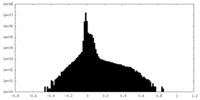
-追加マップ: local refinement closed
| ファイル | emd_37910_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local refinement closed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
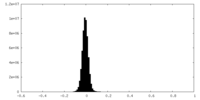
-ハーフマップ: #2
| ファイル | emd_37910_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
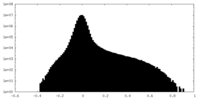
-ハーフマップ: #1
| ファイル | emd_37910_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 BA.2.86 spike glycoprotein
| 全体 | 名称: SARS-CoV-2 BA.2.86 spike glycoprotein |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 BA.2.86 spike glycoprotein
| 超分子 | 名称: SARS-CoV-2 BA.2.86 spike glycoprotein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 420 KDa |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 138.306547 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LLMGCVAETG SSQCVMPLFN LITTTQSYTN SFTRGVYYPD KVFRSSVLHL TQDLFLPFFS NVTWFHAISG TNGTKRFDNP VLPFNDGVY FASTEKSNII RGWIFGTTLD SKTQSLLIVN NATNVFIKVC EFQFCNDPFL DVYHKNNKSW MESESGVYSS A NNCTFEYV ...文字列: LLMGCVAETG SSQCVMPLFN LITTTQSYTN SFTRGVYYPD KVFRSSVLHL TQDLFLPFFS NVTWFHAISG TNGTKRFDNP VLPFNDGVY FASTEKSNII RGWIFGTTLD SKTQSLLIVN NATNVFIKVC EFQFCNDPFL DVYHKNNKSW MESESGVYSS A NNCTFEYV SQPFLMDLEG KQGNFKNLRE FVFKNIDGYF KIYSKHTPII GRDFPQGFSA LEPLVDLPIG INITRFQTLL AL NRSYLTP GDSSSGWTAG AADYYVGYLQ PRTFLLKYNE NGTITDAVDC ALDPLSETKC TLKSFTVEKG IYQTSNFRVQ PTE SIVRFP NVTNLCPFHE VFNATRFASV YAWNRTRISN CVADYSVLYN FAPFFAFKCY GVSPTKLNDL CFTNVYADSF VIKG NEVSQ IAPGQTGNIA DYNYKLPDDF TGCVIAWNSN KLDSKHSGNY DYWYRLFRKS KLKPFERDIS TEIYQAGNKP CKGKG PNCY FPLQSYGFRP TYGVGHQPYR VVVLSFELLH APATVCGPKK STNLVKNKCV NFNFNGLTGT GVLTKSNKKF LPFQQF GRD IVDTTDAVRD PQTLEILDIT PCSFGGVSVI TPGTNTSNQV AVLYQGVNCT EVSVAIHADQ LTPTWRVYST GSNVFQT RA GCLIGAEYVN NSYECDIPIG AGICASYQTQ TKSRGSAGSV ASQSIIAYTM SLGAENSVAY SNNSIAIPTN FTISVTTE I LPVSMTKTSV DCTMYICGDS TECSNLLLQY GSFCTQLKRA LTGIAVEQDK NTQEVFAQVK QIYKTPPIKY FGGFNFSQI LPDPSKPSKR SPIEDLLFNK VTLADAGFIK QYGDCLGDIA ARDLICAQKF NGLTVLPPLL TDEMIAQYTS ALLAGTITSG WTFGAGPAL QIPFPMQMAY RFNGIGVTQN VLYENQKLIA NQFNSAIGKI QDSLFSTPSA LGKLQDVVNH NAQALNTLVK Q LSSKFGAI SSVLNDILSR LDPPEAEVQI DRLITGRLQS LQTYVTQQLI RAAEIRASAN LAATKMSECV LGQSKRVDFC GK GYHLMSF PQSAPHGVVF LHVTYVPAQE KNFTTAPAIC HDGKAHFPRE GVFVSNGTHW FVTQRNFYEP QIITTDNTFV SGN CDVVIG IVNNTVYDPL QLELDSFKEE LDKYFKNHTS PDVDLGDISG INASVVNIQK EIDRLNEVAK NLNESLIDLQ ELGK YEQYI ASSGYIPEAP RDGQAYVRKD GEWVLLSTFL EGTKHHHHHH UniProtKB: Spike glycoprotein |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 13 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV / 詳細: blotting time 5 s and blotting force 5.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 3500 / 平均露光時間: 1.5 sec. / 平均電子線量: 52.45 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)