+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of DDM1-nucleosome complex in the apo state | |||||||||
 マップデータ マップデータ | DDM1-nucleosome complex in the apo state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | complex / nucleosome / chromatin remodeling / structural protein-hydrolase-dna complex / GENE REGULATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA-mediated transformation / retrotransposition / chloroplast thylakoid / chromocenter / thylakoid / response to water deprivation / plant-type vacuole / plasmodesma / chloroplast stroma / plastid ...DNA-mediated transformation / retrotransposition / chloroplast thylakoid / chromocenter / thylakoid / response to water deprivation / plant-type vacuole / plasmodesma / chloroplast stroma / plastid / DNA methylation-dependent constitutive heterochromatin formation / DNA helicase activity / chloroplast / epigenetic regulation of gene expression / response to bacterium / response to wounding / structural constituent of chromatin / peroxisome / nucleosome / heterochromatin formation / DNA helicase / chromatin remodeling / protein heterodimerization activity / nucleolus / ATP hydrolysis activity / DNA binding / extracellular region / ATP binding / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.58 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Zhang Z / Du J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2024 ジャーナル: Nat Plants / 年: 2024タイトル: Molecular basis of chromatin remodelling by DDM1 involved in plant DNA methylation. 著者: Yue Liu / Zhihui Zhang / Hongmiao Hu / Wei Chen / Fan Zhang / Qian Wang / Changshi Wang / Kaige Yan / Jiamu Du /   要旨: Eukaryotic gene regulation occurs at the chromatin level, which requires changing the chromatin structure by a group of ATP-dependent DNA translocases-namely, the chromatin remodellers. In plants, ...Eukaryotic gene regulation occurs at the chromatin level, which requires changing the chromatin structure by a group of ATP-dependent DNA translocases-namely, the chromatin remodellers. In plants, chromatin remodellers function in various biological processes and possess both conserved and plant-specific components. DECREASE IN DNA METHYLATION 1 (DDM1) is a plant chromatin remodeller that plays a key role in the maintenance DNA methylation. Here we determined the structures of Arabidopsis DDM1 in complex with nucleosome in ADP-BeF-bound, ADP-bound and nucleotide-free conformations. We show that DDM1 specifically recognizes the H4 tail and nucleosomal DNA. The conformational differences between ADP-BeF-bound, ADP-bound and nucleotide-free DDM1 suggest a chromatin remodelling cycle coupled to ATP binding, hydrolysis and ADP release. This, in turn, triggers conformational changes in the DDM1-bound nucleosomal DNA, which alters the nucleosome structure and promotes DNA sliding. Together, our data reveal the molecular basis of chromatin remodelling by DDM1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
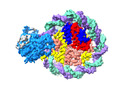
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37529.map.gz emd_37529.map.gz | 7.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37529-v30.xml emd-37529-v30.xml emd-37529.xml emd-37529.xml | 25.8 KB 25.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37529_fsc.xml emd_37529_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37529.png emd_37529.png | 97.9 KB | ||
| Filedesc metadata |  emd-37529.cif.gz emd-37529.cif.gz | 7.7 KB | ||
| その他 |  emd_37529_half_map_1.map.gz emd_37529_half_map_1.map.gz emd_37529_half_map_2.map.gz emd_37529_half_map_2.map.gz | 49.4 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37529 http://ftp.pdbj.org/pub/emdb/structures/EMD-37529 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37529 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37529 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37529_validation.pdf.gz emd_37529_validation.pdf.gz | 688.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37529_full_validation.pdf.gz emd_37529_full_validation.pdf.gz | 688.2 KB | 表示 | |
| XML形式データ |  emd_37529_validation.xml.gz emd_37529_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_37529_validation.cif.gz emd_37529_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37529 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37529 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37529 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37529 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37529.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37529.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DDM1-nucleosome complex in the apo state | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half1 map of DDM1-nucleosome complex in the apo state
| ファイル | emd_37529_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half1 map of DDM1-nucleosome complex in the apo state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
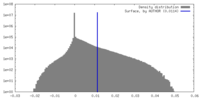
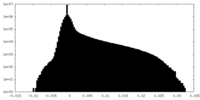
| 密度ヒストグラム |
-ハーフマップ: Half2 map of DDM1-nucleosome complex in the apo state
| ファイル | emd_37529_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half2 map of DDM1-nucleosome complex in the apo state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
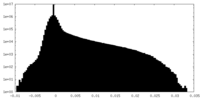
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DDM1-nucleosome complex in the apo state
| 全体 | 名称: DDM1-nucleosome complex in the apo state |
|---|---|
| 要素 |
|
-超分子 #1: DDM1-nucleosome complex in the apo state
| 超分子 | 名称: DDM1-nucleosome complex in the apo state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 290 KDa |
-分子 #1: Histone H3.1
| 分子 | 名称: Histone H3.1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.300968 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PATGGVKKPH RFRPGTVALR EIRKYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQSSA VAALQEAAEA YLVGLFEDTN LCAIHAKRVT IMPKDIQLAR RIRGERA UniProtKB: Histone H3.1 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.436467 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK IFLENVIRDA VTYTEHARRK TVTAMDVVY ALKRQGRTLY GFGG UniProtKB: Histone H4 |
-分子 #3: Histone H2A.6
| 分子 | 名称: Histone H2A.6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.680854 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGRGKTLGS GGAKKATSRS SKAGLQFPVG RIARFLKAGK YAERVGAGAP VYLAAVLEYL AAEVLELAGN AARDNKKTRI VPRHIQLAV RNDEELSKLL GDVTIANGGV MPNIHNLLLP KKAGASKPQE D UniProtKB: Histone H2A.6 |
-分子 #4: Histone H2B.6
| 分子 | 名称: Histone H2B.6 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.474459 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAPRAEKKPA EKKPAAEKPV EEKSKAEKAP AEKKPKAGKK LPKEAGAGGD KKKKMKKKSV ETYKIYIFKV LKQVHPDIGI SSKAMGIMN SFINDIFEKL ASESSKLARY NKKPTITSRE IQTAVRLVLP GELAKHAVSE GTKAVTKFTS S UniProtKB: Histone H2B.6 |
-分子 #7: ATP-dependent DNA helicase DDM1
| 分子 | 名称: ATP-dependent DNA helicase DDM1 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 86.844836 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SMVSLRSRKV IPASEMVSDG KTEKDASGDS PTSVLNEEEN CEEKSVTVVE EEILLAKNGD SSLISEAMAQ EEEQLLKLRE DEEKANNAG SAVAPNLNET QFTKLDELLT QTQLYSEFLL EKMEDITING IESESQKAEP EKTGRGRKRK AASQYNNTKA K RAVAAMIS ...文字列: SMVSLRSRKV IPASEMVSDG KTEKDASGDS PTSVLNEEEN CEEKSVTVVE EEILLAKNGD SSLISEAMAQ EEEQLLKLRE DEEKANNAG SAVAPNLNET QFTKLDELLT QTQLYSEFLL EKMEDITING IESESQKAEP EKTGRGRKRK AASQYNNTKA K RAVAAMIS RSKEDGETIN SDLTEEETVI KLQNELCPLL TGGQLKSYQL KGVKWLISLW QNGLNGILAD QMGLGKTIQT IG FLSHLKG NGLDGPYLVI APLSTLSNWF NEIARFTPSI NAIIYHGDKN QRDELRRKHM PKTVGPKFPI VITSYEVAMN DAK RILRHY PWKYVVIDEG HRLKNHKCKL LRELKHLKMD NKLLLTGTPL QNNLSELWSL LNFILPDIFT SHDEFESWFD FSEK NKNEA TKEEEEKRRA QVVSKLHGIL RPFILRRMKC DVELSLPRKK EIIMYATMTD HQKKFQEHLV NNTLEAHLGE NAIRG QGWK GKLNNLVIQL RKNCNHPDLL QGQIDGSYLY PPVEEIVGQC GKFRLLERLL VRLFANNHKV LIFSQWTKLL DIMDYY FSE KGFEVCRIDG SVKLDERRRQ IKDFSDEKSS CSIFLLSTRA GGLGINLTAA DTCILYDSDW NPQMDLQAMD RCHRIGQ TK PVHVYRLSTA QSIETRVLKR AYSKLKLEHV VIGQGQFHQE RAKSSTPLEE EDILALLKED ETAEDKLIQT DISDADLD R LLDRSDLTIT APGETQAAEA FPVKGPGWEV VLPSSGGMLS SLNS UniProtKB: ATP-dependent DNA helicase DDM1 |
-分子 #5: DNA (sense strand)
| 分子 | 名称: DNA (sense strand) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 51.310664 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC) ...文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT) (DA)(DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT) (DA)(DA) (DA)(DC)(DG)(DC)(DA)(DC)(DG) (DT)(DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT) (DC)(DC)(DC) (DC)(DC)(DG)(DC)(DG)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DC) (DA)(DA)(DG)(DG) (DG)(DG)(DA)(DT)(DT) (DA)(DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT) (DC)(DT)(DC)(DC)(DA) (DG)(DG)(DC)(DA) (DC)(DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT) (DA)(DT)(DA)(DT)(DA)(DC) (DA)(DT)(DC) (DC)(DG)(DA)(DT)(DT)(DC)(DC)(DA)(DG)(DT) (DG)(DC)(DC)(DG)(DG)(DT)(DG) (DT)(DC) (DG)(DC)(DT)(DG)(DA) |
-分子 #6: DNA (antisense strand)
| 分子 | 名称: DNA (antisense strand) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 51.799977 KDa |
| 配列 | 文字列: (DT)(DC)(DA)(DG)(DC)(DG)(DA)(DC)(DA)(DC) (DC)(DG)(DG)(DC)(DA)(DC)(DT)(DG)(DG)(DA) (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT) (DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA) (DC) (DA)(DC)(DG)(DT)(DG) ...文字列: (DT)(DC)(DA)(DG)(DC)(DG)(DA)(DC)(DA)(DC) (DC)(DG)(DG)(DC)(DA)(DC)(DT)(DG)(DG)(DA) (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT) (DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA) (DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT) (DG)(DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG) (DG)(DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC) (DC)(DC)(DT)(DT)(DG)(DG)(DG)(DC)(DG)(DG) (DT)(DT)(DA) (DA)(DA)(DC)(DG)(DC)(DG) (DG)(DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG) (DC)(DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC) (DG)(DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG) (DT)(DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC) (DT)(DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC) (DC)(DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC) (DG)(DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA) (DC)(DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC) (DT)(DC)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-32 / 実像数: 2858 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-8wh5: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)