+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
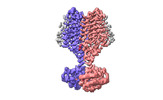
| タイトル | Cryo-EM structure of PAO1-ImcA with GMPCPP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GGDEF domain / diguanylate cyclase activity / membrane protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of bacterial-type flagellum-dependent cell motility / diguanylate cyclase / diguanylate cyclase activity / cell adhesion involved in single-species biofilm formation / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Zhan XL / Zhang K / Wang CC / Fan Q / Tang XJ / Zhang X / Wang K / Fu Y / Liang HH | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: A c-di-GMP signaling module controls responses to iron in Pseudomonas aeruginosa. 著者: Xueliang Zhan / Kuo Zhang / Chenchen Wang / Qiao Fan / Xiujia Tang / Xi Zhang / Ke Wang / Yang Fu / Haihua Liang /  要旨: Cyclic dimeric guanosine monophosphate (c-di-GMP) serves as a bacterial second messenger that modulates various processes including biofilm formation, motility, and host-microbe symbiosis. Numerous ...Cyclic dimeric guanosine monophosphate (c-di-GMP) serves as a bacterial second messenger that modulates various processes including biofilm formation, motility, and host-microbe symbiosis. Numerous studies have conducted comprehensive analysis of c-di-GMP. However, the mechanisms by which certain environmental signals such as iron control intracellular c-di-GMP levels are unclear. Here, we show that iron regulates c-di-GMP levels in Pseudomonas aeruginosa by modulating the interaction between an iron-sensing protein, IsmP, and a diguanylate cyclase, ImcA. Binding of iron to the CHASE4 domain of IsmP inhibits the IsmP-ImcA interaction, which leads to increased c-di-GMP synthesis by ImcA, thus promoting biofilm formation and reducing bacterial motility. Structural characterization of the apo-CHASE4 domain and its binding to iron allows us to pinpoint residues defining its specificity. In addition, the cryo-electron microscopy structure of ImcA in complex with a c-di-GMP analog (GMPCPP) suggests a unique conformation in which the compound binds to the catalytic pockets and to the membrane-proximal side located at the cytoplasm. Thus, our results indicate that a CHASE4 domain directly senses iron and modulates the crosstalk between c-di-GMP metabolic enzymes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37444.map.gz emd_37444.map.gz | 165.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37444-v30.xml emd-37444-v30.xml emd-37444.xml emd-37444.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_37444.png emd_37444.png | 53.2 KB | ||
| Filedesc metadata |  emd-37444.cif.gz emd-37444.cif.gz | 5.9 KB | ||
| その他 |  emd_37444_half_map_1.map.gz emd_37444_half_map_1.map.gz emd_37444_half_map_2.map.gz emd_37444_half_map_2.map.gz | 164.9 MB 164.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37444 http://ftp.pdbj.org/pub/emdb/structures/EMD-37444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37444 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37444_validation.pdf.gz emd_37444_validation.pdf.gz | 756.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37444_full_validation.pdf.gz emd_37444_full_validation.pdf.gz | 756.3 KB | 表示 | |
| XML形式データ |  emd_37444_validation.xml.gz emd_37444_validation.xml.gz | 15 KB | 表示 | |
| CIF形式データ |  emd_37444_validation.cif.gz emd_37444_validation.cif.gz | 17.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37444 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37444 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37444 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37444 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8wcnMC  8wctC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37444.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37444.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.668 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_37444_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37444_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GGDEF domain-containing protein
| 全体 | 名称: GGDEF domain-containing protein |
|---|---|
| 要素 |
|
-超分子 #1: GGDEF domain-containing protein
| 超分子 | 名称: GGDEF domain-containing protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Diguanylate cyclase
| 分子 | 名称: Diguanylate cyclase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 51.329805 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSDYDIPTT ENLYFQGSML ARDSLVQAGL PDNPYARQLR NGFRWLRFEK ELENEFREFL SWNSLMQRRA AIGVAFLIWA LFIVADWMM VDIRLHPSLF EQLLGVRLGM IGLLLVVWPA AFLPSLRKVG DAIAPYCLLL INLAVLACDV LFEWHGVPRF T QLGATLGI ...文字列: MGSDYDIPTT ENLYFQGSML ARDSLVQAGL PDNPYARQLR NGFRWLRFEK ELENEFREFL SWNSLMQRRA AIGVAFLIWA LFIVADWMM VDIRLHPSLF EQLLGVRLGM IGLLLVVWPA AFLPSLRKVG DAIAPYCLLL INLAVLACDV LFEWHGVPRF T QLGATLGI LAVFFPLGLA FWACVRLALL CLALNLAVFL LFGGEENLRT NLLNTLYNGL VVLICSFALY LQDYAQREQF LG RRLLGMM AEQDSLTGLV NRRYYELLAQ RALEQGAREE KGVALILVDV DDFKAYNDHY GHPAGDAALR QLGVVLRQGA RRP LDIAAR LGGEEFAVLL YDSEEGNTLA IAERLRQAVE ALGIEHLGSS AGPCLTISLG VAYSTSGMGL DALYREADRA LYEA KDAGR NAVRVAFRQH DRLEGSFLSA WSHPQFEKGG GSGGGSGGGS WSHPQFEKLE HHHHHH UniProtKB: diguanylate cyclase |
-分子 #2: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER
| 分子 | 名称: PHOSPHOMETHYLPHOSPHONIC ACID GUANYLATE ESTER / タイプ: ligand / ID: 2 / コピー数: 4 / 式: G2P |
|---|---|
| 分子量 | 理論値: 521.208 Da |
| Chemical component information | 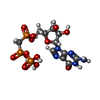 ChemComp-G2P: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)