[日本語] English
 万見
万見- EMDB-37423: CryoEM structure of non-structural protein 1 tetramer from ZIKA virus -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of non-structural protein 1 tetramer from ZIKA virus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | flavivirus / non-structural protein 1 / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribonucleoside triphosphate phosphatase activity / viral capsid / double-stranded RNA binding / methyltransferase cap1 activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / RNA helicase activity / protein dimerization activity / host cell endoplasmic reticulum membrane / symbiont-mediated suppression of host innate immune response / symbiont entry into host cell ...ribonucleoside triphosphate phosphatase activity / viral capsid / double-stranded RNA binding / methyltransferase cap1 activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / RNA helicase activity / protein dimerization activity / host cell endoplasmic reticulum membrane / symbiont-mediated suppression of host innate immune response / symbiont entry into host cell / serine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / fusion of virus membrane with host endosome membrane / virion attachment to host cell / host cell nucleus / virion membrane / structural molecule activity / proteolysis / extracellular region / ATP binding / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  dengue virus type 4 (デング熱ウイルス) dengue virus type 4 (デング熱ウイルス) | |||||||||
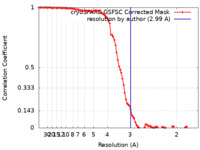
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.99 Å | |||||||||
 データ登録者 データ登録者 | Jiao HZ / Pan Q / Hu HL | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: The step-by-step assembly mechanism of secreted flavivirus NS1 tetramer and hexamer captured at atomic resolution. 著者: Qi Pan / Haizhan Jiao / Wanqin Zhang / Qiang Chen / Geshu Zhang / Jianhai Yu / Wei Zhao / Hongli Hu /  要旨: Flaviviruses encode a conserved, membrane-associated nonstructural protein 1 (NS1) with replication and immune evasion functions. The current knowledge of secreted NS1 (sNS1) oligomers is based on ...Flaviviruses encode a conserved, membrane-associated nonstructural protein 1 (NS1) with replication and immune evasion functions. The current knowledge of secreted NS1 (sNS1) oligomers is based on several low-resolution structures, thus hindering the development of drugs and vaccines against flaviviruses. Here, we revealed that recombinant sNS1 from flaviviruses exists in a dynamic equilibrium of dimer-tetramer-hexamer states. Two DENV4 hexameric NS1 structures and several tetrameric NS1 structures from multiple flaviviruses were solved at atomic resolution by cryo-EM. The stacking of the tetrameric NS1 and hexameric NS1 is facilitated by the hydrophobic β-roll and connector domains. Additionally, a triacylglycerol molecule located within the central cavity may play a role in stabilizing the hexamer. Based on differentiated interactions between the dimeric NS1, two distinct hexamer models (head-to-head and side-to-side hexamer) and the step-by-step assembly mechanisms of NS1 dimer into hexamer were proposed. We believe that our study sheds light on the understanding of the NS1 oligomerization and contributes to NS1-based therapies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37423.map.gz emd_37423.map.gz | 79 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37423-v30.xml emd-37423-v30.xml emd-37423.xml emd-37423.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37423_fsc.xml emd_37423_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37423.png emd_37423.png | 147.9 KB | ||
| Filedesc metadata |  emd-37423.cif.gz emd-37423.cif.gz | 5.4 KB | ||
| その他 |  emd_37423_additional_1.map.gz emd_37423_additional_1.map.gz emd_37423_half_map_1.map.gz emd_37423_half_map_1.map.gz emd_37423_half_map_2.map.gz emd_37423_half_map_2.map.gz | 42 MB 77.6 MB 77.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37423 http://ftp.pdbj.org/pub/emdb/structures/EMD-37423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37423 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37423 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8wbfMC  8wbbC  8wbcC  8wbdC  8wbeC  8wbgC  8wbhC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37423.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37423.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
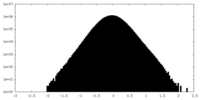
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_37423_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
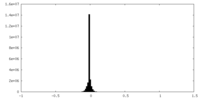
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37423_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
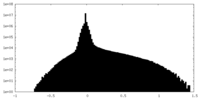
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37423_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
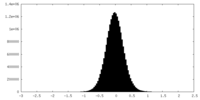
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ZIKA virus non-structural protein 1 tetramer
| 全体 | 名称: ZIKA virus non-structural protein 1 tetramer |
|---|---|
| 要素 |
|
-超分子 #1: ZIKA virus non-structural protein 1 tetramer
| 超分子 | 名称: ZIKA virus non-structural protein 1 tetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  dengue virus type 4 (デング熱ウイルス) dengue virus type 4 (デング熱ウイルス) |
-分子 #1: Genome polyprotein
| 分子 | 名称: Genome polyprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  dengue virus type 4 (デング熱ウイルス) dengue virus type 4 (デング熱ウイルス) |
| 分子量 | 理論値: 44.49816 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHVGCS VDFSKKETRC GTGVFVYNDV EAWRDRYKYH PDSPRRLAAA VKQAWEDGIC GISSVSRMEN IMWRSVEGEL NAILEENGV QLTVVVGSVK NPMWRGPQRL PVPVNELPHG WKAWGKSYFV RAAKTNNSFV VDGDTLKECP LKHRAWNSFL V EDHGFGVF ...文字列: HHHHHHVGCS VDFSKKETRC GTGVFVYNDV EAWRDRYKYH PDSPRRLAAA VKQAWEDGIC GISSVSRMEN IMWRSVEGEL NAILEENGV QLTVVVGSVK NPMWRGPQRL PVPVNELPHG WKAWGKSYFV RAAKTNNSFV VDGDTLKECP LKHRAWNSFL V EDHGFGVF HTSVWLKVRE DYSLECDPAV IGTAVKGKEA VHSDLGYWIE SEKNDTWRLK RAHLIEMKTC EWPKSHTLWT DG IEESDLI IPKSLAGPLS HHNTREGYRT QMKGPWHSEE LEIRFEECPG TKVHVEETCG TRGPSLRSTT ASGRVIEEWC CRE CTMPPL SFRAKDGCWY GMEIRPRKEP ESNLVRSMVT AGSLVPRGSW SHPQFEKGGG SGGGSGGSAW SHPQFEK UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)