+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the GPI inositol-deacylase (PGAP1/Bst1) from Chaetomium thermophilum | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bst1 / Glycosylphosphatidylinositol / GPI anchoring / GPI-AP / GPI-AP remodelase / Integral membrane enzyme / Lipase / Lipid remodeling / Membrane enzyme / Membrane protein / Nanodisc / PGAP1 / Transmembrane enzyme / Triad enzyme | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GPI anchor metabolic process / phosphatidylinositol deacylase activity / endoplasmic reticulum to Golgi vesicle-mediated transport / protein transport / 加水分解酵素; エステル加水分解酵素 / endoplasmic reticulum membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Thermochaetoides thermophila (菌類) / synthetic construct (人工物) Thermochaetoides thermophila (菌類) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.84 Å | |||||||||
 データ登録者 データ登録者 | Hong J / Li T / Qu Q / Li D | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Molecular basis of the inositol deacylase PGAP1 involved in quality control of GPI-AP biogenesis. 著者: Jingjing Hong / Tingting Li / Yulin Chao / Yidan Xu / Zhini Zhu / Zixuan Zhou / Weijie Gu / Qianhui Qu / Dianfan Li /  要旨: The secretion and quality control of glycosylphosphatidylinositol-anchored proteins (GPI-APs) necessitates post-attachment remodeling initiated by the evolutionarily conserved PGAP1, which deacylates ...The secretion and quality control of glycosylphosphatidylinositol-anchored proteins (GPI-APs) necessitates post-attachment remodeling initiated by the evolutionarily conserved PGAP1, which deacylates the inositol in nascent GPI-APs. Impairment of PGAP1 activity leads to developmental diseases in humans and fatality and infertility in animals. Here, we present three PGAP1 structures (2.66-2.84 Å), revealing its 10-transmembrane architecture and product-enzyme interaction details. PGAP1 holds GPI-AP acyl chains in an optimally organized, guitar-shaped cavity with apparent energetic penalties from hydrophobic-hydrophilic mismatches. However, abundant glycan-mediated interactions in the lumen counterbalance these repulsions, likely conferring substrate fidelity and preventing off-target hydrolysis of bulk membrane lipids. Structural and biochemical analyses uncover a serine hydrolase-type catalysis with atypical features and imply mechanisms for substrate entrance and product release involving a drawing compass movement of GPI-APs. Our findings advance the mechanistic understanding of GPI-AP remodeling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36995.map.gz emd_36995.map.gz | 78.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36995-v30.xml emd-36995-v30.xml emd-36995.xml emd-36995.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36995.png emd_36995.png | 50.9 KB | ||
| Filedesc metadata |  emd-36995.cif.gz emd-36995.cif.gz | 7.1 KB | ||
| その他 |  emd_36995_half_map_1.map.gz emd_36995_half_map_1.map.gz emd_36995_half_map_2.map.gz emd_36995_half_map_2.map.gz | 77.7 MB 77.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36995 http://ftp.pdbj.org/pub/emdb/structures/EMD-36995 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36995 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36995 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36995_validation.pdf.gz emd_36995_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36995_full_validation.pdf.gz emd_36995_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_36995_validation.xml.gz emd_36995_validation.xml.gz | 13 KB | 表示 | |
| CIF形式データ |  emd_36995_validation.cif.gz emd_36995_validation.cif.gz | 15.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36995 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36995 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36995 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36995 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36995.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36995.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36995_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36995_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GPI inositol-deacylase
| 全体 | 名称: GPI inositol-deacylase |
|---|---|
| 要素 |
|
-超分子 #1: GPI inositol-deacylase
| 超分子 | 名称: GPI inositol-deacylase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
| 分子量 | 理論値: 163 KDa |
-分子 #1: GPI inositol-deacylase,fused thermostable green fluorescent protein
| 分子 | 名称: GPI inositol-deacylase,fused thermostable green fluorescent protein タイプ: protein_or_peptide / ID: 1 詳細: The protein is a fusion protein with expression tag. Residues 1187-1196 is the linker with a 3 C protease digestion site. Residues 1197-1427 is a fused thermostable green fluorescent protein ...詳細: The protein is a fusion protein with expression tag. Residues 1187-1196 is the linker with a 3 C protease digestion site. Residues 1197-1427 is a fused thermostable green fluorescent protein (PDB entry 4TZA, residue 5-229). Residues 1428-1469 is the linker a Strep II tag and a His tag. コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 163.153094 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGSRSLSSAS SDDDDAPPIR VPRVNQCATS RTKDSQSPAQ SASKLDRRRS ADRRPSFSAN RRSGTGAGTG TGTGIANWRP FDSRDATVE RAGSSTATTA TTPPPSSSLG LMLAANGAVQ EKEMVMMGKA QEHGFVGRRA PWRSPWAISV FAFVTSLLGI G LLLAVIHS ...文字列: MGSRSLSSAS SDDDDAPPIR VPRVNQCATS RTKDSQSPAQ SASKLDRRRS ADRRPSFSAN RRSGTGAGTG TGTGIANWRP FDSRDATVE RAGSSTATTA TTPPPSSSLG LMLAANGAVQ EKEMVMMGKA QEHGFVGRRA PWRSPWAISV FAFVTSLLGI G LLLAVIHS SVTRQIDPKG CRMSYMRPSY AKLSDFDTEH TRLASKYSLY LYREQGIDHD VKVRGVPVLF IPGNAGSYKQ VR PIAAEAA NYFHDVLQHD EAALRAGVRS LDFFTVDFNE DITAFHGQTL LDQAEYLNEA IRYILSLYLD PRVSERDPDL PDP TSVIVL GHSMGGIVAR TMLIMPNYQH NSINTIITMS APHARPPVSF DGQIVQTYKD INNYWRHAYS QKWANDNPLW HVTL VSIAG GGLDTVVPSD YASIESLVPD THGFTVFTST IPNVWTSMDH QAILWCDQFR KVIIRALFDI VDVHRASQTK PRAQR MRVF KKWFLSGMET VAEKIAPTSD PTTLLIVDDK SDSITAEGER LVLRELGTQG SVRAHLMPIP PPGSPELKRF TLLTDT KLD KPGENGKLEV MFCSVIPSQP NPTGPAIPSQ LDLSKGNAGT TRLACTNVAP DVITLPASTR FARFPFSVRK EAEIPPF SY LEYVLDDISE HQFVAVIEKA TIPTPGFVIA EFSDHSNSHH TRHIGLRNLL TFGISLRLPS NRPMMSEVRI PSVKSSLL A YNLRISALEC SGRKDLFAPL VRQYLAEPYE SKYFVNARQA AVSLHGVAPY VPPPMSREPE AEGLAFQLWT DPTCNSSIQ VDLTVDVMGS LGKLYMRYRT VFAAFPLFIV SLVLRKQFQV YDSTGSFITF AEGLDLSLRQ SIPVMLIVLA ALTLSTTKMA PSSSAGLWH WGGNTTFTNF HQNDLLIGTQ DPFFLFLIPL IGIICVGVCT VVNYIALSLT RLISVVISFI GFLTVRFGWV N AEDRRRPS NPAIFPPSSP RRRMITTAVL LFLVSTMIPY QLAYLVACLV QLGTLVRAQR ISSELRSPAN SNFHNYVHSI FI LMLWILP INLPTLVVWM HNLSVHWLTP FTSHHNVFSI MPFILLVETH TTGQMIPRTG GTGNGRCCVL LRHITSILLL SLA LYAAVY GVSYAYTLHQ FVNLFAFWLV MVHSTADDWS LTGLRQLILH NRNNANNKSE TGSRKRGKEP GTLEVLFQGP GGSG GSASV IKPEMKIKLR MEGAVNGHKF VIEGEGIGKP YEGTQTLDLT VEEGAPLPFS YDILTPAFQY GNRAFTKYPE DIPDY FKQA FPEGYSWERS MTYEDQGICI ATSDITMEGD CFFYEIRFDG TNFPPNGPVM QKKTLKWEPS TEKMYVEDGV LKGDVE MAL LLEGGGHYRC DFKTTYKAKK DVRLPDAHEV DHRIEILSHD KDYNKVRLYE HAEARYSGGG SGGGSAWSHP QFEKGGG SG GGSGGSAWSH PQFEKGSHHH HHHHHHH UniProtKB: GPI inositol-deacylase |
-分子 #2: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 2 / コピー数: 3 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #3: (2~{S})-2-azanyl-3-[[(2~{R})-3-hexadecanoyloxy-2-[(~{Z})-octadec-...
| 分子 | 名称: (2~{S})-2-azanyl-3-[[(2~{R})-3-hexadecanoyloxy-2-[(~{Z})-octadec-9-enoyl]oxy-propoxy]-oxidanyl-phosphoryl]oxy-propanoic acid タイプ: ligand / ID: 3 / コピー数: 2 / 式: D39 |
|---|---|
| 分子量 | 理論値: 762.006 Da |
| Chemical component information | 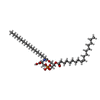 ChemComp-D39: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 7.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 50 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.026000000000000002 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 4.5s before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.84 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. v3.3.2) / 使用した粒子像数: 407921 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: cryoSPARC (ver. v3.3.2) |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: cryoSPARC (ver. v3.3.2) |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




































