+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | G1 nucleosome gyre proximal | |||||||||
 マップデータ マップデータ | G1 nucleosome gyre proximal class 1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cell / RPE-1 / nucleus / nucleosome / NUCLEAR PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 30.6 Å | |||||||||
 データ登録者 データ登録者 | Chen JK / Cai S / Liu T / Ruan W / Ng CT / Shi J / Surana U / Gan L | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2025 ジャーナル: EMBO J / 年: 2025タイトル: Nanoscale analysis of human G1 and metaphase chromatin in situ. 著者: Jon Ken Chen / Tingsheng Liu / Shujun Cai / Weimei Ruan / Cai Tong Ng / Jian Shi / Uttam Surana / Lu Gan /   要旨: The structure of chromatin at the nucleosome level inside cells is still incompletely understood. Here we present in situ electron cryotomography analyses of chromatin in both G1 and metaphase RPE-1 ...The structure of chromatin at the nucleosome level inside cells is still incompletely understood. Here we present in situ electron cryotomography analyses of chromatin in both G1 and metaphase RPE-1 cells. G1 nucleosomes are concentrated in globular chromatin domains, and metaphase nucleosomes are concentrated in the chromatids. Classification analysis reveals that canonical mononucleosomes, and in some conditions ordered stacked dinucleosomes and mononucleosomes with a disordered gyre-proximal density, are abundant in both cell-cycle states. We do not detect class averages that have more than two stacked nucleosomes or side-by-side dinucleosomes, suggesting that groups of more than two nucleosomes are heterogeneous. Large multi-megadalton structures are abundant in G1 nucleoplasm, but not found in G1 chromatin domains and metaphase chromatin. The macromolecular phenotypes studied here represent a starting point for the comparative analysis of compaction in normal vs. unhealthy human cells, in other cell-cycle states, other organisms, and in vitro chromatin assemblies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36994.map.gz emd_36994.map.gz | 741.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36994-v30.xml emd-36994-v30.xml emd-36994.xml emd-36994.xml | 24.1 KB 24.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36994.png emd_36994.png | 49.8 KB | ||
| Filedesc metadata |  emd-36994.cif.gz emd-36994.cif.gz | 4.7 KB | ||
| その他 |  emd_36994_additional_1.map.gz emd_36994_additional_1.map.gz emd_36994_additional_2.map.gz emd_36994_additional_2.map.gz emd_36994_additional_3.map.gz emd_36994_additional_3.map.gz emd_36994_additional_4.map.gz emd_36994_additional_4.map.gz emd_36994_half_map_1.map.gz emd_36994_half_map_1.map.gz emd_36994_half_map_2.map.gz emd_36994_half_map_2.map.gz | 742.8 KB 741.8 KB 742.4 KB 742 KB 740.7 KB 740 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36994 http://ftp.pdbj.org/pub/emdb/structures/EMD-36994 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36994 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36994 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36994.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36994.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | G1 nucleosome gyre proximal class 1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.766 Å | ||||||||||||||||||||||||||||||||||||
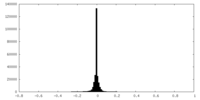
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: G1 nucleosome gyre proximal class 2
| ファイル | emd_36994_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | G1 nucleosome gyre proximal class 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
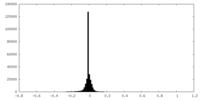
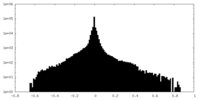
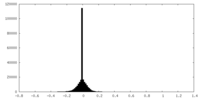
| 密度ヒストグラム |
-追加マップ: G1 nucleosome gyre proximal class 3
| ファイル | emd_36994_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | G1 nucleosome gyre proximal class 3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
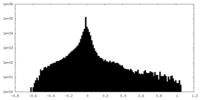
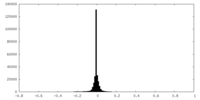
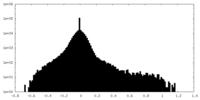
| 密度ヒストグラム |
-追加マップ: G1 nucleosome gyre proximal class 4
| ファイル | emd_36994_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | G1 nucleosome gyre proximal class 4 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
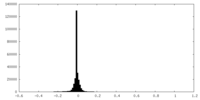
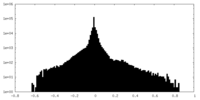
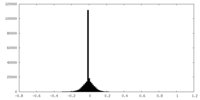
| 密度ヒストグラム |
-追加マップ: G1 nucleosome gyre proximal class 5
| ファイル | emd_36994_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | G1 nucleosome gyre proximal class 5 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
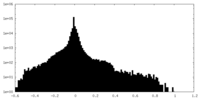
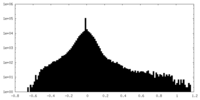
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_36994_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_36994_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome gyre proximal from RPE-1
| 全体 | 名称: Nucleosome gyre proximal from RPE-1 |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome gyre proximal from RPE-1
| 超分子 | 名称: Nucleosome gyre proximal from RPE-1 / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Nucleus Homo sapiens (ヒト) / Organelle: Nucleus |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: C-flat / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 詳細: C-flat 2/4 |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 30369 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.1 µm / 最小 デフォーカス(公称値): 0.1 µm / 倍率(公称値): 18000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)