+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the RC-LH core comples from Halorhodospira halochloris | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RC-LH complex / HALORHODOSPIRA HALOCHLORIS / PHOTOSYNTHESIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / : / endomembrane system / electron transfer activity / iron ion binding ...organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / : / endomembrane system / electron transfer activity / iron ion binding / heme binding / metal ion binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Halorhodospira halochloris (紅色硫黄細菌) Halorhodospira halochloris (紅色硫黄細菌) | |||||||||
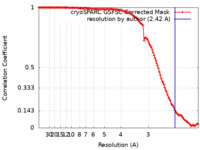
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.42 Å | |||||||||
 データ登録者 データ登録者 | Wang G-L / Qi C-H / Yu L-J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Integr Plant Biol / 年: 2024 ジャーナル: J Integr Plant Biol / 年: 2024タイトル: Structural insights into the unusual core photocomplex from a triply extremophilic purple bacterium, Halorhodospira halochloris. 著者: Chen-Hui Qi / Guang-Lei Wang / Fang-Fang Wang / Jie Wang / Xiang-Ping Wang / Mei-Juan Zou / Fei Ma / Michael T Madigan / Yukihiro Kimura / Zheng-Yu Wang-Otomo / Long-Jiang Yu /    要旨: Halorhodospira (Hlr.) halochloris is a triply extremophilic phototrophic purple sulfur bacterium, as it is thermophilic, alkaliphilic, and extremely halophilic. The light-harvesting-reaction center ...Halorhodospira (Hlr.) halochloris is a triply extremophilic phototrophic purple sulfur bacterium, as it is thermophilic, alkaliphilic, and extremely halophilic. The light-harvesting-reaction center (LH1-RC) core complex of this bacterium displays an LH1-Q transition at 1,016 nm, which is the lowest-energy wavelength absorption among all known phototrophs. Here we report the cryo-EM structure of the LH1-RC at 2.42 Å resolution. The LH1 complex forms a tricyclic ring structure composed of 16 αβγ-polypeptides and one αβ-heterodimer around the RC. From the cryo-EM density map, two previously unrecognized integral membrane proteins, referred to as protein G and protein Q, were identified. Both of these proteins are single transmembrane-spanning helices located between the LH1 ring and the RC L-subunit and are absent from the LH1-RC complexes of all other purple bacteria of which the structures have been determined so far. Besides bacteriochlorophyll b molecules (B1020) located on the periplasmic side of the Hlr. halochloris membrane, there are also two arrays of bacteriochlorophyll b molecules (B800 and B820) located on the cytoplasmic side. Only a single copy of a carotenoid (lycopene) was resolved in the Hlr. halochloris LH1-α3β3 and this was positioned within the complex. The potential quinone channel should be the space between the LH1-α3β3 that accommodates the single lycopene but does not contain a γ-polypeptide, B800 and B820. Our results provide a structural explanation for the unusual Q red shift and carotenoid absorption in the Hlr. halochloris spectrum and reveal new insights into photosynthetic mechanisms employed by a species that thrives under the harshest conditions of any phototrophic microorganism known. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36907.map.gz emd_36907.map.gz | 167.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36907-v30.xml emd-36907-v30.xml emd-36907.xml emd-36907.xml | 27.7 KB 27.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36907_fsc.xml emd_36907_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36907.png emd_36907.png | 53.1 KB | ||
| Filedesc metadata |  emd-36907.cif.gz emd-36907.cif.gz | 7.7 KB | ||
| その他 |  emd_36907_half_map_1.map.gz emd_36907_half_map_1.map.gz emd_36907_half_map_2.map.gz emd_36907_half_map_2.map.gz | 165.5 MB 165.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36907 http://ftp.pdbj.org/pub/emdb/structures/EMD-36907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36907 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36907_validation.pdf.gz emd_36907_validation.pdf.gz | 1016.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36907_full_validation.pdf.gz emd_36907_full_validation.pdf.gz | 1015.8 KB | 表示 | |
| XML形式データ |  emd_36907_validation.xml.gz emd_36907_validation.xml.gz | 20.4 KB | 表示 | |
| CIF形式データ |  emd_36907_validation.cif.gz emd_36907_validation.cif.gz | 26.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36907 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36907 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36907 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36907 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8k5oMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36907.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36907.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.02 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36907_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36907_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : RC-LH core complex
+超分子 #1: RC-LH core complex
+分子 #1: Photosynthetic reaction center cytochrome c subunit
+分子 #2: Reaction center protein L chain
+分子 #3: Reaction center protein M chain
+分子 #4: Photosynthetic reaction center H subunit
+分子 #5: Antenna complex alpha/beta subunit domain-containing protein
+分子 #6: Light-harvesting LHI
+分子 #7: Antenna complex alpha/beta subunit domain-containing protein
+分子 #8: Beta subunit of light-harvesting 1
+分子 #9: Gamma subunit of light-harvesting 1
+分子 #10: reaction center small polypeptide
+分子 #11: reactin center small polypeptide
+分子 #12: HEME C
+分子 #13: (1R)-2-{[{[(2S)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
+分子 #14: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #15: Trans-Geranyl Bacteriochlorophyll B
+分子 #16: Trans-Geranyl Bacteriopheophytin B
+分子 #17: Ubiquinone-8
+分子 #18: UNKNOWN LIGAND
+分子 #19: FE (III) ION
+分子 #20: MENAQUINONE 8
+分子 #21: CARDIOLIPIN
+分子 #22: LYCOPENE
+分子 #23: Trans-Geranyl 8-vinyl-bacteriochlorophyll B
+分子 #24: DI-PALMITOYL-3-SN-PHOSPHATIDYLETHANOLAMINE
+分子 #25: 2-O-octyl-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) 平均電子線量: 60.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)