+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of AE2 in complex with PIP2 | |||||||||
 マップデータ マップデータ | sharpened map of AE2 in complex with PIP2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AE2 / SLC4A2 / PIP2 / membrane protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of CD8-positive, alpha-beta T cell differentiation / negative regulation of CD8-positive, alpha-beta T cell proliferation / positive regulation of enamel mineralization / Bicarbonate transporters / amelogenesis / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity / bicarbonate transport / monoatomic anion transport ...negative regulation of CD8-positive, alpha-beta T cell differentiation / negative regulation of CD8-positive, alpha-beta T cell proliferation / positive regulation of enamel mineralization / Bicarbonate transporters / amelogenesis / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity / bicarbonate transport / monoatomic anion transport / digestive tract development / regulation of bone resorption / osteoclast differentiation / regulation of intracellular pH / regulation of actin cytoskeleton organization / transmembrane transport / spermatogenesis / basolateral plasma membrane / apical plasma membrane / focal adhesion / enzyme binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Yin YX / Ding D | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural and functional insights into the lipid regulation of human anion exchanger 2. 著者: Weiqi Zhang / Dian Ding / Yishuo Lu / Hongyi Chen / Peijun Jiang / Peng Zuo / Guangxi Wang / Juan Luo / Yue Yin / Jianyuan Luo / Yuxin Yin /  要旨: Anion exchanger 2 (AE2) is an electroneutral Na-independent Cl/HCO exchanger belongs to the SLC4 transporter family. The widely expressed AE2 participates in a variety of physiological processes, ...Anion exchanger 2 (AE2) is an electroneutral Na-independent Cl/HCO exchanger belongs to the SLC4 transporter family. The widely expressed AE2 participates in a variety of physiological processes, including transepithelial acid-base secretion and osteoclastogenesis. Both the transmembrane domains (TMDs) and the N-terminal cytoplasmic domain (NTD) are involved in regulation of AE2 activity. However, the regulatory mechanism remains unclear. Here, we report a 3.2 Å cryo-EM structure of the AE2 TMDs in complex with PIP and a 3.3 Å full-length mutant AE2 structure in the resting state without PIP. We demonstrate that PIP at the TMD dimer interface is involved in the substrate exchange process. Mutation in the PIP binding site leads to the displacement of TM7 and further stabilizes the interaction between the TMD and the NTD. Reduced substrate transport activity and conformation similar to AE2 in acidic pH indicating the central contribution of PIP to the function of AE2. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36448.map.gz emd_36448.map.gz | 230.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36448-v30.xml emd-36448-v30.xml emd-36448.xml emd-36448.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36448.png emd_36448.png | 91.5 KB | ||
| Filedesc metadata |  emd-36448.cif.gz emd-36448.cif.gz | 6.3 KB | ||
| その他 |  emd_36448_half_map_1.map.gz emd_36448_half_map_1.map.gz emd_36448_half_map_2.map.gz emd_36448_half_map_2.map.gz | 226 MB 226 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36448 http://ftp.pdbj.org/pub/emdb/structures/EMD-36448 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36448 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36448 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36448_validation.pdf.gz emd_36448_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36448_full_validation.pdf.gz emd_36448_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_36448_validation.xml.gz emd_36448_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_36448_validation.cif.gz emd_36448_validation.cif.gz | 18.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36448 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36448 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36448 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36448 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jniMC  8jnjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36448.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36448.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map of AE2 in complex with PIP2 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.821 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half A map of AE2 in complex with PIP2
| ファイル | emd_36448_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
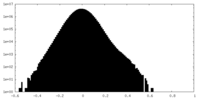
| 注釈 | half_A map of AE2 in complex with PIP2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half B map of AE2 in complex with PIP2
| ファイル | emd_36448_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
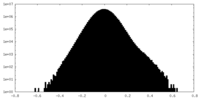
| 注釈 | half_B map of AE2 in complex with PIP2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Anion exchange protein 2, isoform A
| 全体 | 名称: Anion exchange protein 2, isoform A |
|---|---|
| 要素 |
|
-超分子 #1: Anion exchange protein 2, isoform A
| 超分子 | 名称: Anion exchange protein 2, isoform A / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Anion exchange protein 2
| 分子 | 名称: Anion exchange protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 137.176906 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSSAPRRPAK GADSFCTPEP ESLGPGTPGF PEQEEDELHR TLGVERFEEI LQEAGSRGGE EPGRSYGEED FEYHRQSSHH IHHPLSTHL PPDARRRKTP QGPGRKPRRR PGASPTGETP TIEEGEEDED EASEAEGARA LTQPSPVSTP SSVQFFLQED D SADRKAER ...文字列: MSSAPRRPAK GADSFCTPEP ESLGPGTPGF PEQEEDELHR TLGVERFEEI LQEAGSRGGE EPGRSYGEED FEYHRQSSHH IHHPLSTHL PPDARRRKTP QGPGRKPRRR PGASPTGETP TIEEGEEDED EASEAEGARA LTQPSPVSTP SSVQFFLQED D SADRKAER TSPSSPAPLP HQEATPRASK GAQAGTQVEE AEAEAVAVAS GTAGGDDGGA SGRPLPKAQP GHRSYNLQER RR IGSMTGA EQALLPRVPT DEIEAQTLAT ADLDLMKSHR FEDVPGVRRH LVRKNAKGST QSGREGREPG PTPRARPRAP HKP HEVFVE LNELLLDKNQ EPQWRETARW IKFEEDVEEE TERWGKPHVA SLSFRSLLEL RRTLAHGAVL LDLDQQTLPG VAHQ VVEQM VISDQIKAED RANVLRALLL KHSHPSDEKD FSFPRNISAG SLGSLLGHHH GQGAESDPHV TEPLMGGVPE TRLEV ERER ELPPPAPPAG ITRSKSKHEL KLLEKIPENA EATVVLVGCV EFLSRPTMAF VRLREAVELD AVLEVPVPVR FLFLLL GPS SANMDYHEIG RSISTLMSDK QFHEAAYLAD EREDLLTAIN AFLDCSVVLP PSEVQGEELL RSVAHFQRQM LKKREEQ GR LLPTGAGLEP KSAQDKALLQ MVEAAGAAED DPLRRTGRPF GGLIRDVRRR YPHYLSDFRD ALDPQCLAAV IFIYFAAL S PAITFGGLLG EKTQDLIGVS ELIMSTALQG VVFCLLGAQP LLVIGFSGPL LVFEEAFFSF CSSNHLEYLV GRVWIGFWL VFLALLMVAL EGSFLVRFVS RFTQEIFAFL ISLIFIYETF YKLVKIFQEH PLHGCSASNS SEVDGGENMT WAGARPTLGP GNRSLAGQS GQGKPRGQPN TALLSLVLMA GTFFIAFFLR KFKNSRFFPG RIRRVIGDFG VPIAILIMVL VDYSIEDTYT Q KLSVPSGF SVTAPEKRGW VINPLGEKSP FPVWMMVASL LPAILVFILI FMETQITTLI ISKKERMLQK GSGFHLDLLL IV AMGGICA LFGLPWLAAA TVRSVTHANA LTVMSKAVAP GDKPKIQEVK EQRVTGLLVA LLVGLSIVIG DLLRQIPLAV LFG IFLYMG VTSLNGIQFY ERLHLLLMPP KHHPDVTYVK KVRTLRMHLF TALQLLCLAL LWAVMSTAAS LAFPFILILT VPLR MVVLT RIFTDREMKC LDANEAEPVF DEREGVDEYN EMPMPV UniProtKB: Anion exchange protein 2 |
-分子 #2: CHLORIDE ION
| 分子 | 名称: CHLORIDE ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: CL |
|---|---|
| 分子量 | 理論値: 35.453 Da |
-分子 #3: [(2R)-1-octadecanoyloxy-3-[oxidanyl-[(1R,2R,3S,4R,5R,6S)-2,3,6-tr...
| 分子 | 名称: [(2R)-1-octadecanoyloxy-3-[oxidanyl-[(1R,2R,3S,4R,5R,6S)-2,3,6-tris(oxidanyl)-4,5-diphosphonooxy-cyclohexyl]oxy-phospho ryl]oxy-propan-2-yl] (8Z)-icosa-5,8,11,14-tetraenoate タイプ: ligand / ID: 3 / コピー数: 2 / 式: PT5 |
|---|---|
| 分子量 | 理論値: 1.047088 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.1.0) / 使用した粒子像数: 107451 |
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: OTHER |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)