+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3585 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the VirD4 bound to a Type IV secretion system | |||||||||
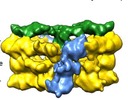
 マップデータ マップデータ | Negative stain electron microscopy of the T4SS3-10 D4 complex enableslocalization of the VirD4 coupling protein within the complex. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 28.0 Å | |||||||||
 データ登録者 データ登録者 | Redzej A / Ukleja M | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2017 ジャーナル: EMBO J / 年: 2017タイトル: Structure of a VirD4 coupling protein bound to a VirB type IV secretion machinery. 著者: Adam Redzej / Marta Ukleja / Sarah Connery / Martina Trokter / Catarina Felisberto-Rodrigues / Adam Cryar / Konstantinos Thalassinos / Richard D Hayward / Elena V Orlova / Gabriel Waksman /  要旨: Type IV secretion (T4S) systems are versatile bacterial secretion systems mediating transport of protein and/or DNA T4S systems are generally composed of 11 VirB proteins and 1 VirD protein (VirD4). ...Type IV secretion (T4S) systems are versatile bacterial secretion systems mediating transport of protein and/or DNA T4S systems are generally composed of 11 VirB proteins and 1 VirD protein (VirD4). The VirB1-11 proteins assemble to form a secretion machinery and a pilus while the VirD4 protein is responsible for substrate recruitment. The structure of VirD4 in isolation is known; however, its structure bound to the VirB1-11 apparatus has not been determined. Here, we purify a T4S system with VirD4 bound, define the biochemical requirements for complex formation and describe the protein-protein interaction network in which VirD4 is involved. We also solve the structure of this complex by negative stain electron microscopy, demonstrating that two copies of VirD4 dimers locate on both sides of the apparatus, in between the VirB4 ATPases. Given the central role of VirD4 in type IV secretion, our study provides mechanistic insights on a process that mediates the dangerous spread of antibiotic resistance genes among bacterial populations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3585.map.gz emd_3585.map.gz | 12.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3585-v30.xml emd-3585-v30.xml emd-3585.xml emd-3585.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3585.png emd_3585.png | 226.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3585 http://ftp.pdbj.org/pub/emdb/structures/EMD-3585 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3585 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3585 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3585_validation.pdf.gz emd_3585_validation.pdf.gz | 207.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3585_full_validation.pdf.gz emd_3585_full_validation.pdf.gz | 206.2 KB | 表示 | |
| XML形式データ |  emd_3585_validation.xml.gz emd_3585_validation.xml.gz | 6.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3585 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3585 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3585 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3585 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3585.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3585.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain electron microscopy of the T4SS3-10 D4 complex enableslocalization of the VirD4 coupling protein within the complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.54 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Type IV secretion system, inner membrane complex
| 全体 | 名称: Type IV secretion system, inner membrane complex |
|---|---|
| 要素 |
|
-超分子 #1: Type IV secretion system, inner membrane complex
| 超分子 | 名称: Type IV secretion system, inner membrane complex / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 1.5 MDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 50mM HEPES pH=7.6, 200mM sodium acetate, 0.1% digitonin, 0.05mM TDAO |
| 染色 | タイプ: NEGATIVE / 材質: 2 % uranyl acetate / 詳細: Staining for 3 min |
| グリッド | モデル: type B 400, Agar Scientific / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 4.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 検出モード: INTEGRATING / デジタル化 - 画像ごとのフレーム数: 2-19 / 平均露光時間: 2.5 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 41500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















