+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Full-Length AsfvPrimPol in Complex-Form | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | polymerase / primase / PrimPol / Helicase / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hydrolase activity, acting on acid anhydrides / helicase activity / DNA replication / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  African swine fever virus BA71V (ウイルス) / synthetic construct (人工物) African swine fever virus BA71V (ウイルス) / synthetic construct (人工物) | |||||||||
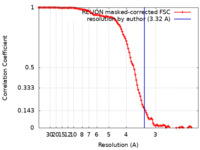
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.32 Å | |||||||||
 データ登録者 データ登録者 | Shao ZW / Su SC / Gan JH | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Structures and implications of the C962R protein of African swine fever virus. 著者: Zhiwei Shao / Shichen Su / Jie Yang / Weizhen Zhang / Yanqing Gao / Xin Zhao / Yixi Zhang / Qiyuan Shao / Chulei Cao / Huili Li / Hehua Liu / Jinru Zhang / Jinzhong Lin / Jinbiao Ma / Jianhua Gan /  要旨: African swine fever virus (ASFV) is highly contagious and can cause lethal disease in pigs. Although it has been extensively studied in the past, no vaccine or other useful treatment against ASFV is ...African swine fever virus (ASFV) is highly contagious and can cause lethal disease in pigs. Although it has been extensively studied in the past, no vaccine or other useful treatment against ASFV is available. The genome of ASFV encodes more than 170 proteins, but the structures and functions for the majority of the proteins remain elusive, which hindered our understanding on the life cycle of ASFV and the development of ASFV-specific inhibitors. Here, we report the structural and biochemical studies of the highly conserved C962R protein of ASFV, showing that C962R is a multidomain protein. The N-terminal AEP domain is responsible for the DNA polymerization activity, whereas the DNA unwinding activity is catalyzed by the central SF3 helicase domain. The middle PriCT2 and D5_N domains and the C-terminal Tail domain all contribute to the DNA unwinding activity of C962R. C962R preferentially works on forked DNA, and likely functions in Base-excision repair (BER) or other repair pathway in ASFV. Although it is not essential for the replication of ASFV, C962R can serve as a model and provide mechanistic insight into the replicative primase proteins from many other species, such as nitratiruptor phage NrS-1, vaccinia virus (VACV) and other viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35671.map.gz emd_35671.map.gz | 12.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35671-v30.xml emd-35671-v30.xml emd-35671.xml emd-35671.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35671_fsc.xml emd_35671_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35671.png emd_35671.png | 135 KB | ||
| Filedesc metadata |  emd-35671.cif.gz emd-35671.cif.gz | 6.4 KB | ||
| その他 |  emd_35671_half_map_1.map.gz emd_35671_half_map_1.map.gz emd_35671_half_map_2.map.gz emd_35671_half_map_2.map.gz | 98.2 MB 98.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35671 http://ftp.pdbj.org/pub/emdb/structures/EMD-35671 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35671 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35671 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35671_validation.pdf.gz emd_35671_validation.pdf.gz | 721.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35671_full_validation.pdf.gz emd_35671_full_validation.pdf.gz | 721.4 KB | 表示 | |
| XML形式データ |  emd_35671_validation.xml.gz emd_35671_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_35671_validation.cif.gz emd_35671_validation.cif.gz | 24.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35671 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35671 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35671 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35671 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35671.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35671.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35671_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
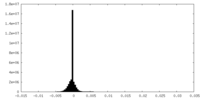
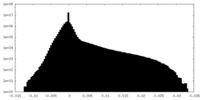
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35671_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
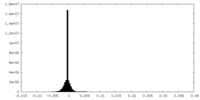
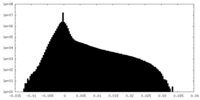
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Full-Length AsfvPrimPol in complex with DNA and AMPPNP
| 全体 | 名称: Full-Length AsfvPrimPol in complex with DNA and AMPPNP |
|---|---|
| 要素 |
|
-超分子 #1: Full-Length AsfvPrimPol in complex with DNA and AMPPNP
| 超分子 | 名称: Full-Length AsfvPrimPol in complex with DNA and AMPPNP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / 詳細: ASFV ORF C962R |
|---|---|
| 分子量 | 理論値: 660 KDa |
-超分子 #2: Full-Length AsfvPrimPol
| 超分子 | 名称: Full-Length AsfvPrimPol / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  African swine fever virus BA71V (ウイルス) African swine fever virus BA71V (ウイルス) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Putative primase C962R
| 分子 | 名称: Putative primase C962R / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  African swine fever virus BA71V (ウイルス) African swine fever virus BA71V (ウイルス) |
| 分子量 | 理論値: 111.706273 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMREESWEE HDTIQLTAQR KYLAEVQALE TLLARELSVF LTEPGSKKTN IINRITGKTY ALPSTELLRF YEHLEQCRKQ GALMYFLER QGTYSGLMLD YDLKLNTNAA PSLESSVLSR LCHRIFVHIK NSSVLPEGSH KIHFFFTLKP EAVQGKYGFH V LIPGLKMA ...文字列: GSMREESWEE HDTIQLTAQR KYLAEVQALE TLLARELSVF LTEPGSKKTN IINRITGKTY ALPSTELLRF YEHLEQCRKQ GALMYFLER QGTYSGLMLD YDLKLNTNAA PSLESSVLSR LCHRIFVHIK NSSVLPEGSH KIHFFFTLKP EAVQGKYGFH V LIPGLKMA ASTKKSIIAS LQHDATVQKI LHEQGVANPE SCLDPHSASV PSLLYGSSKL NHRPYQLKTG FELVFDSSDP DY IPIHQIK NIESYNLVSE LSLTNEQGSL VRPVYCAADI AAEKEEEIPA DDHSLSILML HDPEARYLHK ILNLLPPEYY VEY PLWSNV VFALANTSAN YRPLAEWFSQ KCPEKWNTGG KEKLEKLWND ASRHTEKKIT KRSIMYWAHK HAPQQYKEIV EQGY FSILA EYVYSYNGTL EHYMIAKVIY AMMGNKFVVD VDSNGKYVWF EFVLPGQPMN QGEIWKWRKE VNPDELHIYI SENFS RVMD RITEHIKYHL SQPHETNILN YYKKLLKAFE RSKSKIFNDS FKKGVIRQAE FLFRQRSFIQ TLDTNPYLLG VGNGVL SIE TIPAKLINHF HEHPIHQYTH ICYEPFNPEN PWTKLLLNAL QDIIPELDAR LWIMFYLSTA IFRGLKEALM LLWLGGG CN GKTFLMRLVA MVLGDHYASK LNISLLTSYR ETAEKPNSAF MRLKGRGYGY FEETNKSEIL NTSRLKEMVN PGDVTARE L NQKQESFQMT ATMVAASNYN FIIDTTDHGT WRRLRHYRSK VKFCHNPDPN NSYEKKEDPR FIHEYIMDPN CQNAFFSIL VYFWEKLQKE YNGQIKKVFC PTIESETEAY RKSQDTLHRF ITERVVESPS AETVYNLSEV VTAYAEWYNA NINVKRHIAL ELSQELENS VLEKYLQWSP NKTRILKGCR ILHKFETLQP GESYIGVSST GTLLNTPICE PKNKWWEWSP NPSAPPEKEA S APTP UniProtKB: Putative primase C962R |
-分子 #2: DNA (32-MER)
| 分子 | 名称: DNA (32-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 9.689213 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT) |
-分子 #3: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 3 / コピー数: 6 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 64000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X