[日本語] English
 万見
万見- EMDB-35163: Cryo-EM structure of nanodisc (PE:PS:PC) reconstituted GLIC at pH 5.5 -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of nanodisc (PE:PS:PC) reconstituted GLIC at pH 5.5 | |||||||||
 マップデータ マップデータ | post process unmasked map of GLIC at pH 5.5 (PE:PS:PC) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pentameric ligand-gated ion channels / cis-loop / cryo-EM / nanodisc / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sodium channel activity / potassium channel activity / transmembrane transporter complex / extracellular ligand-gated monoatomic ion channel activity / transmembrane signaling receptor activity / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Gloeobacter violaceus (バクテリア) Gloeobacter violaceus (バクテリア) | |||||||||
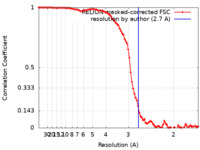
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Bharambe N / Li Z / Basak S | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structures of prokaryotic ligand-gated ion channel GLIC provide insights into gating in a lipid environment. 著者: Nikhil Bharambe / Zhuowen Li / David Seiferth / Asha Manikkoth Balakrishna / Philip C Biggin / Sandip Basak /   要旨: GLIC, a proton-activated prokaryotic ligand-gated ion channel, served as a model system for understanding the eukaryotic counterparts due to their structural and functional similarities. Despite ...GLIC, a proton-activated prokaryotic ligand-gated ion channel, served as a model system for understanding the eukaryotic counterparts due to their structural and functional similarities. Despite extensive studies conducted on GLIC, the molecular mechanism of channel gating in the lipid environment requires further investigation. Here, we present the cryo-EM structures of nanodisc-reconstituted GLIC at neutral and acidic pH in the resolution range of 2.6 - 3.4 Å. In our apo state at pH 7.5, the extracellular domain (ECD) displays conformational variations compared to the existing apo structures. At pH 4.0, three distinct conformational states (C1, C2 and O states) are identified. The protonated structures exhibit a compacted and counter-clockwise rotated ECD compared with our apo state. A gradual widening of the pore in the TMD is observed upon reducing the pH, with the widest pore in O state, accompanied by several layers of water pentagons. The pore radius and molecular dynamics (MD) simulations suggest that the O state represents an open conductive state. We also observe state-dependent interactions between several lipids and proteins that may be involved in the regulation of channel gating. Our results provide comprehensive insights into the importance of lipids impact on gating. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35163.map.gz emd_35163.map.gz | 33 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35163-v30.xml emd-35163-v30.xml emd-35163.xml emd-35163.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35163_fsc.xml emd_35163_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35163.png emd_35163.png | 131.9 KB | ||
| マスクデータ |  emd_35163_msk_1.map emd_35163_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-35163.cif.gz emd-35163.cif.gz | 6.2 KB | ||
| その他 |  emd_35163_additional_1.map.gz emd_35163_additional_1.map.gz emd_35163_half_map_1.map.gz emd_35163_half_map_1.map.gz emd_35163_half_map_2.map.gz emd_35163_half_map_2.map.gz | 32.1 MB 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35163 http://ftp.pdbj.org/pub/emdb/structures/EMD-35163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35163 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35163_validation.pdf.gz emd_35163_validation.pdf.gz | 895.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35163_full_validation.pdf.gz emd_35163_full_validation.pdf.gz | 895.1 KB | 表示 | |
| XML形式データ |  emd_35163_validation.xml.gz emd_35163_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_35163_validation.cif.gz emd_35163_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35163 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35163 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8i47MC  8i41C  8i42C  8i48C  8jj3C  8wcqC  8wcrC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35163.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35163.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post process unmasked map of GLIC at pH 5.5 (PE:PS:PC) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8452 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_35163_msk_1.map emd_35163_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: refinement map of GLIC at pH 5.5 (PE:PS:PC)
| ファイル | emd_35163_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | refinement map of GLIC at pH 5.5 (PE:PS:PC) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1 of GLIC at pH 5.5 (PE:PS:PC)
| ファイル | emd_35163_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 of GLIC at pH 5.5 (PE:PS:PC) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2 of GLIC at pH 5.5 (PE:PS:PC)
| ファイル | emd_35163_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 of GLIC at pH 5.5 (PE:PS:PC) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : pentameric ligand-gated ion channel
| 全体 | 名称: pentameric ligand-gated ion channel |
|---|---|
| 要素 |
|
-超分子 #1: pentameric ligand-gated ion channel
| 超分子 | 名称: pentameric ligand-gated ion channel / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Gloeobacter violaceus (バクテリア) Gloeobacter violaceus (バクテリア) |
-分子 #1: Proton-gated ion channel
| 分子 | 名称: Proton-gated ion channel / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Gloeobacter violaceus (バクテリア) Gloeobacter violaceus (バクテリア) |
| 分子量 | 理論値: 35.699094 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VSPPPPIADE PLTVNTGIYL IECYSLDDKA ETFKVNAFLS LSWKDRRLAF DPVRSGVRVK TYEPEAIWIP EIRFVNVENA RDADVVDIS VSPDGTVQYL ERFSARVLSP LDFRRYPFDS QTLHIYLIVR SVDTRNIVLA VDLEKVGKND DVFLTGWDIE S FTAVVKPA ...文字列: VSPPPPIADE PLTVNTGIYL IECYSLDDKA ETFKVNAFLS LSWKDRRLAF DPVRSGVRVK TYEPEAIWIP EIRFVNVENA RDADVVDIS VSPDGTVQYL ERFSARVLSP LDFRRYPFDS QTLHIYLIVR SVDTRNIVLA VDLEKVGKND DVFLTGWDIE S FTAVVKPA NFALEDRLES KLDYQLRISR QYFSYIPNII LPMLFILFIS WTAFWSTSYE ANVTLVVSTL IAHIAFNILV ET NLPKTPY MTYTGAIIFM IYLFYFVAVI EVTVQHYLKV ESQPARAASI TRASRIAFPV VFLLANIILA FLFFG UniProtKB: Proton-gated ion channel |
-分子 #2: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine
| 分子 | 名称: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine / タイプ: ligand / ID: 2 / コピー数: 30 / 式: PEE |
|---|---|
| 分子量 | 理論値: 744.034 Da |
| Chemical component information |  ChemComp-PEE: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 30 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 5.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE / 支持フィルム - トポロジー: CONTINUOUS | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 6625 / 平均露光時間: 7.0 sec. / 平均電子線量: 57.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)