+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of abscisic acid transporter AtABCG25 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transport / ABC transporter / plant hormone / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | Protein of unknown function DUF1425 / YcfL-like superfamily / Protein of unknown function (DUF1425) / Prokaryotic membrane lipoprotein lipid attachment site profile. / DUF1425 domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
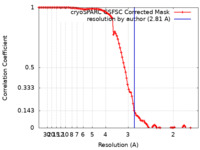
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.81 Å | |||||||||
 データ登録者 データ登録者 | Huang X / Zhang X / Zhang P | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2023 ジャーナル: Nat Plants / 年: 2023タイトル: Cryo-EM structure and molecular mechanism of abscisic acid transporter ABCG25. 著者: Xiaowei Huang / Xue Zhang / Ning An / Minhua Zhang / Miaolian Ma / Yang Yang / Lianyan Jing / Yongfei Wang / Zhenguo Chen / Peng Zhang /  要旨: Abscisic acid (ABA) is one of the plant hormones that regulate various physiological processes, including stomatal closure, seed germination and development. ABA is synthesized mainly in vascular ...Abscisic acid (ABA) is one of the plant hormones that regulate various physiological processes, including stomatal closure, seed germination and development. ABA is synthesized mainly in vascular tissues and transported to distal sites to exert its physiological functions. Many ABA transporters have been identified, however, the molecular mechanism of ABA transport remains elusive. Here we report the cryogenic electron microscopy structure of the Arabidopsis thaliana adenosine triphosphate-binding cassette G subfamily ABA exporter ABCG25 (AtABCG25) in inward-facing apo conformation, ABA-bound pre-translocation conformation and outward-facing occluded conformation. Structural and biochemical analyses reveal that the ABA bound with ABCG25 adopts a similar configuration as that in ABA receptors and that the ABA-specific binding is dictated by residues from transmembrane helices TM1, TM2 and TM5a of each protomer at the transmembrane domain interface. Comparison of different conformational structures reveals conformational changes, especially those of transmembrane helices and residues constituting the substrate translocation pathway during the cross-membrane transport process. Based on the structural data, a 'gate-flipper' translocation model of ABCG25-mediated ABA cross-membrane transport is proposed. Our structural data on AtABCG25 provide new clues to the physiological study of ABA and shed light on its potential applications in plants and agriculture. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35150.map.gz emd_35150.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35150-v30.xml emd-35150-v30.xml emd-35150.xml emd-35150.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35150_fsc.xml emd_35150_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35150.png emd_35150.png | 79.4 KB | ||
| Filedesc metadata |  emd-35150.cif.gz emd-35150.cif.gz | 5.3 KB | ||
| その他 |  emd_35150_additional_1.map.gz emd_35150_additional_1.map.gz emd_35150_half_map_1.map.gz emd_35150_half_map_1.map.gz emd_35150_half_map_2.map.gz emd_35150_half_map_2.map.gz | 50.8 MB 95.4 MB 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35150 http://ftp.pdbj.org/pub/emdb/structures/EMD-35150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35150 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35150 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35150_validation.pdf.gz emd_35150_validation.pdf.gz | 924.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35150_full_validation.pdf.gz emd_35150_full_validation.pdf.gz | 923.7 KB | 表示 | |
| XML形式データ |  emd_35150_validation.xml.gz emd_35150_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_35150_validation.cif.gz emd_35150_validation.cif.gz | 22.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35150 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35150 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35150 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35150.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35150.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
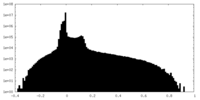
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_35150_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
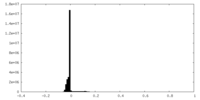
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35150_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
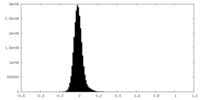
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35150_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
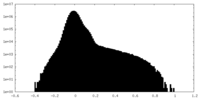
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Arabidopsis thaliana ABCG25
| 全体 | 名称: Arabidopsis thaliana ABCG25 |
|---|---|
| 要素 |
|
-超分子 #1: Arabidopsis thaliana ABCG25
| 超分子 | 名称: Arabidopsis thaliana ABCG25 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: ABC transporter G family member 25
| 分子 | 名称: ABC transporter G family member 25 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 72.983867 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAFDGVENQ MNGPDSSPRL SQDPREPRSL LSSSCFPITL KFVDVCYRVK IHGMSNDSCN IKKLLGLKQK PSDETRSTEE RTILSGVTG MISPGEFMAV LGPSGSGKST LLNAVAGRLH GSNLTGKILI NDGKITKQTL KRTGFVAQDD LLYPHLTVRE T LVFVALLR ...文字列: MSAFDGVENQ MNGPDSSPRL SQDPREPRSL LSSSCFPITL KFVDVCYRVK IHGMSNDSCN IKKLLGLKQK PSDETRSTEE RTILSGVTG MISPGEFMAV LGPSGSGKST LLNAVAGRLH GSNLTGKILI NDGKITKQTL KRTGFVAQDD LLYPHLTVRE T LVFVALLR LPRSLTRDVK LRAAESVISE LGLTKCENTV VGNTFIRGIS GGERKRVSIA HELLINPSLL VLDEPTSGLD AT AALRLVQ TLAGLAHGKG KTVVTSIHQP SSRVFQMFDT VLLLSEGKCL FVGKGRDAMA YFESVGFSPA FPMNPADFLL DLA NGVCQT DGVTEREKPN VRQTLVTAYD TLLAPQVKTC IEVSHFPQDN ARFVKTRVNG GGITTCIATW FSQLCILLHR LLKE RRHES FDLLRIFQVV AASILCGLMW WHSDYRDVHD RLGLLFFISI FWGVLPSFNA VFTFPQERAI FTRERASGMY TLSSY FMAH VLGSLSMELV LPASFLTFTY WMVYLRPGIV PFLLTLSVLL LYVLASQGLG LALGAAIMDA KKASTIVTVT MLAFVL TGG YYVNKVPSGM VWMKYVSTTF YCYRLLVAIQ YGSGEEILRM LGCDSKGKQG ASAATSAGCR FVEEEVIGDV GMWTSVG VL FLMFFGYRVL AYLALRRIKH UniProtKB: DUF1425 domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 58.62 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)