+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | F1 domain of FoF1-ATPase from Bacillus PS3,120 degrees,highATP | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | ATP synthase F1 ATPase FoF1 / MOTOR PROTEIN / TRANSLOCASE-MOTOR PROTEIN complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton motive force-driven plasma membrane ATP synthesis / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / ADP binding / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
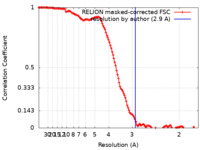
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||
 データ登録者 データ登録者 | Nakano A / Kishikawa J / Nakanishi A / Mitsuoka K / Yokoyama K | |||||||||||||||
| 資金援助 |  日本, 4件 日本, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Mechanism of ATP hydrolysis dependent rotation of bacterial ATP synthase. 著者: Atsuki Nakano / Jun-Ichi Kishikawa / Kaoru Mitsuoka / Ken Yokoyama /  要旨: F domain of ATP synthase is a rotary ATPase complex in which rotation of central γ-subunit proceeds in 120° steps against a surrounding αβ fueled by ATP hydrolysis. How the ATP hydrolysis ...F domain of ATP synthase is a rotary ATPase complex in which rotation of central γ-subunit proceeds in 120° steps against a surrounding αβ fueled by ATP hydrolysis. How the ATP hydrolysis reactions occurring in three catalytic αβ dimers are coupled to mechanical rotation is a key outstanding question. Here we describe catalytic intermediates of the F domain in FF synthase from Bacillus PS3 sp. during ATP mediated rotation captured using cryo-EM. The structures reveal that three catalytic events and the first 80° rotation occur simultaneously in F domain when nucleotides are bound at all the three catalytic αβ dimers. The remaining 40° rotation of the complete 120° step is driven by completion of ATP hydrolysis at αβ, and proceeds through three sub-steps (83°, 91°, 101°, and 120°) with three associated conformational intermediates. All sub-steps except for one between 91° and 101° associated with phosphate release, occur independently of the chemical cycle, suggesting that the 40° rotation is largely driven by release of intramolecular strain accumulated by the 80° rotation. Together with our previous results, these findings provide the molecular basis of ATP driven rotation of ATP synthases. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34752.map.gz emd_34752.map.gz | 141.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34752-v30.xml emd-34752-v30.xml emd-34752.xml emd-34752.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34752_fsc.xml emd_34752_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34752.png emd_34752.png | 83.7 KB | ||
| マスクデータ |  emd_34752_msk_1.map emd_34752_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34752.cif.gz emd-34752.cif.gz | 6.8 KB | ||
| その他 |  emd_34752_additional_1.map.gz emd_34752_additional_1.map.gz emd_34752_half_map_1.map.gz emd_34752_half_map_1.map.gz emd_34752_half_map_2.map.gz emd_34752_half_map_2.map.gz | 165.3 MB 141.6 MB 141.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34752 http://ftp.pdbj.org/pub/emdb/structures/EMD-34752 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34752 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34752 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34752_validation.pdf.gz emd_34752_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34752_full_validation.pdf.gz emd_34752_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_34752_validation.xml.gz emd_34752_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_34752_validation.cif.gz emd_34752_validation.cif.gz | 26.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34752 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34752 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34752 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34752 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hh5MC  8hh1C  8hh2C  8hh3C  8hh4C  8hh6C  8hh7C  8hh8C  8hh9C  8hhaC  8hhbC  8hhcC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34752.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34752.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34752_msk_1.map emd_34752_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_34752_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34752_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34752_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : FoF1 from Bacillus PS3
| 全体 | 名称: FoF1 from Bacillus PS3 |
|---|---|
| 要素 |
|
-超分子 #1: FoF1 from Bacillus PS3
| 超分子 | 名称: FoF1 from Bacillus PS3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 530 KDa |
-分子 #1: ATP synthase subunit alpha
| 分子 | 名称: ATP synthase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: H+-transporting two-sector ATPase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 54.717398 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SIRAEEISAL IKQQIENYES QIQVSDVGTV IQVGDGIARA HGLDNVMSGE LVEFANGVMG MALNLEENNV GIVILGPYTG IKEGDEVRR TGRIMEVPVG EALIGRVVNP LGQPVDGLGP VETTETRPIE SPAPGVMDRR SVHEPLQTGI KAIDALVPIG R GQRELIIG ...文字列: SIRAEEISAL IKQQIENYES QIQVSDVGTV IQVGDGIARA HGLDNVMSGE LVEFANGVMG MALNLEENNV GIVILGPYTG IKEGDEVRR TGRIMEVPVG EALIGRVVNP LGQPVDGLGP VETTETRPIE SPAPGVMDRR SVHEPLQTGI KAIDALVPIG R GQRELIIG DRQTGKTSVA IDTIINQKDQ NMISIYVAIG QKESTVRTVV ETLRKHGALD YTIVVTASAS QPAPLLFLAP YA GVAMGEY FMYKGKHVLV VYDDLSKQAA AYRELSLLLR RPPGREAYPG DIFYLHSRLL ERAAKLSDAK GGGSLTALPF VET QAGDIS AYIPTNVISI TDGQIFLQSD LFFSGVRPAI NAGLSVSRVG GAAQIKAMKK VAGTLRLDLA AYRELEAFAQ FGSD LDKAT QAKLARGART VEVLKQDLHQ PIPVEKQVLI IYALTRGFLD DIPVEDVRRF EKEFYLFLDQ NGQHLLEHIR TTKDL PNED DLNKAIEAFK KTFVVSQ UniProtKB: ATP synthase subunit alpha |
-分子 #2: ATP synthase subunit beta
| 分子 | 名称: ATP synthase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号: H+-transporting two-sector ATPase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.424625 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHHHH HMTRGRVIQV MGPVVDVKFE NGHLPAIYNA LKIQHKARNE NEVDIDLTLE VALHLGDDTV RTIAMASTDG LIRGMEVID TGAPISVPVG EVTLGRVFNV LGEPIDLEGD IPADARRDPI HRPAPKFEEL ATEVEILETG IKVVDLLAPY I KGGKIGLF ...文字列: MHHHHHHHHH HMTRGRVIQV MGPVVDVKFE NGHLPAIYNA LKIQHKARNE NEVDIDLTLE VALHLGDDTV RTIAMASTDG LIRGMEVID TGAPISVPVG EVTLGRVFNV LGEPIDLEGD IPADARRDPI HRPAPKFEEL ATEVEILETG IKVVDLLAPY I KGGKIGLF GGAGVGKTVL IQELIHNIAQ EHGGISVFAG VGERTREGND LYHEMKDSGV ISKTAMVFGQ MNEPPGARMR VA LTGLTMA EYFRDEQGQD VLLFIDNIFR FTQAGSEVSA LLGRMPSAVG YQPTLATEMG QLQERITSTA KGSITSIQAI YVP ADDYTD PAPATTFSHL DATTNLERKL AEMGIYPAVD PLASTSRALA PEIVGEEHYQ VARKVQQTLQ RYKELQDIIA ILGM DELSD EDKLVVHRAR RIQFFLSQNF HVAEQFTGQP GSYVPVKETV RGFKEILEGK YDHLPEDAFR LVGRIEEVVE KAKAM GVEV UniProtKB: ATP synthase subunit beta |
-分子 #3: ATP synthase gamma chain
| 分子 | 名称: ATP synthase gamma chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 31.728328 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ASLRDIKTRI NATKKTSQIT KAMEMVSTSK LNRAEQNAKS FVPYMEKIQE VVANVALGAG GASHPMLVSR PVKKTGYLVI TSDRGLAGA YNSNVLRLVY QTIQKRHASP DEYAIIVIGR VGLSFFRKRN MPVILDITRL PDQPSFADIK EIARKTVGLF A DGTFDELY ...文字列: ASLRDIKTRI NATKKTSQIT KAMEMVSTSK LNRAEQNAKS FVPYMEKIQE VVANVALGAG GASHPMLVSR PVKKTGYLVI TSDRGLAGA YNSNVLRLVY QTIQKRHASP DEYAIIVIGR VGLSFFRKRN MPVILDITRL PDQPSFADIK EIARKTVGLF A DGTFDELY MYYNHYVSAI QQEVTERKLL PLTDLAENKQ RTVYEFEPSQ EEILDVLLPQ YAESLIYGAL LDAKASEHAA RM TAMKNAT DNANELIRTL TLSYNRARQA AITQEITEIV AGANALQ UniProtKB: ATP synthase gamma chain |
-分子 #4: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 5 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-分子 #7: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 5922 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.03843 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)