+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of yeast Erf2/Erf4 complex | |||||||||
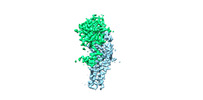
 マップデータ マップデータ | the yeast refined map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | palmitoyltransferase / Saccaromyces cerevisiae / palmitoylation / RAS2 / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endoplasmic reticulum palmitoyltransferase complex / protein S-acyltransferase / protein palmitoylation / protein-cysteine S-palmitoyltransferase activity / palmitoyltransferase activity / perinuclear endoplasmic reticulum / cortical endoplasmic reticulum / protein targeting to membrane / Neutrophil degranulation / endoplasmic reticulum membrane ...endoplasmic reticulum palmitoyltransferase complex / protein S-acyltransferase / protein palmitoylation / protein-cysteine S-palmitoyltransferase activity / palmitoyltransferase activity / perinuclear endoplasmic reticulum / cortical endoplasmic reticulum / protein targeting to membrane / Neutrophil degranulation / endoplasmic reticulum membrane / endoplasmic reticulum / Golgi apparatus 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Wu J / Hu Q / Zhang Y / Yang A / Liu S | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Regulation of RAS palmitoyltransferases by accessory proteins and palmitoylation. 著者: Anlan Yang / Shengjie Liu / Yuqi Zhang / Jia Chen / Yujing Fan / Fengxiang Wang / Yilong Zou / Shan Feng / Jianping Wu / Qi Hu /  要旨: Palmitoylation of cysteine residues at the C-terminal hypervariable regions in human HRAS and NRAS, which is necessary for RAS signaling, is catalyzed by the acyltransferase DHHC9 in complex with its ...Palmitoylation of cysteine residues at the C-terminal hypervariable regions in human HRAS and NRAS, which is necessary for RAS signaling, is catalyzed by the acyltransferase DHHC9 in complex with its accessory protein GCP16. The molecular basis for the acyltransferase activity and the regulation of DHHC9 by GCP16 is not clear. Here we report the cryo-electron microscopy structures of the human DHHC9-GCP16 complex and its yeast counterpart-the Erf2-Erf4 complex, demonstrating that GCP16 and Erf4 are not directly involved in the catalytic process but stabilize the architecture of DHHC9 and Erf2, respectively. We found that a phospholipid binding to an arginine-rich region of DHHC9 and palmitoylation on three residues (C24, C25 and C288) were essential for the catalytic activity of the DHHC9-GCP16 complex. Moreover, we showed that GCP16 also formed complexes with DHHC14 and DHHC18 to catalyze RAS palmitoylation. These findings provide insights into the regulatory mechanism of RAS palmitoyltransferases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34717.map.gz emd_34717.map.gz | 25.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34717-v30.xml emd-34717-v30.xml emd-34717.xml emd-34717.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34717.png emd_34717.png | 21.7 KB | ||
| Filedesc metadata |  emd-34717.cif.gz emd-34717.cif.gz | 6.4 KB | ||
| その他 |  emd_34717_half_map_1.map.gz emd_34717_half_map_1.map.gz emd_34717_half_map_2.map.gz emd_34717_half_map_2.map.gz | 25.1 MB 25.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34717 http://ftp.pdbj.org/pub/emdb/structures/EMD-34717 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34717 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34717 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34717_validation.pdf.gz emd_34717_validation.pdf.gz | 717.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34717_full_validation.pdf.gz emd_34717_full_validation.pdf.gz | 717 KB | 表示 | |
| XML形式データ |  emd_34717_validation.xml.gz emd_34717_validation.xml.gz | 10.5 KB | 表示 | |
| CIF形式データ |  emd_34717_validation.cif.gz emd_34717_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34717 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34717 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34717 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34717 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hfcMC  8hf3C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34717.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34717.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | the yeast refined map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.087 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map B
| ファイル | emd_34717_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_34717_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Erf2/Erf4 complex
| 全体 | 名称: Erf2/Erf4 complex |
|---|---|
| 要素 |
|
-超分子 #1: Erf2/Erf4 complex
| 超分子 | 名称: Erf2/Erf4 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Palmitoyltransferase ERF2
| 分子 | 名称: Palmitoyltransferase ERF2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: protein S-acyltransferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.811672 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHHH HHHHGSDEVD AHMALVSRRS TRSESTSITK EEHTGEGSLT KLFFRWLVTL EGDQDINDGK GYISLPNVSN YIFFLGGRF RTVKGAKPLW LGVLLAIVCP MVLFSIFEAH KLWHTQNGYK VLVIFFYYFW VITLASFIRT ATSDPGVLPR N IHLSQLRN ...文字列: MGHHHHHHHH HHHHGSDEVD AHMALVSRRS TRSESTSITK EEHTGEGSLT KLFFRWLVTL EGDQDINDGK GYISLPNVSN YIFFLGGRF RTVKGAKPLW LGVLLAIVCP MVLFSIFEAH KLWHTQNGYK VLVIFFYYFW VITLASFIRT ATSDPGVLPR N IHLSQLRN NYQIPQEYYN LITLPTHSSI SKDITIKYCP SCRIWRPPRS SHCSTCNVCV MVHDHHCIWV NNCIGKRNYR FF LIFLLGA ILSSVILLTN CAIHIARESG GPRDCPVAIL LLCYAGLTLW YPAILFTYHI FMAGNQQTTR EFLKGIGSKK NPV FHRVVK EENIYNKGSF LKNMGHLMLE PRGPSFVSAR KPHEAGDWRF MDLSPAHSFE KIQKI UniProtKB: Palmitoyltransferase ERF2 |
-分子 #2: Ras modification protein ERF4
| 分子 | 名称: Ras modification protein ERF4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 29.652705 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDYKDDDDKG SDYKDDDDKG SDEVDAHMCD SHQKEEDNAN TSERALFFNY HEFSYSFYED LGSEDAKPTE HDEDHKLCIT HFPNVYAAR GSAEFQVTRV VRVPRRFDES RSSLETPQFS TQLPGSEPAA IVGDDGTSFV RCGRYDIGDH VFGCSSVSPL S EYLSAAEL ...文字列: MDYKDDDDKG SDYKDDDDKG SDEVDAHMCD SHQKEEDNAN TSERALFFNY HEFSYSFYED LGSEDAKPTE HDEDHKLCIT HFPNVYAAR GSAEFQVTRV VRVPRRFDES RSSLETPQFS TQLPGSEPAA IVGDDGTSFV RCGRYDIGDH VFGCSSVSPL S EYLSAAEL AEVVHRVNGF LLREEGEVFG WRNLSGLLLD MLTGGLWSWV LGPLLSRPVF QESLALEQYV AQLNSPGGLL HE RGVRLVL PRRSGCLSLD FVVPRPK UniProtKB: Ras modification protein ERF4 |
-分子 #3: PALMITIC ACID
| 分子 | 名称: PALMITIC ACID / タイプ: ligand / ID: 3 / コピー数: 1 / 式: PLM |
|---|---|
| 分子量 | 理論値: 256.424 Da |
| Chemical component information |  ChemComp-PLM: |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)