+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
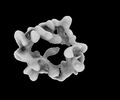
| タイトル | Structure of Crm1-RanGTP complex | |||||||||
 マップデータ マップデータ | Crm1-RanGTP complex half A map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nuclear export factor / NUCLEAR PROTEIN | |||||||||
| 生物種 |  | |||||||||
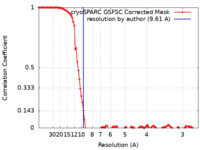
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.61 Å | |||||||||
 データ登録者 データ登録者 | Li ZQ / Chen SJ / Sui SF | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Nuclear export of pre-60S particles through the nuclear pore complex. 著者: Zongqiang Li / Shuaijiabin Chen / Liang Zhao / Guoqiang Huang / Huiqin Xu / Xiaoyun Yang / Peiyi Wang / Ning Gao / Sen-Fang Sui /  要旨: The nuclear pore complex (NPC) is the bidirectional gate that mediates the exchange of macromolecules or their assemblies between nucleus and cytoplasm. The assembly intermediates of the ribosomal ...The nuclear pore complex (NPC) is the bidirectional gate that mediates the exchange of macromolecules or their assemblies between nucleus and cytoplasm. The assembly intermediates of the ribosomal subunits, pre-60S and pre-40S particles, are among the largest cargoes of the NPC and the export of these gigantic ribonucleoproteins requires numerous export factors. Here we report the cryo-electron microscopy structure of native pre-60S particles trapped in the channel of yeast NPCs. In addition to known assembly factors, multiple factors with export functions are also included in the structure. These factors in general bind to either the flexible regions or subunit interface of the pre-60S particle, and virtually form many anchor sites for NPC binding. Through interactions with phenylalanine-glycine (FG) repeats from various nucleoporins of NPC, these factors collectively facilitate the passage of the pre-60S particle through the central FG repeat network of the NPC. Moreover, in silico analysis of the axial and radial distribution of pre-60S particles within the NPC shows that a single NPC can take up to four pre-60S particles simultaneously, and pre-60S particles are enriched in the inner ring regions close to the wall of the NPC with the solvent-exposed surface facing the centre of the nuclear pore. Our data suggest a translocation model for the export of pre-60S particles through the NPC. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34641.map.gz emd_34641.map.gz | 60.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34641-v30.xml emd-34641-v30.xml emd-34641.xml emd-34641.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34641_fsc.xml emd_34641_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34641.png emd_34641.png | 33.9 KB | ||
| その他 |  emd_34641_half_map_1.map.gz emd_34641_half_map_1.map.gz emd_34641_half_map_2.map.gz emd_34641_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34641 http://ftp.pdbj.org/pub/emdb/structures/EMD-34641 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34641 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34641 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34641_validation.pdf.gz emd_34641_validation.pdf.gz | 683.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34641_full_validation.pdf.gz emd_34641_full_validation.pdf.gz | 683 KB | 表示 | |
| XML形式データ |  emd_34641_validation.xml.gz emd_34641_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_34641_validation.cif.gz emd_34641_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34641 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34641 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34641 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34641 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34641.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34641.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Crm1-RanGTP complex half A map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.668 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Crm1-RanGTP complex
| ファイル | emd_34641_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Crm1-RanGTP complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
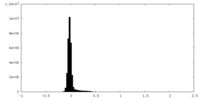
| 密度ヒストグラム |
-ハーフマップ: Crm1-RanGTP complex half B map
| ファイル | emd_34641_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Crm1-RanGTP complex half B map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
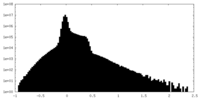
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Crm1-RanGTP complex
| 全体 | 名称: Crm1-RanGTP complex |
|---|---|
| 要素 |
|
-超分子 #1: Crm1-RanGTP complex
| 超分子 | 名称: Crm1-RanGTP complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Nuclear export factor
| 分子 | 名称: Nuclear export factor / タイプ: protein_or_peptide / ID: 1 詳細: Author stated this map is a local refinement map from the peripheric region of the larger pre-60S particle (EMD-34725) with a high resolution. It is likely that the unmasked-calculated curve ...詳細: Author stated this map is a local refinement map from the peripheric region of the larger pre-60S particle (EMD-34725) with a high resolution. It is likely that the unmasked-calculated curve has a higher resolution due to the influence of pre-60S particle. 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MEGILDFSND LDIALLDQVV STFYQGSGVQ QKQAQEILTK FQDNPDAWQK ADQILQFSTN PQSKFIALS ILDKLITRKW KLLPNDHRIG IRNFVVGMII SMCQDDEVFK TQKNLINKSD L TLVQILKQ EWPQNWPEFI PELIGSSSSS VNVCENNMIV LKLLSEEVFD ...文字列: MEGILDFSND LDIALLDQVV STFYQGSGVQ QKQAQEILTK FQDNPDAWQK ADQILQFSTN PQSKFIALS ILDKLITRKW KLLPNDHRIG IRNFVVGMII SMCQDDEVFK TQKNLINKSD L TLVQILKQ EWPQNWPEFI PELIGSSSSS VNVCENNMIV LKLLSEEVFD FSAEQMTQAK AL HLKNSMS KEFEQIFKLC FQVLEQGSSS SLIVATLESL LRYLHWIPYR YIYETNILEL LST KFMTSP DTRAITLKCL TEVSNLKIPQ DNDLIKRQTV LFFQNTLQQI ATSVMPVTAD LKAT YANAN GNDQSFLQDL AMFLTTYLAR NRALLESDES LRELLLNAHQ YLIQLSKIEE RELFK TTLD YWHNLVADLF YEVQRLPATE MSPLIQLSVG SQAISTGSGA LNPEYMKRFP LKKHIY EEI CSQLRLVIIE NMVRPEEVLV VENDEGEIVR EFVKESDTIQ LYKSEREVLV YLTHLNV ID TEEIMISKLA RQIDGSEWSW HNINTLSWAI GSISGTMSED TEKRFVVTVI KDLLDLTV K KRGKDNKAVV ASDIMYVVGQ YPRFLKAHWN FLRTVILKLF EFMHETHEGV QDMACDTFI KIVQKCKYHF VIQQPRESEP FIQTIIRDIQ KTTADLQPQQ VHTFYKACGI IISEERSVAE RNRLLSDLM QLPNMAWDTI VEQSTANPTL LLDSETVKII ANIIKTNVAV CTSMGADFYP Q LGHIYYNM LQLYRAVSSM ISAQVAAEGL IATKTPKVRG LRTIKKEILK LVETYISKAR NL DDVVKVL VEPLLNAVLE DYMNNVPDAR DAEVLNCMTT VVEKVGHMIP QGVILILQSV FEC TLDMIN KDFTEYPEHR VEFYKLLKVI NEKSFAAFLE LPPAAFKLFV DAICWAFKHN NRDV EVNGL QIALDLVKNI ERMGNVPFAN EFHKNYFFIF VSETFFVLTD SDHKSGFSKQ ALLLM KLIS LVYDNKISVP LYQEAEVPQG TSNQVYLSQY LANMLSNAFP HLTSEQIASF LSALTK QYK DLVVFKGTLR DFLVQIKEVG GDPTDYLFAE DKENALMEQN RLEREKAAKI GGLLKPS EL DD |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)