[English] 日本語
 Yorodumi
Yorodumi- EMDB-34386: Cryo-EM map of Tetrahymena thermophila respiratory complex I memb... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM map of Tetrahymena thermophila respiratory complex I membrane arm distal portion, digitonin sample | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Electron transport chain / supercomplex / membrane protein / Tetrahymena thermophila / ELECTRON TRANSPORT | |||||||||
| Biological species |  | |||||||||
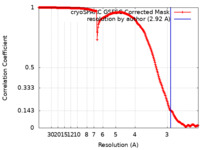
| Method | single particle reconstruction / cryo EM / Resolution: 2.92 Å | |||||||||
 Authors Authors | Zhou L | |||||||||
| Funding support | 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2023 Journal: Nat Commun / Year: 2023Title: Structures of Tetrahymena thermophila respiratory megacomplexes on the tubular mitochondrial cristae. Authors: Fangzhu Han / Yiqi Hu / Mengchen Wu / Zhaoxiang He / Hongtao Tian / Long Zhou /  Abstract: Tetrahymena thermophila, a classic ciliate model organism, has been shown to possess tubular mitochondrial cristae and highly divergent electron transport chain involving four transmembrane protein ...Tetrahymena thermophila, a classic ciliate model organism, has been shown to possess tubular mitochondrial cristae and highly divergent electron transport chain involving four transmembrane protein complexes (I-IV). Here we report cryo-EM structures of its ~8 MDa megacomplex IV+ (I + III+ II), as well as a ~ 10.6 MDa megacomplex (IV + I + III+ II) at lower resolution. In megacomplex IV+ (I + III+ II), each CIV protomer associates one copy of supercomplex I + III and one copy of CII, forming a half ring-shaped architecture that adapts to the membrane curvature of mitochondrial cristae. Megacomplex (IV+ I + III+ II) defines the relative position between neighbouring half rings and maintains the proximity between CIV and CIII cytochrome c binding sites. Our findings expand the current understanding of divergence in eukaryotic electron transport chain organization and how it is related to mitochondrial morphology. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_34386.map.gz emd_34386.map.gz | 1.2 GB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-34386-v30.xml emd-34386-v30.xml emd-34386.xml emd-34386.xml | 16.3 KB 16.3 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_34386_fsc.xml emd_34386_fsc.xml | 23 KB | Display |  FSC data file FSC data file |
| Images |  emd_34386.png emd_34386.png | 30.3 KB | ||
| Others |  emd_34386_half_map_1.map.gz emd_34386_half_map_1.map.gz emd_34386_half_map_2.map.gz emd_34386_half_map_2.map.gz | 1.2 GB 1.2 GB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34386 http://ftp.pdbj.org/pub/emdb/structures/EMD-34386 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34386 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34386 | HTTPS FTP |
-Validation report
| Summary document |  emd_34386_validation.pdf.gz emd_34386_validation.pdf.gz | 1.1 MB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_34386_full_validation.pdf.gz emd_34386_full_validation.pdf.gz | 1.1 MB | Display | |
| Data in XML |  emd_34386_validation.xml.gz emd_34386_validation.xml.gz | 32.2 KB | Display | |
| Data in CIF |  emd_34386_validation.cif.gz emd_34386_validation.cif.gz | 42.8 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34386 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34386 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34386 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34386 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_34386.map.gz / Format: CCP4 / Size: 1.3 GB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_34386.map.gz / Format: CCP4 / Size: 1.3 GB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.2 Å | ||||||||||||||||||||||||||||||||||||
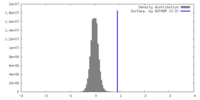
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #1
| File | emd_34386_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
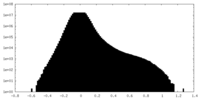
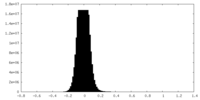
| Density Histograms |
-Half map: #2
| File | emd_34386_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
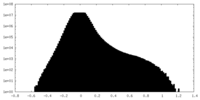
| Density Histograms |
- Sample components
Sample components
-Entire : Tetrahymena thermophila respiratory complex I membrane arm distal...
| Entire | Name: Tetrahymena thermophila respiratory complex I membrane arm distal portion, digitonin sample |
|---|---|
| Components |
|
-Supramolecule #1: Tetrahymena thermophila respiratory complex I membrane arm distal...
| Supramolecule | Name: Tetrahymena thermophila respiratory complex I membrane arm distal portion, digitonin sample type: complex / ID: 1 / Parent: 0 |
|---|---|
| Source (natural) | Organism:  |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 1.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.4 Component:
Details: SEC buffer (20 mM Tris pH 7.4, 50 mM NaCl, 0.002% PMSF, 0.1% digitonin (w/v)) | |||||||||||||||
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Support film - Material: CARBON / Support film - topology: CONTINUOUS / Support film - Film thickness: 2 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 15 sec. / Pretreatment - Atmosphere: AIR / Pretreatment - Pressure: 0.039 kPa / Details: 15 mA | |||||||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277.15 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Digitization - Dimensions - Width: 4096 pixel / Digitization - Dimensions - Height: 4096 pixel / Number grids imaged: 1 / Number real images: 11182 / Average exposure time: 8.46 sec. / Average electron dose: 51.4 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.0 µm / Nominal defocus min: 0.8 µm / Nominal magnification: 140000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)