+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of Membrane-Bound Alcohol Dehydrogenase from Gluconobacter oxydans | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / Oxidereductase / Membrane-bound protein / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alcohol dehydrogenase (quinone) / oxidoreductase activity, acting on CH-OH group of donors / outer membrane-bounded periplasmic space / electron transfer activity / iron ion binding / heme binding / calcium ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Gluconobacter oxydans (バクテリア) / Gluconobacter oxydans (バクテリア) /  Gluconobacter oxydans 621H (バクテリア) Gluconobacter oxydans 621H (バクテリア) | |||||||||
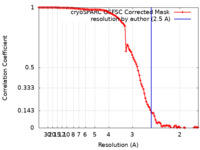
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Adachi T / Miyata T / Makino F / Tanaka H / Namba K / Sowa K / Kitazumi Y / Shirai O | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Acs Catalysis / 年: 2023 ジャーナル: Acs Catalysis / 年: 2023タイトル: Experimental and Theoretical Insights into Bienzymatic Cascade for Mediatorless Bioelectrochemical Ethanol Oxidation with Alcohol and Aldehyde Dehydrogenases 著者: Adachi T / Miyata T / Makino F / Tanaka H / Namba K / Kano K / Sowa K / Kitazumi Y / Shirai O | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34368.map.gz emd_34368.map.gz | 62.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34368-v30.xml emd-34368-v30.xml emd-34368.xml emd-34368.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34368_fsc.xml emd_34368_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34368.png emd_34368.png | 90.9 KB | ||
| マスクデータ |  emd_34368_msk_1.map emd_34368_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_34368_half_map_1.map.gz emd_34368_half_map_1.map.gz emd_34368_half_map_2.map.gz emd_34368_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34368 http://ftp.pdbj.org/pub/emdb/structures/EMD-34368 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34368 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34368 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34368_validation.pdf.gz emd_34368_validation.pdf.gz | 919.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34368_full_validation.pdf.gz emd_34368_full_validation.pdf.gz | 919 KB | 表示 | |
| XML形式データ |  emd_34368_validation.xml.gz emd_34368_validation.xml.gz | 19.1 KB | 表示 | |
| CIF形式データ |  emd_34368_validation.cif.gz emd_34368_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34368 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34368 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34368 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34368 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gy2MC  8gy3C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34368.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34368.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34368_msk_1.map emd_34368_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34368_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34368_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Alcohol dehydrogenase from Gluconobacter oxydans
| 全体 | 名称: Alcohol dehydrogenase from Gluconobacter oxydans |
|---|---|
| 要素 |
|
-超分子 #1: Alcohol dehydrogenase from Gluconobacter oxydans
| 超分子 | 名称: Alcohol dehydrogenase from Gluconobacter oxydans / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Gluconobacter oxydans (バクテリア) Gluconobacter oxydans (バクテリア) |
| 分子量 | 理論値: 150 KDa |
-分子 #1: Alcohol dehydrogenase (quinone), dehydrogenase subunit
| 分子 | 名称: Alcohol dehydrogenase (quinone), dehydrogenase subunit タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: alcohol dehydrogenase (quinone) |
|---|---|
| 由来(天然) | 生物種:  Gluconobacter oxydans 621H (バクテリア) / 株: 621H Gluconobacter oxydans 621H (バクテリア) / 株: 621H |
| 分子量 | 理論値: 82.938906 KDa |
| 組換発現 | 生物種:  Gluconobacter oxydans (バクテリア) Gluconobacter oxydans (バクテリア) |
| 配列 | 文字列: MTSGLLTPIK VTKKRLLSCA AALAFSAAVP VAFAQEDTGT AITSSDNGGH PGDWLSYGRS YSEQRYSPLD QINTENVGKL KLAWHYDLD TNRGQEGTPL IVNGVMYATT NWSKMKALDA ATGKLLWSYD PKVPGNIADR GCCDTVSRGA AYWNGKVYFG T FDGRLIAL ...文字列: MTSGLLTPIK VTKKRLLSCA AALAFSAAVP VAFAQEDTGT AITSSDNGGH PGDWLSYGRS YSEQRYSPLD QINTENVGKL KLAWHYDLD TNRGQEGTPL IVNGVMYATT NWSKMKALDA ATGKLLWSYD PKVPGNIADR GCCDTVSRGA AYWNGKVYFG T FDGRLIAL DAKTGKLVWS VYTIPKEAQL GHQRSYTVDG APRIAKGKVL IGNGGAEFGA RGFVSAFDAE TGKLDWRFFT VP NPENKPD GAASDDILMS KAYPTWGKNG AWKQQGGGGT VWDSLVYDPV TDLVYLGVGN GSPWNYKFRS EGKGDNLFLG SIV AINPDT GKYVWHFQET PMDEWDYTSV QQIMTLDMPV NGEMRHVIVH APKNGFFYII DAKTGKFITG KPYTYENWAN GLDP VTGRP NYVPDALWTL TGKPWLGIPG ELGGHNFAAM AYSPKTKLVY IPAQQIPLLY DGQKGGFKAY HDAWNLGLDM NKIGL FDDN DPEHVAAKKD FLKVLKGWTV AWDPEKMAPA FTINHKGPWN GGLLATAGNV IFQGLANGEF HAYDATNGND LYSFPA QSA IIAPPVTYTA NGKQYVAVEV GWGGIYPFLY GGVARTSGWT VNHSRVIAFS LDGKDSLPPK NELGFTPVKP VPTYDEA RQ KDGYFMYQTF CSACHGDNAI SGGVLPDLRW SGAPRGRESF YKLVGRGALT AYGMDRFDTS MTPEQIEDIR NFIVKRAN E SYDDEVKARE NSTGVPNDQF LNVPQSTADV PTADHP UniProtKB: Alcohol dehydrogenase (quinone), dehydrogenase subunit |
-分子 #2: Alcohol dehydrogenase (quinone), cytochrome c subunit
| 分子 | 名称: Alcohol dehydrogenase (quinone), cytochrome c subunit タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: alcohol dehydrogenase (quinone) |
|---|---|
| 由来(天然) | 生物種:  Gluconobacter oxydans 621H (バクテリア) / 株: 621H Gluconobacter oxydans 621H (バクテリア) / 株: 621H |
| 分子量 | 理論値: 51.249598 KDa |
| 組換発現 | 生物種:  Gluconobacter oxydans (バクテリア) Gluconobacter oxydans (バクテリア) |
| 配列 | 文字列: MLNALTRDRL VSEMKQGWKL AAAIGLMAVS FGAAHAQDAD EALIKRGEYV ARLSDCIACH TALHGQPYAG GLEIKSPIGT IYSTNITPD PEHGIGNYTL EDFTKALRKG IRKDGATVYP AMPYPEFARL SDDDIRAMYA FFMHGVKPVA LQNKAPDISW P LSMRWPLG ...文字列: MLNALTRDRL VSEMKQGWKL AAAIGLMAVS FGAAHAQDAD EALIKRGEYV ARLSDCIACH TALHGQPYAG GLEIKSPIGT IYSTNITPD PEHGIGNYTL EDFTKALRKG IRKDGATVYP AMPYPEFARL SDDDIRAMYA FFMHGVKPVA LQNKAPDISW P LSMRWPLG MWRAMFVPSM TPGVDKSISD PEVARGEYLV NGPGHCGECH TPRGFGMQVK AYGTAGGNAY LAGGAPIDNW IA PSLRSNS DTGLGRWSED DIVTFLKSGR IDHSAVFGGM ADVVAYSTQH WSDDDLRATA KYLKSMPAVP EGKNLGQDDG QTT ALLNKG GQGNAGAEVY LHNCAICHMN DGTGVNRMFP PLAGNPVVIT DDPTSLANVV AFGGILPPTN SAPSAVAMPG FKNH LSDQE MADVVNFMRK GWGNNAPGTV SASDIQKLRT TGAPVSTAGW NVSSKGWMAY MPQPYGEDWT FSPQTHTGVD DAQ UniProtKB: Alcohol dehydrogenase (quinone), cytochrome c subunit |
-分子 #3: Small subunit of alcohol dehydrogenase
| 分子 | 名称: Small subunit of alcohol dehydrogenase / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: alcohol dehydrogenase (quinone) |
|---|---|
| 由来(天然) | 生物種:  Gluconobacter oxydans (バクテリア) Gluconobacter oxydans (バクテリア) |
| 分子量 | 理論値: 14.282063 KDa |
| 組換発現 | 生物種:  Gluconobacter oxydans (バクテリア) Gluconobacter oxydans (バクテリア) |
| 配列 | 文字列: MFRRIVPVLG LALGLGLASQ AAMAQEQSPP PPPAVQGTPG KDFTGVSPAN LAGIMNYCVE QQYVSYDEGN PVLYGLSEKY KATEQTVGN FDYALGTAGY FDSNGKRFYL VAYTNEDDRR AACHAAVKAA QPML UniProtKB: Alcohol dehydrogenase, 15 kDa subunit |
-分子 #4: HEME C
| 分子 | 名称: HEME C / タイプ: ligand / ID: 4 / コピー数: 4 / 式: HEC |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEC: |
-分子 #5: PYRROLOQUINOLINE QUINONE
| 分子 | 名称: PYRROLOQUINOLINE QUINONE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: PQQ |
|---|---|
| 分子量 | 理論値: 330.206 Da |
| Chemical component information | 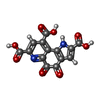 ChemComp-PQQ: |
-分子 #6: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #7: UBIQUINONE-10
| 分子 | 名称: UBIQUINONE-10 / タイプ: ligand / ID: 7 / コピー数: 1 / 式: U10 |
|---|---|
| 分子量 | 理論値: 863.343 Da |
| Chemical component information |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| アライメント法 | Coma free - Residual tilt: 0.01 mrad |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 3224 / 平均露光時間: 3.0 sec. / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.5 µm / 倍率(補正後): 56754 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8gy2: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)