+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the C-terminal domain of the human sodium-chloride cotransporter | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transporter / cation-chloride cotransporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Defective SLC12A3 causes Gitelman syndrome (GS) / sodium:chloride symporter activity / sodium:potassium:chloride symporter activity / Cation-coupled Chloride cotransporters / sodium ion homeostasis / chloride ion homeostasis / renal sodium ion absorption / response to salt / potassium ion homeostasis / response to aldosterone ...Defective SLC12A3 causes Gitelman syndrome (GS) / sodium:chloride symporter activity / sodium:potassium:chloride symporter activity / Cation-coupled Chloride cotransporters / sodium ion homeostasis / chloride ion homeostasis / renal sodium ion absorption / response to salt / potassium ion homeostasis / response to aldosterone / cell volume homeostasis / sodium ion transport / potassium ion import across plasma membrane / monoatomic ion transport / sodium ion transmembrane transport / chloride transmembrane transport / apical plasma membrane / extracellular exosome / ATP binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
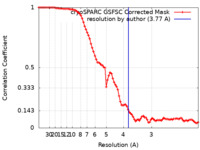
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.77 Å | |||||||||
 データ登録者 データ登録者 | Nan J / Yang XM / Shan ZY / Yuan YF / Zhang YQ | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Cryo-EM structure of the human sodium-chloride cotransporter NCC. 著者: Jing Nan / Yafei Yuan / Xuemei Yang / Ziyang Shan / Huihui Liu / Feiwen Wei / Wei Zhang / Yanqing Zhang /  要旨: The sodium-chloride cotransporter NCC mediates the coupled import of sodium and chloride across the plasma membrane, playing vital roles in kidney extracellular fluid volume and blood pressure ...The sodium-chloride cotransporter NCC mediates the coupled import of sodium and chloride across the plasma membrane, playing vital roles in kidney extracellular fluid volume and blood pressure control. Here, we present the full-length structure of human NCC, with 2.9 Å for the transmembrane domain and 3.8 Å for the carboxyl-terminal domain. NCC adopts an inward-open conformation and a domain-swap dimeric assembly. Conserved ion binding sites among the cation-chloride cotransporters and the Na2 site are observed in our structure. A unique His residue in the substrate pocket in NCC potentially interacts with Na1 and Cl1 and might also mediate the coordination of Na2 through a Ser residue. Putative observed water molecules are indicated to participate in the coordination of ions and TM coupling. Together with transport activity assays, our structure provides the first glimpse of NCC and defines ion binding sites, promoting drug development for hypertension targeting on NCC. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33804.map.gz emd_33804.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33804-v30.xml emd-33804-v30.xml emd-33804.xml emd-33804.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33804_fsc.xml emd_33804_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33804.png emd_33804.png | 74.3 KB | ||
| Filedesc metadata |  emd-33804.cif.gz emd-33804.cif.gz | 5.9 KB | ||
| その他 |  emd_33804_half_map_1.map.gz emd_33804_half_map_1.map.gz emd_33804_half_map_2.map.gz emd_33804_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33804 http://ftp.pdbj.org/pub/emdb/structures/EMD-33804 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33804 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33804 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33804_validation.pdf.gz emd_33804_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33804_full_validation.pdf.gz emd_33804_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_33804_validation.xml.gz emd_33804_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_33804_validation.cif.gz emd_33804_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33804 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33804 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33804 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33804 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33804.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33804.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.046 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33804_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33804_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sodium-chloride cotransporter
| 全体 | 名称: Sodium-chloride cotransporter |
|---|---|
| 要素 |
|
-超分子 #1: Sodium-chloride cotransporter
| 超分子 | 名称: Sodium-chloride cotransporter / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Solute carrier family 12 member 3
| 分子 | 名称: Solute carrier family 12 member 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 116.921461 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAELPTTETP GDATLCSGRF TISTLLSSDE PSPPAAYDSS HPSHLTHSST FCMRTFGYNT IDVVPTYEHY ANSTQPGEPR KVRPTLADL HSFLKEGRHL HALAFDSRPS HEMTDGLVEG EAGTSSEKNP EEPVRFGWVK GVMIRCMLNI WGVILYLRLP W ITAQAGIV ...文字列: MAELPTTETP GDATLCSGRF TISTLLSSDE PSPPAAYDSS HPSHLTHSST FCMRTFGYNT IDVVPTYEHY ANSTQPGEPR KVRPTLADL HSFLKEGRHL HALAFDSRPS HEMTDGLVEG EAGTSSEKNP EEPVRFGWVK GVMIRCMLNI WGVILYLRLP W ITAQAGIV LTWIIILLSV TVTSITGLSI SAISTNGKVK SGGTYFLISR SLGPELGGSI GLIFAFANAV GVAMHTVGFA ET VRDLLQE YGAPIVDPIN DIRIIGVVSV TVLLAISLAG MEWESKAQVL FFLVIMVSFA NYLVGTLIPP SEDKASKGFF SYR ADIFVQ NLVPDWRGPD GTFFGMFSIF FPSATGILAG ANISGDLKDP AIAIPKGTLM AIFWTTISYL AISATIGSCV VRDA SGVLN DTVTPGWGAC EGLACSYGWN FTECTQQHSC HYGLINYYQT MSMVSGFAPL ITAGIFGATL SSALACLVSA AKVFQ CLCE DQLYPLIGFF GKGYGKNKEP VRGYLLAYAI AVAFIIIAEL NTIAPIISNF FLCSYALINF SCFHASITNS PGWRPS FQY YNKWAALFGA IISVVIMFLL TWWAALIAIG VVLFLLLYVI YKKPEVNWGS SVQAGSYNLA LSYSVGLNEV EDHIKNY RP QCLVLTGPPN FRPALVDFVG TFTRNLSLMI CGHVLIGPHK QRMPELQLIA NGHTKWLNKR KIKAFYSDVI AEDLRRGV Q ILMQAAGLGR MKPNILVVGF KKNWQSAHPA TVEDYIGILH DAFDFNYGVC VMRMREGLNV SKMMQAHINP VFDPAEDGK EASARVDPKA LVKEEQATTI FQSEQGKKTI DIYWLFDDGG LTLLIPYLLG RKRRWSKCKI RVFVGGQINR MDQERKAIIS LLSKFRLGF HEVHILPDIN QNPRAEHTKR FEDMIAPFRL NDGFKDEATV NEMRRDCPWK ISDEEITKNR VKSLRQVRLN E IVLDYSRD AALIVITLPI GRKGKCPSSL YMAWLETLSQ DLRPPVILIR GNQENVLTFY CQLEGSDEVD AGSHHHHHHH HH HGSVEDY KDDDDK UniProtKB: Solute carrier family 12 member 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum ER / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 52.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)