[日本語] English
 万見
万見- EMDB-33217: Structure of human TRPV3_G573S in complex with Trpvicin in C4 symmetry -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human TRPV3_G573S in complex with Trpvicin in C4 symmetry | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | channel / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.64 Å | |||||||||
 データ登録者 データ登録者 | Fan J / Yue Z / Jiang D / Lei X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: Structural basis of TRPV3 inhibition by an antagonist. 著者: Junping Fan / Linghan Hu / Zongwei Yue / Daohong Liao / Fusheng Guo / Han Ke / Daohua Jiang / Yong Yang / Xiaoguang Lei /  要旨: The TRPV3 channel plays vital roles in skin physiology. Dysfunction of TRPV3 causes skin diseases, including Olmsted syndrome. However, the lack of potent and selective inhibitors impedes the ...The TRPV3 channel plays vital roles in skin physiology. Dysfunction of TRPV3 causes skin diseases, including Olmsted syndrome. However, the lack of potent and selective inhibitors impedes the validation of TRPV3 as a therapeutic target. In this study, we identified Trpvicin as a potent and subtype-selective inhibitor of TRPV3. Trpvicin exhibits pharmacological potential in the inhibition of itch and hair loss in mouse models. Cryogenic electron microscopy structures of TRPV3 and the pathogenic G573S mutant complexed with Trpvicin reveal detailed ligand-binding sites, suggesting that Trpvicin inhibits the TRPV3 channel by stabilizing it in a closed state. Our G573S mutant structures demonstrate that the mutation causes a dilated pore, generating constitutive opening activity. Trpvicin accesses additional binding sites inside the central cavity of the G573S mutant to remodel the channel symmetry and block the channel. Together, our results provide mechanistic insights into the inhibition of TRPV3 by Trpvicin and support TRPV3-related drug development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33217.map.gz emd_33217.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33217-v30.xml emd-33217-v30.xml emd-33217.xml emd-33217.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33217.png emd_33217.png | 86.3 KB | ||
| Filedesc metadata |  emd-33217.cif.gz emd-33217.cif.gz | 6.6 KB | ||
| その他 |  emd_33217_half_map_1.map.gz emd_33217_half_map_1.map.gz emd_33217_half_map_2.map.gz emd_33217_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33217 http://ftp.pdbj.org/pub/emdb/structures/EMD-33217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33217 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33217 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33217_validation.pdf.gz emd_33217_validation.pdf.gz | 952.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33217_full_validation.pdf.gz emd_33217_full_validation.pdf.gz | 952 KB | 表示 | |
| XML形式データ |  emd_33217_validation.xml.gz emd_33217_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_33217_validation.cif.gz emd_33217_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33217 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33217 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33217 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33217 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33217.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33217.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33217_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
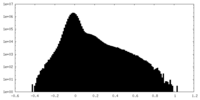
| 投影像・断面図 |
| ||||||||||||
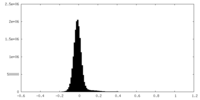
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33217_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
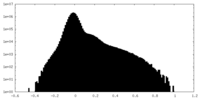
| 投影像・断面図 |
| ||||||||||||
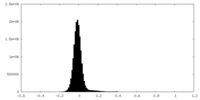
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TRPV3 in complex with an antagonist
| 全体 | 名称: TRPV3 in complex with an antagonist |
|---|---|
| 要素 |
|
-超分子 #1: TRPV3 in complex with an antagonist
| 超分子 | 名称: TRPV3 in complex with an antagonist / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 360 KDa |
-分子 #1: Fusion protein of Transient receptor potential cation channel sub...
| 分子 | 名称: Fusion protein of Transient receptor potential cation channel subfamily V member 3 and 3C-GFP タイプ: protein_or_peptide / ID: 1 詳細: The domain (792-799) is PreScission Site. The rest domain (800-1033) is corresponding to this sfGFP (2-235 amino acids). コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 120.865406 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKAHPKEMVP LMGKRVAAPS GNPAILPEKR PAEITPTKKS AHFFLEIEGF EPNPTVAKTS PPVFSKPMDS NIRQCISGNC DDMDSPQSP QDDVTETPSN PNSPSAQLAK EEQRRKKRRL KKRIFAAVSE GCVEELVELL VELQELCRRR HDEDVPDFLM H KLTASDTG ...文字列: MKAHPKEMVP LMGKRVAAPS GNPAILPEKR PAEITPTKKS AHFFLEIEGF EPNPTVAKTS PPVFSKPMDS NIRQCISGNC DDMDSPQSP QDDVTETPSN PNSPSAQLAK EEQRRKKRRL KKRIFAAVSE GCVEELVELL VELQELCRRR HDEDVPDFLM H KLTASDTG KTCLMKALLN INPNTKEIVR ILLAFAEEND ILGRFINAEY TEEAYEGQTA LNIAIERRQG DIAALLIAAG AD VNAHAKG AFFNPKYQHE GFYFGETPLA LAACTNQPEI VQLLMEHEQT DITSRDSRGN NILHALVTVA EDFKTQNDFV KRM YDMILL RSGNWELETT RNNDGLTPLQ LAAKMGKAEI LKYILSREIK EKRLRSLSRK FTDWAYGPVS SSLYDLTNVD TTTD NSVLE ITVYNTNIDN RHEMLTLEPL HTLLHMKWKK FAKHMFFLSF CFYFFYNITL TLVSYYRPRE EEAIPHPLAL THKMG WLQL LGRMFVLIWA MCISVKEGIA IFLLRPSDLQ SILSDAWFHF VFFIQAVLVI LSVFLYLFAY KEYLACLVLA MALGWA NML YYTRGFQSMS MYSVMIQKVI LHDVLKFLFV YIVFLLGFGV ALASLIEKCP KDNKDCSSYG SFSDAVLELF KLTIGLG DL NIQQNSKYPI LFLFLLITYV ILTFVLLLNM LIALMGETVE NVSKESERIW RLQRARTILE FEKMLPEWLR SRFRMGEL C KVAEDDFRLC LRINEVKWTE WKTHVSFLNE DPGPVRRTAD FNKIQDSSRN NSKTTLNAFE EVEEFPETSV LEVLFQGPS KGEELFTGVV PILVELDGDV NGHKFSVRGE GEGDATNGKL TLKFICTTGK LPVPWPTLVT TLTYGVQCFS RYPDHMKRHD FFKSAMPEG YVQERTISFK DDGTYKTRAE VKFEGDTLVN RIELKGIDFK EDGNILGHKL EYNFNSHNVY ITADKQKNGI K ANFKIRHN VEDGSVQLAD HYQQNTPIGD GPVLLPDNHY LSTQSVLSKD PNEKRDHMVL LEFVTAAGIT HGMDEWSHPQ FE KGGGSGG GSGGSAWSHP QFEK UniProtKB: Transient receptor potential cation channel subfamily V member 3 |
-分子 #2: N-[5-[2-(2-cyanopropan-2-yl)pyridin-4-yl]-4-(trifluoromethyl)-1,3...
| 分子 | 名称: N-[5-[2-(2-cyanopropan-2-yl)pyridin-4-yl]-4-(trifluoromethyl)-1,3-thiazol-2-yl]-4,6-dimethoxy-pyrimidine-5-carboxamide タイプ: ligand / ID: 2 / コピー数: 4 / 式: EQK |
|---|---|
| 分子量 | 理論値: 478.448 Da |
| Chemical component information | 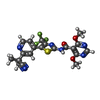 ChemComp-EQK: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)