+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of type IV-A NTS-nicked dsDNA bound Csf-crRNA ternary complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nuclease / STRUCTURAL PROTEIN-RNA-DNA COMPLEX | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Zhang JT / Cui N / Huang HD / Jia N | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Type IV-A CRISPR-Csf complex: Assembly, dsDNA targeting, and CasDinG recruitment. 著者: Ning Cui / Jun-Tao Zhang / Yongrui Liu / Yanhong Liu / Xiao-Yu Liu / Chongyuan Wang / Hongda Huang / Ning Jia /  要旨: Type IV CRISPR-Cas systems, which are primarily found on plasmids and exhibit a strong plasmid-targeting preference, are the only one of the six known CRISPR-Cas types for which the mechanistic ...Type IV CRISPR-Cas systems, which are primarily found on plasmids and exhibit a strong plasmid-targeting preference, are the only one of the six known CRISPR-Cas types for which the mechanistic details of their function remain unknown. Here, we provide high-resolution functional snapshots of type IV-A Csf complexes before and after target dsDNA binding, either in the absence or presence of CasDinG, revealing the mechanisms underlying Csf complex assembly, "DWN" PAM-dependent dsDNA targeting, R-loop formation, and CasDinG recruitment. Furthermore, we establish that CasDinG, a signature DinG family helicase, harbors ssDNA-stimulated ATPase activity and ATP-dependent 5'-3' DNA helicase activity. In addition, we show that CasDinG unwinds the non-target strand (NTS) and target strand (TS) of target dsDNA from the Csf complex. These molecular details advance our mechanistic understanding of type IV-A CRISPR-Csf function and should enable Csf complexes to be harnessed as genome-engineering tools for biotechnological applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33183.map.gz emd_33183.map.gz | 5.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33183-v30.xml emd-33183-v30.xml emd-33183.xml emd-33183.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33183.png emd_33183.png | 62.6 KB | ||
| マスクデータ |  emd_33183_msk_1.map emd_33183_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-33183.cif.gz emd-33183.cif.gz | 6.4 KB | ||
| その他 |  emd_33183_half_map_1.map.gz emd_33183_half_map_1.map.gz emd_33183_half_map_2.map.gz emd_33183_half_map_2.map.gz | 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33183 http://ftp.pdbj.org/pub/emdb/structures/EMD-33183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33183 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33183_validation.pdf.gz emd_33183_validation.pdf.gz | 760.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33183_full_validation.pdf.gz emd_33183_full_validation.pdf.gz | 760.2 KB | 表示 | |
| XML形式データ |  emd_33183_validation.xml.gz emd_33183_validation.xml.gz | 12.1 KB | 表示 | |
| CIF形式データ |  emd_33183_validation.cif.gz emd_33183_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33183 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33183 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33183.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33183.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
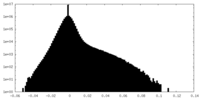
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33183_msk_1.map emd_33183_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
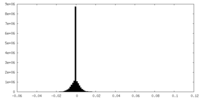
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33183_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
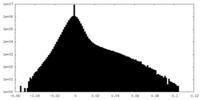
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33183_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
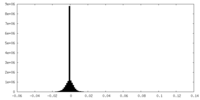
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Type IV-A NTS-nicked dsDNA bound Csf-crRNA ternary complex
+超分子 #1: Type IV-A NTS-nicked dsDNA bound Csf-crRNA ternary complex
+超分子 #2: Csf
+超分子 #3: crRNA
+超分子 #4: dsDNA
+分子 #1: Csf1
+分子 #2: Csf3
+分子 #3: Csf2
+分子 #4: Csf5
+分子 #5: crRNA
+分子 #6: NTS
+分子 #7: TS
+分子 #8: ZINC ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 136552 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: RANDOM ASSIGNMENT |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)