+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of RuvA-Holliday junction complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RuvA / Holliday junction / Homologous recombination / DNA damage repair / DNA BINDING PROTEIN-DNA COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Holliday junction helicase complex / Holliday junction resolvase complex / four-way junction helicase activity / SOS response / four-way junction DNA binding / DNA recombination / DNA repair / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas aeruginosa PAO1 (緑膿菌) / synthetic construct (人工物) Pseudomonas aeruginosa PAO1 (緑膿菌) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.01 Å | |||||||||
 データ登録者 データ登録者 | Lin Z / Qu Q / Zhang X / Zhou Z | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Front Plant Sci / 年: 2023 ジャーナル: Front Plant Sci / 年: 2023タイトル: Cryo-EM structure of the RuvAB-Holliday junction intermediate complex from . 著者: Xu Zhang / Zixuan Zhou / Lin Dai / Yulin Chao / Zheng Liu / Mingdong Huang / Qianhui Qu / Zhonghui Lin /  要旨: Holliday junction (HJ) is a four-way structured DNA intermediate in homologous recombination. In bacteria, the HJ-specific binding protein RuvA and the motor protein RuvB together form the RuvAB ...Holliday junction (HJ) is a four-way structured DNA intermediate in homologous recombination. In bacteria, the HJ-specific binding protein RuvA and the motor protein RuvB together form the RuvAB complex to catalyze HJ branch migration. (, Pa) is a ubiquitous opportunistic bacterial pathogen that can cause serious infection in a variety of host species, including vertebrate animals, insects and plants. Here, we describe the cryo-Electron Microscopy (cryo-EM) structure of the RuvAB-HJ intermediate complex from . The structure shows that two RuvA tetramers sandwich HJ at the junction center and disrupt base pairs at the branch points of RuvB-free HJ arms. Eight RuvB subunits are recruited by the RuvA octameric core and form two open-rings to encircle two opposite HJ arms. Each RuvB subunit individually binds a RuvA domain III. The four RuvB subunits within the ring display distinct subdomain conformations, and two of them engage the central DNA duplex at both strands with their C-terminal β-hairpins. Together with the biochemical analyses, our structure implicates a potential mechanism of RuvB motor assembly onto HJ DNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33013.map.gz emd_33013.map.gz | 217.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33013-v30.xml emd-33013-v30.xml emd-33013.xml emd-33013.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33013.png emd_33013.png | 12.8 KB | ||
| その他 |  emd_33013_half_map_1.map.gz emd_33013_half_map_1.map.gz emd_33013_half_map_2.map.gz emd_33013_half_map_2.map.gz | 391.1 MB 391.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33013 http://ftp.pdbj.org/pub/emdb/structures/EMD-33013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33013 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33013 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33013_validation.pdf.gz emd_33013_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33013_full_validation.pdf.gz emd_33013_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_33013_validation.xml.gz emd_33013_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_33013_validation.cif.gz emd_33013_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33013 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33013 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33013 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33013 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33013.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33013.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33013_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
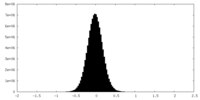
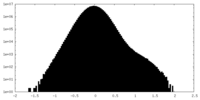
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33013_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
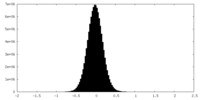
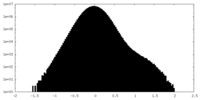
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RuvA-Holliday junction complex
| 全体 | 名称: RuvA-Holliday junction complex |
|---|---|
| 要素 |
|
-超分子 #1: RuvA-Holliday junction complex
| 超分子 | 名称: RuvA-Holliday junction complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Holliday junction
| 超分子 | 名称: Holliday junction / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 |
|---|
-超分子 #3: RuvA
| 超分子 | 名称: RuvA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
-分子 #1: DNA (26-MER)
| 分子 | 名称: DNA (26-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.999266 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DT)(DA)(DT)(DA)(DA)(DT) (DA)(DT)(DT)(DT)(DA)(DA)(DT)(DA)(DT)(DT) (DA)(DT)(DA)(DA)(DT)(DA) |
-分子 #2: DNA (26-MER)
| 分子 | 名称: DNA (26-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.972225 KDa |
| 配列 | 文字列: (DT)(DA)(DT)(DT)(DA)(DT)(DA)(DA)(DT)(DA) (DT)(DT)(DA)(DA)(DA)(DT)(DA)(DT)(DT)(DA) (DT)(DA)(DT)(DT)(DT)(DA) |
-分子 #3: DNA (26-MER)
| 分子 | 名称: DNA (26-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.981238 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DT)(DT)(DA)(DA)(DT)(DA) (DT)(DT)(DA)(DA)(DA)(DT)(DA)(DT)(DT)(DA) (DT)(DA)(DT)(DT)(DT)(DT) |
-分子 #4: DNA (26-MER)
| 分子 | 名称: DNA (26-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.972224 KDa |
| 配列 | 文字列: (DT)(DA)(DA)(DA)(DT)(DA)(DT)(DA)(DA)(DT) (DA)(DT)(DT)(DT)(DA)(DA)(DT)(DA)(DT)(DT) (DA)(DA)(DT)(DT)(DT)(DT) |
-分子 #5: Holliday junction ATP-dependent DNA helicase RuvA
| 分子 | 名称: Holliday junction ATP-dependent DNA helicase RuvA / タイプ: protein_or_peptide / ID: 5 / コピー数: 8 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) / 株: PAO1 Pseudomonas aeruginosa PAO1 (緑膿菌) / 株: PAO1 |
| 分子量 | 理論値: 15.432009 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIGRLRGTLA EKQPPHLILD VNGVGYEVEV PMTTLYRLPS VGEPVTLHTH LVVREDAHLL YGFAEKRERE LFRELIRLNG VGPKLALAL MSGLEVDELV RCVQAQDTST LVKIPGVGKK TAERLLVELK DRFKAWEN UniProtKB: Holliday junction branch migration complex subunit RuvA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 260.0 kPa |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 60241 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.01 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.1) / 使用した粒子像数: 143397 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.1) |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7x5a: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)