+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GK domain of Drosophila P5CS filament with glutamate, ATP, and NADPH | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Glutamate and glutamine metabolism / Mitochondrial protein degradation / glutamate-5-semialdehyde dehydrogenase / glutamate-5-semialdehyde dehydrogenase activity / glutamate 5-kinase / glutamate 5-kinase activity / L-proline biosynthetic process / proline biosynthetic process / mitochondrion / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Liu JL / Zhong J / Guo CJ / Zhou X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Structural basis of dynamic P5CS filaments. 著者: Jiale Zhong / Chen-Jun Guo / Xian Zhou / Chia-Chun Chang / Boqi Yin / Tianyi Zhang / Huan-Huan Hu / Guang-Ming Lu / Ji-Long Liu /  要旨: The bifunctional enzyme Δ-pyrroline-5-carboxylate synthase (P5CS) is vital to the synthesis of proline and ornithine, playing an essential role in human health and agriculture. Pathogenic mutations ...The bifunctional enzyme Δ-pyrroline-5-carboxylate synthase (P5CS) is vital to the synthesis of proline and ornithine, playing an essential role in human health and agriculture. Pathogenic mutations in the P5CS gene (ALDH18A1) lead to neurocutaneous syndrome and skin relaxation connective tissue disease in humans, and P5CS deficiency seriously damages the ability to resist adversity in plants. We have recently found that P5CS forms cytoophidia in vivo and filaments in vitro. However, it is difficult to appreciate the function of P5CS filamentation without precise structures. Using cryo-electron microscopy, here we solve the structures of full-length P5CS in three states at resolution from 3.1 to 4.3 Å. We observe distinct ligand-binding states and conformational changes for the GK and GPR domains, respectively. Divergent helical filaments are assembled by P5CS tetramers and stabilized by multiple interfaces. Point mutations disturbing those interfaces prevent P5CS filamentation and greatly reduce the enzymatic activity. Our findings reveal that filamentation is crucial for the coordination between the GK and GPR domains, providing a structural basis for the catalytic function of P5CS filaments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32875.map.gz emd_32875.map.gz | 62 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32875-v30.xml emd-32875-v30.xml emd-32875.xml emd-32875.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32875_fsc.xml emd_32875_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32875.png emd_32875.png | 92.5 KB | ||
| マスクデータ |  emd_32875_msk_1.map emd_32875_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_32875_half_map_1.map.gz emd_32875_half_map_1.map.gz emd_32875_half_map_2.map.gz emd_32875_half_map_2.map.gz | 78.4 MB 78.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32875 http://ftp.pdbj.org/pub/emdb/structures/EMD-32875 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32875 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32875 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32875_validation.pdf.gz emd_32875_validation.pdf.gz | 580.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32875_full_validation.pdf.gz emd_32875_full_validation.pdf.gz | 579.9 KB | 表示 | |
| XML形式データ |  emd_32875_validation.xml.gz emd_32875_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_32875_validation.cif.gz emd_32875_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32875 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32875 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32875 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32875 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7wx3MC  7f5tC  7f5uC  7f5vC  7f5xC  7wx4C  7wxfC  7wxgC  7wxhC  7wxiC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32875.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32875.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_32875_msk_1.map emd_32875_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
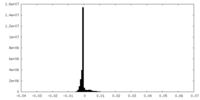
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_32875_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
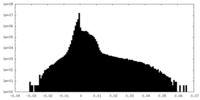
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_32875_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Drosophila glutamate-bound Delta-1-pyrroline-5-carboxylate synthase
| 全体 | 名称: Drosophila glutamate-bound Delta-1-pyrroline-5-carboxylate synthase |
|---|---|
| 要素 |
|
-超分子 #1: Drosophila glutamate-bound Delta-1-pyrroline-5-carboxylate synthase
| 超分子 | 名称: Drosophila glutamate-bound Delta-1-pyrroline-5-carboxylate synthase タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: Delta-1-pyrroline-5-carboxylate synthase
| 分子 | 名称: Delta-1-pyrroline-5-carboxylate synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: glutamate 5-kinase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 84.198227 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLQNSFKLAQ SLRNGFYRNA WRAFSSHGPR QPLVSPERRL EKAHPTFTER SQLKYARRLV VKLGSAVITR EDNHGLALGR LASIVEQVA ECHLEGREVM MVTSGAVAFG KQKLAQELLM SLSMRETLNP KDSKEFDGAT LEPRAAAAVG QSGLMSLYDA M FAQYGVKI ...文字列: MLQNSFKLAQ SLRNGFYRNA WRAFSSHGPR QPLVSPERRL EKAHPTFTER SQLKYARRLV VKLGSAVITR EDNHGLALGR LASIVEQVA ECHLEGREVM MVTSGAVAFG KQKLAQELLM SLSMRETLNP KDSKEFDGAT LEPRAAAAVG QSGLMSLYDA M FAQYGVKI AQVLVTKPDF YNEETRNNLF CTLSELISLN IVPIINTNDA VSPPMFIRDD EPAGGARRGI PIKDNDSLSA ML AAEVQAD LLILMSDVDG IYNKPPWEDG AKLMHTYTSD DSNSIEFGKK SKVGTGGMDS KVKAATWALD RGVSVVICNG MQE KAIKTI IGGRKVGTFF TEATESANAV PVEVMAENAR TGSRQMQALT PAQRASAVNT LADLLVSREK FILDANAKDL AEAQ KSGLA KPLLSRLSLN PAKLKNLSVG LKQIAEDSHK NVGRVLRRTR LADQLELKQV TVPIGVLLVI FESRPDSLPQ VAALA MASA NGLLLKGGKE AAHSNKALME LVKEALATVG AEHAVSLVST REEISDLLSM ENHIDLIIPR GSSDLVRSIQ QQSLHI PVL GHAEGVCHVY IDRDADLEKA LRIARDAKCD YPAACNAMET LLIHEDLMSG AIFGDVCNML KREGVKIYAG PRLNQQL TF GPPAAKSLKH EYGALECCIE VVPSLDEAIN HIHTYGSSHT DVIVTENDAA ARQFLGSVDS ACVFHNASSR FADGFRFG L GAEVGISTAR IHARGPVGVE GLLTTKWILE GQDHAAADFA EGGGRTWLHE TLPLD |
-分子 #2: GAMMA-GLUTAMYL PHOSPHATE
| 分子 | 名称: GAMMA-GLUTAMYL PHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: RGP |
|---|---|
| 分子量 | 理論値: 227.109 Da |
| Chemical component information |  ChemComp-RGP: |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / 平均電子線量: 72.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)