+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3287 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Evidence for a conformational switch in Influenzavirus M1 and its role in filamentous virion architecture | |||||||||
 マップデータ マップデータ | Reconstruction of trailing tips from influenza A virions. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza A virus / FLUAV / polymerase / M1 / crown / leading tip / trailing tip / filamentous | |||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) | |||||||||
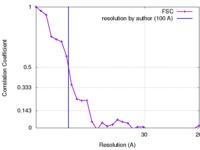
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 100.0 Å | |||||||||
 データ登録者 データ登録者 | Kiss G / Abdulsattar BO / Phapugrangkul P / Birch K / Jones IM / Neuman BW | |||||||||
 引用 引用 | ジャーナル: Proc Natl Acad Sci U S A / 年: 2010 タイトル: Structural organization of a filamentous influenza A virus. 著者: Lesley J Calder / Sebastian Wasilewski / John A Berriman / Peter B Rosenthal /  要旨: Influenza is a lipid-enveloped, pleomorphic virus. We combine electron cryotomography and analysis of images of frozen-hydrated virions to determine the structural organization of filamentous ...Influenza is a lipid-enveloped, pleomorphic virus. We combine electron cryotomography and analysis of images of frozen-hydrated virions to determine the structural organization of filamentous influenza A virus. Influenza A/Udorn/72 virions are capsule-shaped or filamentous particles of highly uniform diameter. We show that the matrix layer adjacent to the membrane is an ordered helix of the M1 protein and its close interaction with the surrounding envelope determines virion morphology. The ribonucleoprotein particles (RNPs) that package the genome segments form a tapered assembly at one end of the virus interior. The neuraminidase, which is present in smaller numbers than the hemagglutinin, clusters in patches and are typically present at the end of the virion opposite to RNP attachment. Incubation of virus at low pH causes a loss of filamentous morphology, during which we observe a structural transition of the matrix layer from its helical, membrane-associated form to a multilayered coil structure inside the virus particle. The polar organization of the virus provides a model for assembly of the virion during budding at the host membrane. Images and tomograms of A/Aichi/68 X-31 virions show the generality of these conclusions to non-filamentous virions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3287.map.gz emd_3287.map.gz | 854 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3287-v30.xml emd-3287-v30.xml emd-3287.xml emd-3287.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3287_fsc.xml emd_3287_fsc.xml | 2.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3287.png emd_3287.png | 74.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3287 http://ftp.pdbj.org/pub/emdb/structures/EMD-3287 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3287 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3287 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3287_validation.pdf.gz emd_3287_validation.pdf.gz | 222.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3287_full_validation.pdf.gz emd_3287_full_validation.pdf.gz | 221.5 KB | 表示 | |
| XML形式データ |  emd_3287_validation.xml.gz emd_3287_validation.xml.gz | 5.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3287 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3287 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3287 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3287 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3287.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3287.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of trailing tips from influenza A virions. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 10 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Trailing tips of filamentous influenza A virions, showing M1 and ...
| 全体 | 名称: Trailing tips of filamentous influenza A virions, showing M1 and parts of the viral ribonucleoprotein |
|---|---|
| 要素 |
|
-超分子 #1000: Trailing tips of filamentous influenza A virions, showing M1 and ...
| 超分子 | 名称: Trailing tips of filamentous influenza A virions, showing M1 and parts of the viral ribonucleoprotein タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Influenza A virus
| 超分子 | 名称: Influenza A virus / タイプ: virus / ID: 1 / Name.synonym: Influenza virus 詳細: Reconstruction of the inner layer of the viral envelope from trailing tips of native virions of mixed A/Aichi/X31 and A/Udorn strains. Map is Gaussian filtered to 7.6 nm the 0.5 FSC. Reconstructed in EMAN2. NCBI-ID: 11320 / 生物種: Influenza A virus / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: Influenza virus |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 株: Combined A/Aichi/2/68 and A/Udorn/307/72 / 別称: VERTEBRATES Homo sapiens (ヒト) / 株: Combined A/Aichi/2/68 and A/Udorn/307/72 / 別称: VERTEBRATES |
| Host system | 生物種:  Homo sapiens (ヒト) / 組換細胞: MDCK Homo sapiens (ヒト) / 組換細胞: MDCK |
| ウイルス殻 | Shell ID: 1 / 名称: M1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: FEI VITROBOT MARK III 詳細: Details for EM and reconstruction published in Calder et al., Proc Natl Acad Sci U S A. 2010 Jun 8; 107(23): 10685 to 10690. 手法: Blotted 4s before plunging |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | OTHER |
|---|---|
| 日付 | 2010年1月1日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI EAGLE (2k x 2k) / 平均電子線量: 50 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)