+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human glucose transporter GLUT4 bound to cytochalasin B in lipid nanodiscs | |||||||||
 マップデータ マップデータ | Cryo-EM structure of human glucose transporter GLUT4 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | glucose transporter / GLUT4 / diabetes / cytochalasin B / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報D-glucose uniporter activity / regulation of synaptic vesicle budding from presynaptic endocytic zone membrane / positive regulation of brain-derived neurotrophic factor receptor signaling pathway / white fat cell proliferation / amylopectin biosynthetic process / dehydroascorbic acid transport / glucose import in response to insulin stimulus / Cellular hexose transport / D-glucose transmembrane transporter activity / insulin-responsive compartment ...D-glucose uniporter activity / regulation of synaptic vesicle budding from presynaptic endocytic zone membrane / positive regulation of brain-derived neurotrophic factor receptor signaling pathway / white fat cell proliferation / amylopectin biosynthetic process / dehydroascorbic acid transport / glucose import in response to insulin stimulus / Cellular hexose transport / D-glucose transmembrane transporter activity / insulin-responsive compartment / D-glucose transmembrane transport / trans-Golgi network transport vesicle / short-term memory / cellular response to osmotic stress / vesicle membrane / clathrin-coated vesicle / D-glucose import / long-term memory / brown fat cell differentiation / transport across blood-brain barrier / clathrin-coated pit / multivesicular body / T-tubule / endomembrane system / cytoplasmic vesicle membrane / sarcoplasmic reticulum / Translocation of SLC2A4 (GLUT4) to the plasma membrane / trans-Golgi network / sarcolemma / Transcriptional regulation of white adipocyte differentiation / cellular response to insulin stimulus / cellular response to tumor necrosis factor / glucose homeostasis / presynapse / response to ethanol / cellular response to hypoxia / carbohydrate metabolic process / learning or memory / membrane raft / external side of plasma membrane / perinuclear region of cytoplasm / extracellular exosome / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.25 Å | |||||||||
 データ登録者 データ登録者 | Yuan Y / Kong F | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Cryo-EM structure of human glucose transporter GLUT4. 著者: Yafei Yuan / Fang Kong / Hanwen Xu / Angqi Zhu / Nieng Yan / Chuangye Yan /   要旨: GLUT4 is the primary glucose transporter in adipose and skeletal muscle tissues. Its cellular trafficking is regulated by insulin signaling. Failed or reduced plasma membrane localization of GLUT4 is ...GLUT4 is the primary glucose transporter in adipose and skeletal muscle tissues. Its cellular trafficking is regulated by insulin signaling. Failed or reduced plasma membrane localization of GLUT4 is associated with diabetes. Here, we report the cryo-EM structures of human GLUT4 bound to a small molecule inhibitor cytochalasin B (CCB) at resolutions of 3.3 Å in both detergent micelles and lipid nanodiscs. CCB-bound GLUT4 exhibits an inward-open conformation. Despite the nearly identical conformation of the transmembrane domain to GLUT1, the cryo-EM structure reveals an extracellular glycosylation site and an intracellular helix that is invisible in the crystal structure of GLUT1. The structural study presented here lays the foundation for further mechanistic investigation of the modulation of GLUT4 trafficking. Our methods for cryo-EM analysis of GLUT4 will also facilitate structural determination of many other small size solute carriers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32760.map.gz emd_32760.map.gz | 25.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32760-v30.xml emd-32760-v30.xml emd-32760.xml emd-32760.xml | 13.5 KB 13.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32760.png emd_32760.png | 146.9 KB | ||
| Filedesc metadata |  emd-32760.cif.gz emd-32760.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32760 http://ftp.pdbj.org/pub/emdb/structures/EMD-32760 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32760 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32760 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32760_validation.pdf.gz emd_32760_validation.pdf.gz | 496.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32760_full_validation.pdf.gz emd_32760_full_validation.pdf.gz | 495.7 KB | 表示 | |
| XML形式データ |  emd_32760_validation.xml.gz emd_32760_validation.xml.gz | 5.8 KB | 表示 | |
| CIF形式データ |  emd_32760_validation.cif.gz emd_32760_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32760 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32760 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32760 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32760 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7wsmMC  7wsnC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32760.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32760.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of human glucose transporter GLUT4 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0825 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : GLUT4
| 全体 | 名称: GLUT4 |
|---|---|
| 要素 |
|
-超分子 #1: GLUT4
| 超分子 | 名称: GLUT4 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 56 kDa/nm |
-分子 #1: Solute carrier family 2, facilitated glucose transporter member 4
| 分子 | 名称: Solute carrier family 2, facilitated glucose transporter member 4 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 56.108199 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKG TMPSGFQQIG SEDGEPPQQR VTGTLVLAVF SAVLGSLQFG YNIGVINAPQ KVIEQSYNET WLGRQGPEGP SSIPPGTLT TLWALSVAIF SVGGMISSFL IGIISQWLGR KRAMLVNNVL AVLGGSLMGL ANAAASYEML ILGRFLIGAY S GLTSGLVP ...文字列: MDYKDDDDKG TMPSGFQQIG SEDGEPPQQR VTGTLVLAVF SAVLGSLQFG YNIGVINAPQ KVIEQSYNET WLGRQGPEGP SSIPPGTLT TLWALSVAIF SVGGMISSFL IGIISQWLGR KRAMLVNNVL AVLGGSLMGL ANAAASYEML ILGRFLIGAY S GLTSGLVP MYVGEIAPTH LRGALGTLNQ LAIVIGILIA QVLGLESLLG TASLWPLLLG LTVLPALLQL VLLPFCPESP RY LYIIQNL EGPARKSLKR LTGWADVSGV LAELKDEKRK LERERPLSLL QLLGSRTHRQ PLIIAVVLQL SQQLSGINAV FYY STSIFE TAGVGQPAYA TIGAGVVNTV FTLVSVLLVE RAGRRTLHLL GLAGMCGCAI LMTVALLLLE RVPAMSYVSI VAIF GFVAF FEIGPGPIPW FIVAELFSQG PRPAAMAVAG FSNWTSNFII GMGFQYVAEA MGPYVFLLFA VLLLGFFIFT FLRVP ETRG RTFDQISAAF HRTPSLLEQE VKPSTELEYL GPDEND UniProtKB: Solute carrier family 2, facilitated glucose transporter member 4 |
-分子 #3: Cytochalasin B
| 分子 | 名称: Cytochalasin B / タイプ: ligand / ID: 3 / コピー数: 1 / 式: 5RH |
|---|---|
| 分子量 | 理論値: 479.608 Da |
| Chemical component information | 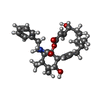 ChemComp-5RH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















