+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3179 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of yeast apo RNA polymerase III at 4.6 A | |||||||||
 マップデータ マップデータ | Reconstruction of apo RNA polymerase III in the closed clamp conformation | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA polymerase III / pol III / transcription / RNA polymerase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / mRNA Capping / RNA polymerase II transcribes snRNA genes / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening ...RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / mRNA Capping / RNA polymerase II transcribes snRNA genes / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / termination of RNA polymerase III transcription / RNA Polymerase II Pre-transcription Events / RNA-templated transcription / Formation of TC-NER Pre-Incision Complex / transcription initiation at RNA polymerase III promoter / RNA Polymerase I Promoter Escape / termination of RNA polymerase I transcription / nucleolar large rRNA transcription by RNA polymerase I / Gap-filling DNA repair synthesis and ligation in TC-NER / transcription initiation at RNA polymerase I promoter / Estrogen-dependent gene expression / transcription by RNA polymerase III / Dual incision in TC-NER / RNA polymerase I complex / transcription elongation by RNA polymerase I / RNA polymerase III complex / RNA polymerase II, core complex / tRNA transcription by RNA polymerase III / transcription by RNA polymerase I / nucleotidyltransferase activity / transcription initiation at RNA polymerase II promoter / transcription elongation by RNA polymerase II / ribonucleoside binding / DNA-directed RNA polymerase / DNA-directed RNA polymerase activity / peroxisome / ribosome biogenesis / single-stranded DNA binding / nucleic acid binding / transcription by RNA polymerase II / protein dimerization activity / nucleotide binding / nucleolus / mitochondrion / DNA binding / zinc ion binding / nucleoplasm / metal ion binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
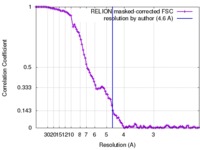
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Hoffmann NA / Jakobi AJ / Moreno-Morcillo M / Glatt S / Kosinski J / Hagen WJ / Sachse C / Muller CW | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2015 ジャーナル: Nature / 年: 2015タイトル: Molecular structures of unbound and transcribing RNA polymerase III. 著者: Niklas A Hoffmann / Arjen J Jakobi / María Moreno-Morcillo / Sebastian Glatt / Jan Kosinski / Wim J H Hagen / Carsten Sachse / Christoph W Müller /  要旨: Transcription of genes encoding small structured RNAs such as transfer RNAs, spliceosomal U6 small nuclear RNA and ribosomal 5S RNA is carried out by RNA polymerase III (Pol III), the largest yet ...Transcription of genes encoding small structured RNAs such as transfer RNAs, spliceosomal U6 small nuclear RNA and ribosomal 5S RNA is carried out by RNA polymerase III (Pol III), the largest yet structurally least characterized eukaryotic RNA polymerase. Here we present the cryo-electron microscopy structures of the Saccharomyces cerevisiae Pol III elongating complex at 3.9 Å resolution and the apo Pol III enzyme in two different conformations at 4.6 and 4.7 Å resolution, respectively, which allow the building of a 17-subunit atomic model of Pol III. The reconstructions reveal the precise orientation of the C82-C34-C31 heterotrimer in close proximity to the stalk. The C53-C37 heterodimer positions residues involved in transcription termination close to the non-template DNA strand. In the apo Pol III structures, the stalk adopts different orientations coupled with closed and open conformations of the clamp. Our results provide novel insights into Pol III-specific transcription and the adaptation of Pol III towards its small transcriptional targets. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3179.map.gz emd_3179.map.gz | 11.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3179-v30.xml emd-3179-v30.xml emd-3179.xml emd-3179.xml | 9 KB 9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3179_fsc.xml emd_3179_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3179.jpg emd_3179.jpg | 66.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3179 http://ftp.pdbj.org/pub/emdb/structures/EMD-3179 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3179 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3179 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3179_validation.pdf.gz emd_3179_validation.pdf.gz | 256 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3179_full_validation.pdf.gz emd_3179_full_validation.pdf.gz | 255.1 KB | 表示 | |
| XML形式データ |  emd_3179_validation.xml.gz emd_3179_validation.xml.gz | 11.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3179 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3179 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3179 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3179 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3179.map.gz / 形式: CCP4 / 大きさ: 96.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3179.map.gz / 形式: CCP4 / 大きさ: 96.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of apo RNA polymerase III in the closed clamp conformation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.084 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : S.cerevisiae apo RNA polymerase III (closed clamp conformation)
| 全体 | 名称: S.cerevisiae apo RNA polymerase III (closed clamp conformation) |
|---|---|
| 要素 |
|
-超分子 #1000: S.cerevisiae apo RNA polymerase III (closed clamp conformation)
| 超分子 | 名称: S.cerevisiae apo RNA polymerase III (closed clamp conformation) タイプ: sample / ID: 1000 / 集合状態: Monomer / Number unique components: 17 |
|---|---|
| 分子量 | 理論値: 690 KDa |
-分子 #1: RNA polymerase III
| 分子 | 名称: RNA polymerase III / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 集合状態: Monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 690 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 150mM (NH4)2SO4, 15mM Tris, 10mM DTT |
| グリッド | 詳細: Mo 400 mesh Quantifoil 1.2/1.3 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK III 手法: 2.5 ul of sample was applied 15 seconds wait time Blot for 9 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 日付 | 2014年12月15日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 2398 / 平均電子線量: 42 e/Å2 詳細: Seven frames were recorded with a dose of 6 e/A2 per frame. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.2 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)