+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31296 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
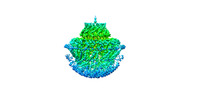
| タイトル | Human Cytomegalovirus, C12 portal | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | C12 portal / partially-enveloped capsid / Human Cytomegalovirus / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Herpesvirus portal protein / Herpesvirus UL6 like / chromosome organization / virion component / host cell nucleus / Capsid portal protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Human cytomegalovirus (ヘルペスウイルス) / Human cytomegalovirus (ヘルペスウイルス) /   Human betaherpesvirus 5 (ヘルペスウイルス) Human betaherpesvirus 5 (ヘルペスウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Li Z / Yu X / Dong L | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural basis for genome packaging, retention, and ejection in human cytomegalovirus. 著者: Zhihai Li / Jingjing Pang / Lili Dong / Xuekui Yu /  要旨: How the human cytomegalovirus (HCMV) genome-the largest among human herpesviruses-is packaged, retained, and ejected remains unclear. We present the in situ structures of the symmetry-mismatched ...How the human cytomegalovirus (HCMV) genome-the largest among human herpesviruses-is packaged, retained, and ejected remains unclear. We present the in situ structures of the symmetry-mismatched portal and the capsid vertex-specific components (CVSCs) of HCMV. The 5-fold symmetric 10-helix anchor-uncommon among known portals-contacts the portal-encircling DNA, which is presumed to squeeze the portal as the genome packaging proceeds. We surmise that the 10-helix anchor dampens this action to delay the portal reaching a "head-full" packaging state, thus facilitating the large genome to be packaged. The 6-fold symmetric turret, latched via a coiled coil to a helix from a major capsid protein, supports the portal to retain the packaged genome. CVSCs at the penton vertices-presumed to increase inner capsid pressure-display a low stoichiometry, which would aid genome retention. We also demonstrate that the portal and capsid undergo conformational changes to facilitate genome ejection after viral cell entry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31296.map.gz emd_31296.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31296-v30.xml emd-31296-v30.xml emd-31296.xml emd-31296.xml | 9.1 KB 9.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31296.png emd_31296.png | 28.6 KB | ||
| Filedesc metadata |  emd-31296.cif.gz emd-31296.cif.gz | 5.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31296 http://ftp.pdbj.org/pub/emdb/structures/EMD-31296 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31296 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31296 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31296_validation.pdf.gz emd_31296_validation.pdf.gz | 594.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31296_full_validation.pdf.gz emd_31296_full_validation.pdf.gz | 594.2 KB | 表示 | |
| XML形式データ |  emd_31296_validation.xml.gz emd_31296_validation.xml.gz | 5 KB | 表示 | |
| CIF形式データ |  emd_31296_validation.cif.gz emd_31296_validation.cif.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31296 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31296 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31296 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31296 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31296.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31296.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.625 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human betaherpesvirus 5
| 全体 | 名称:   Human betaherpesvirus 5 (ヘルペスウイルス) Human betaherpesvirus 5 (ヘルペスウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human betaherpesvirus 5
| 超分子 | 名称: Human betaherpesvirus 5 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10359 / 生物種: Human betaherpesvirus 5 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human cytomegalovirus (ヘルペスウイルス) Human cytomegalovirus (ヘルペスウイルス) |
| 分子量 | 理論値: 78.634805 KDa |
| 配列 | 文字列: MERNHWNEKS SGAKRSRERD LTLSTIRSIL AADERLRIKA SSYLGVGRGV DDEAVIDIFP TGQTMSFLRL LHGFLGTCRG QSMHQVLRD PCVLRKQLLY GVCKTLFDTI TVRRVAEEWK LHAALFPYRA LDEEDLEQYL LVWSASLRQS VQTGVLGALR D ILYQYADN ...文字列: MERNHWNEKS SGAKRSRERD LTLSTIRSIL AADERLRIKA SSYLGVGRGV DDEAVIDIFP TGQTMSFLRL LHGFLGTCRG QSMHQVLRD PCVLRKQLLY GVCKTLFDTI TVRRVAEEWK LHAALFPYRA LDEEDLEQYL LVWSASLRQS VQTGVLGALR D ILYQYADN DDYGLYVDWC VTVGLVPLLD VKTKPSEAAE RAQFVRAAVQ RATETHPLAQ DLLQANLALL LQVAERLGAV RV ANAPEVR VFKKVRSERL EAQLRGKHIR LYVAAEPLAY ERDKLLFTTP VAHLHEEILR YDGLCRHQKI CQLLNTFPVK VVT ASRHEL NCKKLVEMME QHDRGSDAKK SIMKFLLNVS DSKSRIGIED SVESFLQDLT PSLVDQNRLL PARGPGGPGV VGPG GAVVG GPAGHVGLLP PPPGPAAPER DIRDLFKKQV IKCLEEQIQS QVDEIQDLRT LNQTWENRVR ELRDLLTRYA SRRED SMSL GARDAELYHL PVLEAVRKAR DAAPFRPLAV EDNRLVANSF FSQFVPGTES LERFLTQLWE NEYFRTFRLR RLVTHQ GAE EAIVYSNYTV ERVTLPYLCH ILALGTLDPV PEAYLQLSFG EIVAAAYDDS KFCRYVELIC SREKARRRQM SREAAGG VP ERGTASSGGP GTLERSAPRR LITADEERRG PERVGRFRNG GPDDPRRAGG PYGFH UniProtKB: Capsid portal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 40903 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















