+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31078 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cyanophage Pam1 capsid asymmetric unit | |||||||||
 マップデータ マップデータ | An asymmetric unit of Pam1 capsid.Cutting out using phenix Map box program. | |||||||||
 試料 試料 | Pam1 != unidentified Pam1
| |||||||||
 キーワード キーワード | Major capsid and cement proteins / VIRUS | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | |||||||||
 データ登録者 データ登録者 | Zhang JT / Jiang YL | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Structure and assembly pattern of a freshwater short-tailed cyanophage Pam1. 著者: Jun-Tao Zhang / Feng Yang / Kang Du / Wei-Fang Li / Yuxing Chen / Yong-Liang Jiang / Qiong Li / Cong-Zhao Zhou /  要旨: Despite previous structural analyses of bacteriophages, quite little is known about the structures and assembly patterns of cyanophages. Using cryo-EM combined with crystallography, we solve the near- ...Despite previous structural analyses of bacteriophages, quite little is known about the structures and assembly patterns of cyanophages. Using cryo-EM combined with crystallography, we solve the near-atomic-resolution structure of a freshwater short-tailed cyanophage, Pam1, which comprises a 400-Å-long tail and an icosahedral capsid of 650 Å in diameter. The outer capsid surface is reinforced by trimeric cement proteins with a β-sandwich fold, which structurally resemble the distal motif of Pam1's tailspike, suggesting its potential role in host recognition. At the portal vertex, the dodecameric portal and connected adaptor, followed by a hexameric needle head, form a DNA ejection channel, which is sealed by a trimeric needle. Moreover, we identify a right-handed rifling pattern that might help DNA to revolve along the wall of the ejection channel. Our study reveals the precise assembly pattern of a cyanophage and lays the foundation to support its practical biotechnological and environmental applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31078.map.gz emd_31078.map.gz | 2.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31078-v30.xml emd-31078-v30.xml emd-31078.xml emd-31078.xml | 10.2 KB 10.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31078.png emd_31078.png | 160.7 KB | ||
| Filedesc metadata |  emd-31078.cif.gz emd-31078.cif.gz | 5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31078 http://ftp.pdbj.org/pub/emdb/structures/EMD-31078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31078 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31078_validation.pdf.gz emd_31078_validation.pdf.gz | 441.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31078_full_validation.pdf.gz emd_31078_full_validation.pdf.gz | 441.1 KB | 表示 | |
| XML形式データ |  emd_31078_validation.xml.gz emd_31078_validation.xml.gz | 4.3 KB | 表示 | |
| CIF形式データ |  emd_31078_validation.cif.gz emd_31078_validation.cif.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31078 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31078 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31078.map.gz / 形式: CCP4 / 大きさ: 23.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31078.map.gz / 形式: CCP4 / 大きさ: 23.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | An asymmetric unit of Pam1 capsid.Cutting out using phenix Map box program. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.013 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
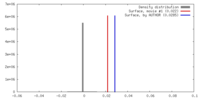
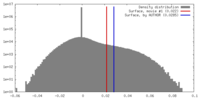
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pam1
| 全体 | 名称: Pam1 |
|---|---|
| 要素 |
|
-超分子 #1: unidentified
| 超分子 | 名称: unidentified / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 32644 / 生物種: unidentified / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Pseudanabaena mucicola (バクテリア) Pseudanabaena mucicola (バクテリア) |
-分子 #1: Major capsid proteins
| 分子 | 名称: Major capsid proteins / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 39.379066 KDa |
| 配列 | 文字列: MADFSLATAS QRKEWSNKAH MEYVRRSRFA PYIRNTENSI FQGYSDLEKR AGDTLNIPLF YKLGGAPVTG DTPIVGNETP LDNYNCGVP VALRGKGVAI TKNQTFRTEI DVMNAAKQSL TRYFGELLRD DIIEALGSVV TTGDTTVNYG SASAANRNAF S AANPDRLF ...文字列: MADFSLATAS QRKEWSNKAH MEYVRRSRFA PYIRNTENSI FQGYSDLEKR AGDTLNIPLF YKLGGAPVTG DTPIVGNETP LDNYNCGVP VALRGKGVAI TKNQTFRTEI DVMNAAKQSL TRYFGELLRD DIIEALGSVV TTGDTTVNYG SASAANRNAF S AANPDRLF FGSISGYSAT WATGLGNVDA AETCTAARVG VMKRLAMSAS PAITPMQVDD DEGREYFVAF HGSRTFRDLK GD TAMLNAN REARPRDVSS NPLLQDGDLI YEGVIHREVP EIDAWAAANG FNTAGAGSAP IRPVFLCGTQ SVFLAYAQRP QAG TEKSDI PALNRRMTVG MDEIIGVKKA AFNGKQHGVV MGFFGAAGD |
-分子 #2: Cement (decoration) proteins
| 分子 | 名称: Cement (decoration) proteins / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 13.831203 KDa |
| 配列 | 文字列: MPATNSAQAR LAAPGHGFGG NVKVSYGSVA FTGTITTADA ATVCNLPVGA IVLGVTLESD DLDTNATPTI TLNVGDAGSA TRYFSASTV AQAGTSSSAP ATTGLLWTVT EGNTAVRIAV ANNAATSADG SVRVAVTYYL P |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.26 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 21210 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: RANDOM ASSIGNMENT |
 ムービー
ムービー コントローラー
コントローラー
















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)