+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30988 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Enolase from Mycobacterium tuberculosis | |||||||||
 マップデータ マップデータ | This is the combined final map with B-factor sharpening | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | METAL BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 ホスホピルビン酸ヒドラターゼ / ホスホピルビン酸ヒドラターゼ /  phosphopyruvate hydratase complex / phosphopyruvate hydratase complex /  phosphopyruvate hydratase activity / glycolytic process / magnesium ion binding / phosphopyruvate hydratase activity / glycolytic process / magnesium ion binding /  細胞膜 / extracellular region 細胞膜 / extracellular region類似検索 - 分子機能 | |||||||||
| 生物種 |   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.08 Å クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||
 データ登録者 データ登録者 | Bose S / Vinothkumar KR | |||||||||
| 資金援助 |  インド, 2件 インド, 2件
| |||||||||
 引用 引用 |  ジャーナル: IUCrJ / 年: 2023 ジャーナル: IUCrJ / 年: 2023タイトル: Structural snapshots of Mycobacterium tuberculosis enolase reveal dual mode of 2PG binding and its implication in enzyme catalysis. 著者: Mohammed Ahmad / Bhavya Jha / Sucharita Bose / Satish Tiwari / Abhisek Dwivedy / Deepshikha Kar / Ravikant Pal / Richard Mariadasse / Tanya Parish / Jeyaraman Jeyakanthan / Kutti R ...著者: Mohammed Ahmad / Bhavya Jha / Sucharita Bose / Satish Tiwari / Abhisek Dwivedy / Deepshikha Kar / Ravikant Pal / Richard Mariadasse / Tanya Parish / Jeyaraman Jeyakanthan / Kutti R Vinothkumar / Bichitra Kumar Biswal /   要旨: Enolase, a ubiquitous enzyme, catalyzes the reversible conversion of 2-phosphoglycerate (2PG) to phosphoenolpyruvate (PEP) in the glycolytic pathway of organisms of all three domains of life. The ...Enolase, a ubiquitous enzyme, catalyzes the reversible conversion of 2-phosphoglycerate (2PG) to phosphoenolpyruvate (PEP) in the glycolytic pathway of organisms of all three domains of life. The underlying mechanism of the 2PG to PEP conversion has been studied in great detail in previous work, however that of the reverse reaction remains to be explored. Here we present structural snapshots of Mycobacterium tuberculosis (Mtb) enolase in apo, PEP-bound and two 2PG-bound forms as it catalyzes the conversion of PEP to 2PG. The two 2PG-bound complex structures differed in the conformation of the bound product (2PG) viz the widely reported canonical conformation and a novel binding pose, which we refer to here as the alternate conformation. Notably, we observed two major differences compared with the forward reaction: the presence of Mg is non-obligatory for the reaction and 2PG assumes an alternate conformation that is likely to facilitate its dissociation from the active site. Molecular dynamics studies and binding free energy calculations further substantiate that the alternate conformation of 2PG causes distortions in both metal ion coordination and hydrogen-bonding interactions, resulting in an increased flexibility of the active-site loops and aiding product release. Taken together, this study presents a probable mechanism involved in PEP to 2PG catalysis that is likely to be mediated by the conformational change of 2PG at the active site. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30988.map.gz emd_30988.map.gz | 117.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30988-v30.xml emd-30988-v30.xml emd-30988.xml emd-30988.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
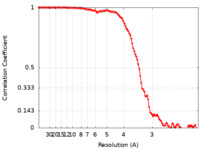
| FSC (解像度算出) |  emd_30988_fsc.xml emd_30988_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30988.png emd_30988.png | 79.8 KB | ||
| Filedesc metadata |  emd-30988.cif.gz emd-30988.cif.gz | 6.3 KB | ||
| その他 |  emd_30988_additional_1.map.gz emd_30988_additional_1.map.gz emd_30988_additional_2.map.gz emd_30988_additional_2.map.gz | 93.6 MB 93.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30988 http://ftp.pdbj.org/pub/emdb/structures/EMD-30988 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30988 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30988 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30988.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30988.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the combined final map with B-factor sharpening | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: The other half-map from the final refinement
| ファイル | emd_30988_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The other half-map from the final refinement | ||||||||||||
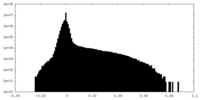
| 投影像・断面図 |
| ||||||||||||
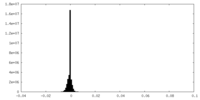
| 密度ヒストグラム |
-追加マップ: One of the half-map from the final refinement
| ファイル | emd_30988_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One of the half-map from the final refinement | ||||||||||||
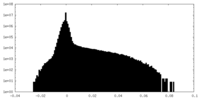
| 投影像・断面図 |
| ||||||||||||
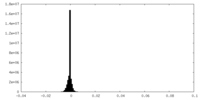
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Octameric Enolase enzyme
| 全体 | 名称: Octameric Enolase enzyme |
|---|---|
| 要素 |
|
-超分子 #1: Octameric Enolase enzyme
| 超分子 | 名称: Octameric Enolase enzyme / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 370 KDa |
-分子 #1: Enolase
| 分子 | 名称: Enolase / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号:  ホスホピルビン酸ヒドラターゼ ホスホピルビン酸ヒドラターゼ |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 45.962355 KDa |
| 組換発現 | 生物種:   Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
| 配列 | 文字列: MHHHHHHMPI IEQVRAREIL DSRGNPTVEV EVALIDGTFA RAAVPSGAST GEHEAVELRD GGDRYGGKGV QKAVQAVLDE IGPAVIGLN ADDQRLVDQA LVDLDGTPDK SRLGGNAILG VSLAVAKAAA DSAELPLFRY VGGPNAHILP VPMMNILNGG A HADTAVDI ...文字列: MHHHHHHMPI IEQVRAREIL DSRGNPTVEV EVALIDGTFA RAAVPSGAST GEHEAVELRD GGDRYGGKGV QKAVQAVLDE IGPAVIGLN ADDQRLVDQA LVDLDGTPDK SRLGGNAILG VSLAVAKAAA DSAELPLFRY VGGPNAHILP VPMMNILNGG A HADTAVDI QEFMVAPIGA PSFVEALRWG AEVYHALKSV LKKEGLSTGL GDEGGFAPDV AGTTAALDLI SRAIESAGLR PG ADVALAL DAAATEFFTD GTGYVFEGTT RTADQMTEFY AGLLGAYPLV SIEDPLSEDD WDGWAALTAS IGDRVQIVGD DIF VTNPER LEEGIERGVA NALLVKVNQI GTLTETLDAV TLAHHGGYRT MISHRSGETE DTMIADLAVA IGSGQIKTGA PARS ERVAK YNQLLRIEEA LGDAARYAGD LAFPRFACET K UniProtKB:  ホスホピルビン酸ヒドラターゼ ホスホピルビン酸ヒドラターゼ |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 130841 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 75000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 27.7 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X