+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30700 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human SARM1 inhibitory state bounded with inhibitor dHNN | |||||||||
 マップデータ マップデータ | Human SARM1 bounded with inhibitor dHNN | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NAD(+) hydrolase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報extrinsic component of synaptic membrane / negative regulation of MyD88-independent toll-like receptor signaling pathway / NADP+ nucleosidase activity / MyD88-independent TLR4 cascade / Toll Like Receptor 3 (TLR3) Cascade / NAD+ catabolic process / NAD+ nucleosidase activity / regulation of synapse pruning / modification of postsynaptic structure / ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase ...extrinsic component of synaptic membrane / negative regulation of MyD88-independent toll-like receptor signaling pathway / NADP+ nucleosidase activity / MyD88-independent TLR4 cascade / Toll Like Receptor 3 (TLR3) Cascade / NAD+ catabolic process / NAD+ nucleosidase activity / regulation of synapse pruning / modification of postsynaptic structure / ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase / protein localization to mitochondrion / NAD+ nucleosidase activity, cyclic ADP-ribose generating / nervous system process / 加水分解酵素; 糖加水分解酵素; N-グリコシル化合物加水分解酵素 / regulation of dendrite morphogenesis / response to axon injury / response to glucose / regulation of neuron apoptotic process / signaling adaptor activity / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / IKK complex recruitment mediated by RIP1 / neuromuscular junction / nervous system development / microtubule / mitochondrial outer membrane / cell differentiation / axon / innate immune response / synapse / dendrite / glutamatergic synapse / cell surface / signal transduction / protein-containing complex / mitochondrion / identical protein binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Cai Y / Zhang H | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Permeant fluorescent probes visualize the activation of SARM1 and uncover an anti-neurodegenerative drug candidate. 著者: Wan Hua Li / Ke Huang / Yang Cai / Qian Wen Wang / Wen Jie Zhu / Yun Nan Hou / Sujing Wang / Sheng Cao / Zhi Ying Zhao / Xu Jie Xie / Yang Du / Chi-Sing Lee / Hon Cheung Lee / Hongmin Zhang / Yong Juan Zhao /  要旨: SARM1 regulates axonal degeneration through its NAD-metabolizing activity and is a drug target for neurodegenerative disorders. We designed and synthesized fluorescent conjugates of styryl derivative ...SARM1 regulates axonal degeneration through its NAD-metabolizing activity and is a drug target for neurodegenerative disorders. We designed and synthesized fluorescent conjugates of styryl derivative with pyridine to serve as substrates of SARM1, which exhibited large red shifts after conversion. With the conjugates, SARM1 activation was visualized in live cells following elevation of endogenous NMN or treatment with a cell-permeant NMN-analog. In neurons, imaging documented mouse SARM1 activation preceded vincristine-induced axonal degeneration by hours. Library screening identified a derivative of nisoldipine (NSDP) as a covalent inhibitor of SARM1 that reacted with the cysteines, especially Cys311 in its ARM domain and blocked its NMN-activation, protecting axons from degeneration. The Cryo-EM structure showed that SARM1 was locked into an inactive conformation by the inhibitor, uncovering a potential neuroprotective mechanism of dihydropyridines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30700.map.gz emd_30700.map.gz | 8.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30700-v30.xml emd-30700-v30.xml emd-30700.xml emd-30700.xml | 12.8 KB 12.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_30700.png emd_30700.png | 168.1 KB | ||
| Filedesc metadata |  emd-30700.cif.gz emd-30700.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30700 http://ftp.pdbj.org/pub/emdb/structures/EMD-30700 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30700 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30700 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30700.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30700.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human SARM1 bounded with inhibitor dHNN | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.076 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human NAD(+) hydrolysis SARM1
| 全体 | 名称: Human NAD(+) hydrolysis SARM1 |
|---|---|
| 要素 |
|
-超分子 #1: Human NAD(+) hydrolysis SARM1
| 超分子 | 名称: Human NAD(+) hydrolysis SARM1 / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: NAD(+) hydrolase SARM1
| 分子 | 名称: NAD(+) hydrolase SARM1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 76.659656 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RLAVPGPDGG GGTGPWWAAG GRGPREVSPG AGTEVQDALE RALPELQQAL SALKQAGGAR AVGAGLAEVF QLVEEAWLLP AVGREVAQG LCDAIRLDGG LDLLLRLLQA PELETRVQAA RLLEQILVAE NRDRVARIGL GVILNLAKER EPVELARSVA G ILEHMFKH ...文字列: RLAVPGPDGG GGTGPWWAAG GRGPREVSPG AGTEVQDALE RALPELQQAL SALKQAGGAR AVGAGLAEVF QLVEEAWLLP AVGREVAQG LCDAIRLDGG LDLLLRLLQA PELETRVQAA RLLEQILVAE NRDRVARIGL GVILNLAKER EPVELARSVA G ILEHMFKH SEETCQRLVA AGGLDAVLYW CRRTDPALLR HCALALGNCA LHGGQAVQRR MVEKRAAEWL FPLAFSKEDE LL RLHACLA VAVLATNKEV EREVERSGTL ALVEPLVASL DPGRFAR(VI3)LV DASDTSQGRG PDDLQRLVPL LDSNRLEAQ CIGAFYLCAE AAIKSLQGKT KVFSDIGAIQ SLKRLVSYST NGTKSALAKR ALRLLGEEVP RPILPSVPSW KEAEVQTWLQ QIGFSKYCE SFREQQVDGD LLLRLTEEEL QTDLGMKSGI TRKRFFRELT ELKTFANYST CDRSNLADWL GSLDPRFRQY T YGLVSCGL DRSLLHRVSE QQLLEDCGIH LGVHRARILT AAREMLHSPL PCTGGKPSGD TPDVFISYRR NSGSQLASLL KV HLQLHGF SVFIDVEKLE AGKFEDKLIQ SVMGARNFVL VLSPGALDKC MQDHDCKDWV HKEIVTALSC GKNIVPIIDG FEW PEPQVL PEDMQAVLTF NGIKWSHEYQ EATIEKIIRF LQGRSSRDSS AGSDTSLEGA APMGPT UniProtKB: NAD(+) hydrolase SARM1 |
-分子 #2: O3-methyl O5-(2-methylpropyl) 2,6-dimethyl-4-[2-(oxidanylamino)ph...
| 分子 | 名称: O3-methyl O5-(2-methylpropyl) 2,6-dimethyl-4-[2-(oxidanylamino)phenyl]pyridine-3,5-dicarboxylate タイプ: ligand / ID: 2 / コピー数: 8 / 式: H8L |
|---|---|
| 分子量 | 理論値: 372.415 Da |
| Chemical component information | 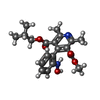 ChemComp-H8L: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.00 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / 詳細: BLOT TIME: 4S BLOT FORCE: -2. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - 画像ごとのフレーム数: 4-36 / 実像数: 2673 / 平均電子線量: 1.28 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): -2.5 µm / 最小 デフォーカス(補正後): -2.0 µm / 倍率(補正後): 130000 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -0.8 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7djt: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)