+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30306 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis for cross-species recognition of COVID-19 virus spike receptor binding domain to bat ACE2 | |||||||||
 マップデータ マップデータ | Cryo-EM structure of bat ACE2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | COVID-19 / receptor binding domain (RBD) / Rhinolophus macrotis / PROTEIN BINDING | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / peptidyl-dipeptidase activity / carboxypeptidase activity / metallopeptidase activity / cilium / apical plasma membrane / proteolysis / extracellular space / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Bat AAV SC2991 (ウイルス) / Bat AAV SC2991 (ウイルス) /  Rhinolophus macrotis (オオミミキクガシラコウモリ) Rhinolophus macrotis (オオミミキクガシラコウモリ) | |||||||||
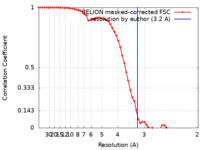
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Liu KF / Wang J | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Cross-species recognition of SARS-CoV-2 to bat ACE2. 著者: Kefang Liu / Shuguang Tan / Sheng Niu / Jia Wang / Lili Wu / Huan Sun / Yanfang Zhang / Xiaoqian Pan / Xiao Qu / Pei Du / Yumin Meng / Yunfei Jia / Qian Chen / Chuxia Deng / Jinghua Yan / ...著者: Kefang Liu / Shuguang Tan / Sheng Niu / Jia Wang / Lili Wu / Huan Sun / Yanfang Zhang / Xiaoqian Pan / Xiao Qu / Pei Du / Yumin Meng / Yunfei Jia / Qian Chen / Chuxia Deng / Jinghua Yan / Hong-Wei Wang / Qihui Wang / Jianxun Qi / George Fu Gao /  要旨: The coronavirus disease 2019 (COVID-19) pandemic caused by severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) has emerged as a major threat to global health. Although varied SARS-CoV-2- ...The coronavirus disease 2019 (COVID-19) pandemic caused by severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) has emerged as a major threat to global health. Although varied SARS-CoV-2-related coronaviruses have been isolated from bats and SARS-CoV-2 may infect bat, the structural basis for SARS-CoV-2 to utilize the human receptor counterpart bat angiotensin-converting enzyme 2 (bACE2) for virus infection remains less understood. Here, we report that the SARS-CoV-2 spike protein receptor binding domain (RBD) could bind to bACE2 from (bACE2-Rm) with substantially lower affinity compared with that to the human ACE2 (hACE2), and its infectivity to host cells expressing bACE2-Rm was confirmed with pseudotyped SARS-CoV-2 virus and SARS-CoV-2 wild virus. The structure of the SARS-CoV-2 RBD with the bACE2-Rm complex was determined, revealing a binding mode similar to that of hACE2. The analysis of binding details between SARS-CoV-2 RBD and bACE2-Rm revealed that the interacting network involving Y41 and E42 of bACE2-Rm showed substantial differences with that to hACE2. Bats have extensive species diversity and the residues for RBD binding in bACE2 receptor varied substantially among different bat species. Notably, the Y41H mutant, which exists in many bats, attenuates the binding capacity of bACE2-Rm, indicating the central roles of Y41 in the interaction network. These findings would benefit our understanding of the potential infection of SARS-CoV-2 in varied species of bats. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30306.map.gz emd_30306.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30306-v30.xml emd-30306-v30.xml emd-30306.xml emd-30306.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30306_fsc.xml emd_30306_fsc.xml | 5.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30306.png emd_30306.png | 56.3 KB | ||
| Filedesc metadata |  emd-30306.cif.gz emd-30306.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30306 http://ftp.pdbj.org/pub/emdb/structures/EMD-30306 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30306 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30306 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30306.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30306.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of bat ACE2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.99375 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
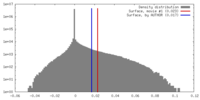
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bat ACE2
| 全体 | 名称: Bat ACE2 |
|---|---|
| 要素 |
|
-超分子 #1: Bat ACE2
| 超分子 | 名称: Bat ACE2 / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Bat AAV SC2991 (ウイルス) Bat AAV SC2991 (ウイルス) |
-分子 #1: Angiotensin-converting enzyme
| 分子 | 名称: Angiotensin-converting enzyme / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Rhinolophus macrotis (オオミミキクガシラコウモリ) Rhinolophus macrotis (オオミミキクガシラコウモリ) |
| 分子量 | 理論値: 69.444312 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: STTEDEAKKF LDKFNSKAED LSYESSLASW DYNTNISDEN VQKMDEAGAK WSAFYEEQSK LAKNYPLEEI QNDTVKRQLQ ILQQSGSPV LSEDKSKRLN SILNAMSTIY STGKVCKPNN PQECLLLEPG LDNIMGTSKD YNERLWAWEG WRAEVGKQLR P LYEEYVVL ...文字列: STTEDEAKKF LDKFNSKAED LSYESSLASW DYNTNISDEN VQKMDEAGAK WSAFYEEQSK LAKNYPLEEI QNDTVKRQLQ ILQQSGSPV LSEDKSKRLN SILNAMSTIY STGKVCKPNN PQECLLLEPG LDNIMGTSKD YNERLWAWEG WRAEVGKQLR P LYEEYVVL KNEMARGYHY EDYGDYWRRD YETEESSGPG YSRDQLMKDV DRIFTEIKPL YEHLHAYVRA KLMDTYPLHI SP TGCLPAH LLGDMWGRFW TNLYPLTVPF GQKPNIDVTD AMLNQGWDAN RIFKEAEKFF VSVSLPKMTE GFWNKSMLTE PGD GRKVVC HPTAWDLGKG DFRIKMCTKV TMEDFLTAHH EMGHIQYDMA YASQPYLLRN GANEGFHEAV GEVMSLSVAT PKHL KTMGL LSPDFREDDE TEINFLLKQA LNIVGTLPFT YMLEKWRWMV FKGEIPKEEW MKKWWEMKRE IVGVVEPVPH DETYC DPAS LFHVANDYSF IRYYTRTIFE FQFHEALCRI AQHNGPLHKC DISNSTDAGK KLHQMLSVGK SQAWTKTLED IVDSRN MDV GPLLRYFKPL YTWLQEQNRK SYVGWNTDWS PYA UniProtKB: Angiotensin-converting enzyme |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)