+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PP5 TPR domain bound to Hsp90:Cdc37:CRaf complex | |||||||||||||||
 マップデータ マップデータ | PP5 TPR domain appears bound to Hsp90:Cdc37:CRaf complex. Map obtained through focused classification with subtraction of PP5 TPR domain, followed by full particle refinement. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
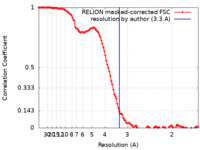
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||||||||
 データ登録者 データ登録者 | Jaime-Garza M / Nowotny CA / Coutandin D / Wang F / Tabios M / Agard DA | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Hsp90 provides a platform for kinase dephosphorylation by PP5. 著者: Maru Jaime-Garza / Carlos A Nowotny / Daniel Coutandin / Feng Wang / Mariano Tabios / David A Agard /  要旨: The Hsp90 molecular chaperone collaborates with the phosphorylated Cdc37 cochaperone for the folding and activation of its many client kinases. As with many kinases, the Hsp90 client kinase CRaf is ...The Hsp90 molecular chaperone collaborates with the phosphorylated Cdc37 cochaperone for the folding and activation of its many client kinases. As with many kinases, the Hsp90 client kinase CRaf is activated by phosphorylation at specific regulatory sites. The cochaperone phosphatase PP5 dephosphorylates CRaf and Cdc37 in an Hsp90-dependent manner. Although dephosphorylating Cdc37 has been proposed as a mechanism for releasing Hsp90-bound kinases, here we show that Hsp90 bound kinases sterically inhibit Cdc37 dephosphorylation indicating kinase release must occur before Cdc37 dephosphorylation. Our cryo-EM structure of PP5 in complex with Hsp90:Cdc37:CRaf reveals how Hsp90 both activates PP5 and scaffolds its association with the bound CRaf to dephosphorylate phosphorylation sites neighboring the kinase domain. Thus, we directly show how Hsp90's role in maintaining protein homeostasis goes beyond folding and activation to include post translationally modifying its client kinases. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29957.map.gz emd_29957.map.gz | 97.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29957-v30.xml emd-29957-v30.xml emd-29957.xml emd-29957.xml | 26 KB 26 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29957_fsc.xml emd_29957_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29957.png emd_29957.png | 75.7 KB | ||
| その他 |  emd_29957_additional_1.map.gz emd_29957_additional_1.map.gz emd_29957_half_map_1.map.gz emd_29957_half_map_1.map.gz emd_29957_half_map_2.map.gz emd_29957_half_map_2.map.gz | 10.9 MB 98.4 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29957 http://ftp.pdbj.org/pub/emdb/structures/EMD-29957 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29957 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29957 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29957.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29957.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PP5 TPR domain appears bound to Hsp90:Cdc37:CRaf complex. Map obtained through focused classification with subtraction of PP5 TPR domain, followed by full particle refinement. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Sharpened postprocessed map.
| ファイル | emd_29957_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened postprocessed map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map. Not yet postprocessed in Relion.
| ファイル | emd_29957_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map. Not yet postprocessed in Relion. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map. Not yet postprocessed in Relion.
| ファイル | emd_29957_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map. Not yet postprocessed in Relion. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hsp90:Cdc37:CRaf complex bound to PP5 TPR domain.
| 全体 | 名称: Hsp90:Cdc37:CRaf complex bound to PP5 TPR domain. |
|---|---|
| 要素 |
|
-超分子 #1: Hsp90:Cdc37:CRaf complex bound to PP5 TPR domain.
| 超分子 | 名称: Hsp90:Cdc37:CRaf complex bound to PP5 TPR domain. / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all 詳細: Yeast purified Hsp90:Cdc37:CRaf complex incubated with e. Coli purified PP5 (mutant H304A). Sample then cross-linked with 0.05% glutaraldehyde for 15m at room temperature, and ran over an ...詳細: Yeast purified Hsp90:Cdc37:CRaf complex incubated with e. Coli purified PP5 (mutant H304A). Sample then cross-linked with 0.05% glutaraldehyde for 15m at room temperature, and ran over an S200 sizing column. Particles were then classified to yield a Hsp90:Cdc37:CRaf complex bound to the PP5 TPR domain. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 57 KDa |
-超分子 #2: Protein Phosphatase 5 (H304A)
| 超分子 | 名称: Protein Phosphatase 5 (H304A) / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: #4 詳細: E coli purified Protein Phosphatase 5, with inactivating H304A mutation. Only PP5 TPR domain can be seen in this complex. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Heat shock protein HSP 90-beta
| 分子 | 名称: Heat shock protein HSP 90-beta / タイプ: protein_or_peptide / ID: 1 詳細: HRV 3C cleavage site, followed by a glycine linker and the Hsp90B sequence. 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGMPEEVHH GEEEVETFAF QAEIAQLMSL IINTFYSNKE IFLRELISNA SDALDKIRYE SLTDPSKLDS GKELKIDIIP NPQERTLTLV DTGIGMTKAD LINNLGTIAK SGTKAFMEAL QAGADISMIG QFGVGFYSAY LVAEKVVVIT KHNDDEQYAW ESSAGGSFTV ...文字列: GPGMPEEVHH GEEEVETFAF QAEIAQLMSL IINTFYSNKE IFLRELISNA SDALDKIRYE SLTDPSKLDS GKELKIDIIP NPQERTLTLV DTGIGMTKAD LINNLGTIAK SGTKAFMEAL QAGADISMIG QFGVGFYSAY LVAEKVVVIT KHNDDEQYAW ESSAGGSFTV RADHGEPIGR GTKVILHLKE DQTEYLEERR VKEVVKKHSQ FIGYPITLYL EKEREKEISD DEAEEEKGEK EEEDKDDEEK PKIEDVGSDE EDDSGKDKKK KTKKIKEKYI DQEELNKTKP IWTRNPDDIT QEEYGEFYKS LTNDWEDHLA VKHFSVEGQL EFRALLFIPR RAPFDLFENK KKKNNIKLYV RRVFIMDSCD ELIPEYLNFI RGVVDSEDLP LNISREMLQQ SKILKVIRKN IVKKCLELFS ELAEDKENYK KFYEAFSKNL KLGIHEDSTN RRRLSELLRY HTSQSGDEMT SLSEYVSRMK ETQKSIYYIT GESKEQVANS AFVERVRKRG FEVVYMTEPI DEYCVQQLKE FDGKSLVSVT KEGLELPEDE EEKKKMEESK AKFENLCKLM KEILDKKVEK VTISNRLVSS PCCIVTSTYG WTANMERIMK AQALRDNSTM GYMMAKKHLE INPDHPIVET LRQKAEADKN DKAVKDLVVL LFETALLSSG FSLEDPQTHS NRIYRMIKLG LGIDEDEVAA EEPNAAVPDE IPPLEGDEDA SRMEEVD |
-分子 #2: Hsp90 co-chaperone Cdc37
| 分子 | 名称: Hsp90 co-chaperone Cdc37 / タイプ: protein_or_peptide / ID: 2 詳細: Cdc37 sequence followed by HRV 3C cleavage site (LEVLFQ) 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVDYSVWDHI EVSDDEDETH PNIDTASLFR WRHQARVERM EQFQKEKEEL DRGCRECKRK VAECQRKLKE LEVAEGGKAE LERLQAEAQQ LRKEERSWEQ KLEEMRKKEK SMPWNVDTLS KDGFSKSMVN TKPEKTEEDS EEVREQKHKT FVEKYEKQIK HFGMLRRWDD ...文字列: MVDYSVWDHI EVSDDEDETH PNIDTASLFR WRHQARVERM EQFQKEKEEL DRGCRECKRK VAECQRKLKE LEVAEGGKAE LERLQAEAQQ LRKEERSWEQ KLEEMRKKEK SMPWNVDTLS KDGFSKSMVN TKPEKTEEDS EEVREQKHKT FVEKYEKQIK HFGMLRRWDD SQKYLSDNVH LVCEETANYL VIWCIDLEVE EKCALMEQVA HQTIVMQFIL ELAKSLKVDP RACFRQFFTK IKTADRQYME GFNDELEAFK ERVRGRAKLR IEKAMKEYEE EERKKRLGPG GLDPVEVYES LPEELQKCFD VKDVQMLQDA ISKMDPTDAK YHMQRCIDSG LWVPNSKASE AKEGEEAGPG DPLLEAVPKT GDEKDVSVLE VLFQ |
-分子 #3: RAF proto-oncogene serine/threonine-protein kinase
| 分子 | 名称: RAF proto-oncogene serine/threonine-protein kinase / タイプ: protein_or_peptide / ID: 3 詳細: Truncated CRaf/RAF-1 kinase domain followed by Strep Tag II sequence (WSHPQFEK) and HRV 3C cleavage site (LEVLFQ) 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GGRDSSYYWE IEASEVMLST RIGSGSFGTV YKGKWHGDVA VKILKVVDPT PEQFQAFRNE VAVLRKTRHV NILLFMGYMT KDNLAIVTQW CEGSSLYKHL HVQETKFQMF QLIDIARQTA QGMDYLHAKN IIHRDMKSNN IFLHEGLTVK IGDFGLATVK SRWSGSQQVE ...文字列: GGRDSSYYWE IEASEVMLST RIGSGSFGTV YKGKWHGDVA VKILKVVDPT PEQFQAFRNE VAVLRKTRHV NILLFMGYMT KDNLAIVTQW CEGSSLYKHL HVQETKFQMF QLIDIARQTA QGMDYLHAKN IIHRDMKSNN IFLHEGLTVK IGDFGLATVK SRWSGSQQVE QPTGSVLWMA PEVIRMQDNN PFSFQSDVYS YGIVLYELMT GELPYSHINN RDQIIFMVGR GYASPDLSKL YKNCPKAMKR LVADCVKKVK EERPLFPQIL SSIELLQHSL PKINRLPESG WSHPQFEKLE VLFQ |
-分子 #4: Serine/threonine-protein phosphatase 5
| 分子 | 名称: Serine/threonine-protein phosphatase 5 / タイプ: protein_or_peptide / ID: 4 詳細: HRV 3C cleavage site followed by GS linker and PP5 sequence. 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGSMAMAEG ERTECAEPPR DEPPADGALK RAEELKTQAN DYFKAKDYEN AIKFYSQAIE LNPSNAIYYG NRSLAYLRTE CYGYALGDAT RAIELDKKYI KGYYRRAASN MALGKFRAAL RDYETVVKVK PHDKDAKMKY QECNKIVKQK AFERAIAGDE HKRSVVDSLD ...文字列: GPGSMAMAEG ERTECAEPPR DEPPADGALK RAEELKTQAN DYFKAKDYEN AIKFYSQAIE LNPSNAIYYG NRSLAYLRTE CYGYALGDAT RAIELDKKYI KGYYRRAASN MALGKFRAAL RDYETVVKVK PHDKDAKMKY QECNKIVKQK AFERAIAGDE HKRSVVDSLD IESMTIEDEY SGPKLEDGKV TISFMKELMQ WYKDQKKLHR KCAYQILVQV KEVLSKLSTL VETTLKETEK ITVCGDTHGQ FYDLLNIFEL NGLPSETNPY IFNGDFVDRG SFSVEVILTL FGFKLLYPDH FHLLRGNAET DNMNQIYGFE GEVKAKYTAQ MYELFSEVFE WLPLAQCING KVLIMHGGLF SEDGVTLDDI RKIERNRQPP DSGPMCDLLW SDPQPQNGRS ISKRGVSCQF GPDVTKAFLE ENNLDYIIRS HEVKAEGYEV AHGGRCVTVF SAPNYCDQMG NKASYIHLQG SDLRPQFHQF TAVPHPNVKP MAYANTLLQL GMM |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 1.0 nm | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV 詳細: Sample Vol: 3UL Temp: 10C Humidity: 100% WAIT TIME: 30S BLOT TIME: 3S BLOT FORCE: -2. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4160 / 平均電子線量: 69.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)