+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a group II intron immediately before branching | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | group II intron / splicing / branching / maturase / SPLICING-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-directed DNA polymerase activity / endonuclease activity / nucleic acid binding / zinc ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Thermosynechococcus vestitus (バクテリア) Thermosynechococcus vestitus (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Haack DB / Rudolfs BG / Zhang C / Lyumkis D / Toor N | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural basis of branching during RNA splicing. 著者: Daniel B Haack / Boris Rudolfs / Cheng Zhang / Dmitry Lyumkis / Navtej Toor /  要旨: Branching is a critical step in RNA splicing that is essential for 5' splice site selection. Recent spliceosome structures have led to competing models for the recognition of the invariant adenosine ...Branching is a critical step in RNA splicing that is essential for 5' splice site selection. Recent spliceosome structures have led to competing models for the recognition of the invariant adenosine at the branch point. However, there are no structures of any splicing complex with the adenosine nucleophile docked in the active site and positioned to attack the 5' splice site. Thus we lack a mechanistic understanding of adenosine selection and splice site recognition during RNA splicing. Here we present a cryo-electron microscopy structure of a group II intron that reveals that active site dynamics are coupled to the formation of a base triple within the branch-site helix that positions the 2'-OH of the adenosine for nucleophilic attack on the 5' scissile phosphate. This structure, complemented with biochemistry and comparative analyses to splicing complexes, supports a base triple model of adenosine recognition for branching within group II introns and the evolutionarily related spliceosome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29279.map.gz emd_29279.map.gz | 136.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29279-v30.xml emd-29279-v30.xml emd-29279.xml emd-29279.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29279.png emd_29279.png | 58.5 KB | ||
| Filedesc metadata |  emd-29279.cif.gz emd-29279.cif.gz | 6.1 KB | ||
| その他 |  emd_29279_half_map_1.map.gz emd_29279_half_map_1.map.gz emd_29279_half_map_2.map.gz emd_29279_half_map_2.map.gz | 134.3 MB 134.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29279 http://ftp.pdbj.org/pub/emdb/structures/EMD-29279 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29279 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29279 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29279_validation.pdf.gz emd_29279_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29279_full_validation.pdf.gz emd_29279_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_29279_validation.xml.gz emd_29279_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_29279_validation.cif.gz emd_29279_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29279 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29279 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29279 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29279 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fliMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29279.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29279.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||
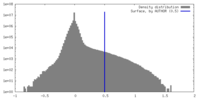
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_29279_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
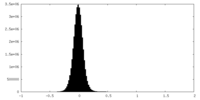
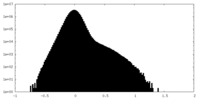
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29279_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
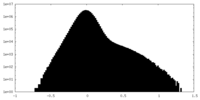
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Group IIB intron in complex with its maturase protein
| 全体 | 名称: Group IIB intron in complex with its maturase protein |
|---|---|
| 要素 |
|
-超分子 #1: Group IIB intron in complex with its maturase protein
| 超分子 | 名称: Group IIB intron in complex with its maturase protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Thermosynechococcus vestitus (バクテリア) Thermosynechococcus vestitus (バクテリア) |
-分子 #1: Group II Intron
| 分子 | 名称: Group II Intron / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Thermosynechococcus vestitus (バクテリア) Thermosynechococcus vestitus (バクテリア) |
| 分子量 | 理論値: 289.257938 KDa |
| 配列 | 文字列: CCCAGGGUUG GCCGAGCGUU GCGACGCGAA AGCUAGCCAG AUGAUUGUCC CACUAGCCCA ACAAGCUAGA ACGGGACCGG UUGUUCCCC CAACCGUAGC CUAGGGAGGC AUGCGUGACU GGUAACGGUC AGGUGUGAAG CCCUCCCGAC AAUGUAGCCC G AACCGCAA ...文字列: CCCAGGGUUG GCCGAGCGUU GCGACGCGAA AGCUAGCCAG AUGAUUGUCC CACUAGCCCA ACAAGCUAGA ACGGGACCGG UUGUUCCCC CAACCGUAGC CUAGGGAGGC AUGCGUGACU GGUAACGGUC AGGUGUGAAG CCCUCCCGAC AAUGUAGCCC G AACCGCAA GGUUGAAGCU GAAUCCGUGA GGAGGAAGCA ACUUCACCAG UGUCAGGUGA UAGGGAACUA GGCUUGAGGG UA UGGUGAG CACAUGCGAA GUGAUGUCAG AAGCCUCGUC ACAGACCAAC AGGCCAAAGA CACUGAUAGG CCUGAGCCAA AAC GGCAAA UGGAUAGGCU ACAUCGCUCG CUCGUCGGUG UACGGGGACG UCAAUCCAUC GGGGCACAGU CACCACCUAA CCCC UCGUG UCAUCUGGUU GGAACGCGGU AAGCCCGUAU CCUCGCCUUG AACACUCAAG GCAGGCAAAC CUUCGGGAAU GCUGA UGGG GGUGCGGGUA UGGGAUGCAG GAGAAAGCGA AUGCCGGUCU GUAAUGGACC GGAUAGGGGU UGAGGAGACA AUCCAA CAU CACCCCGCCC GAAAGGGAGC AGACUUCCUG CUGGUCUCUC UUUGCGAGAU AGCCUGUAGA ACCUCUUGAA UGGAGAC AA GGCAAAUGGC AGUGGAACAA ACCACUGGUG CGGUCACCAA CCAAACGGAA ACAAGCUGGC ACAGCAUAGA CUGGGCCA A AGCCAACCGU GAGGUAAAGA GGCUGCAAGU GCGUAUCGCA AAGGCGUUCG CGCCGGUUCC UCUUGAAAGA GGGGCUUUG AGAGGCCUGA GCCGGAUGUG GGGAAACUCA CAAGUCCGGU UCUUAGGGGG CGGGGAUGGC AUUCGUGCCU CCCUGCUACC CGGCGAUGA GGCA |
-分子 #2: Maturase reverse transcriptase
| 分子 | 名称: Maturase reverse transcriptase / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermosynechococcus vestitus (バクテリア) Thermosynechococcus vestitus (バクテリア) |
| 分子量 | 理論値: 65.065121 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: METRQMAVEQ TTGAVTNQTE TSWHSIDWAK ANREVKRLQV RIAKAVKEGR WGKVKALQWL LTHSFYGKAL AVKRVTDNSG SKTPGVDGI TWSTQEQKAQ AIKSLRRRGY KPQPLRRVYI PKASGKQRPL GIPTTKDRAM QALYALALEP VAETTADRNS Y GFRQGRCT ...文字列: METRQMAVEQ TTGAVTNQTE TSWHSIDWAK ANREVKRLQV RIAKAVKEGR WGKVKALQWL LTHSFYGKAL AVKRVTDNSG SKTPGVDGI TWSTQEQKAQ AIKSLRRRGY KPQPLRRVYI PKASGKQRPL GIPTTKDRAM QALYALALEP VAETTADRNS Y GFRQGRCT ADAAGQCFTV LGRSDCAKYI LDADITGCFD NISHEWLLDN IPLDKEVLRK WLKSGFVWKQ QLFPTHAGTP QG GVISPML ANMTLDGMEE LLKKHLRKQK VNLIRYADDF VVTGESKETL EKVTTVIQEF LKERGLTLSE EKTKVVHIEE GFD FLGWNI RKYGEKLLIK PAKKNIKAFH KKIRDALKEL RTATQEAVID TLNPIIKGWA NYHRNQVSKR IFNRADDNIW HKLW RWAKR RHPNKPARWT KNKYFIKIGN RHWVFGTWKK DKEGRLRSRY LIKAGDTRIQ RHVKIKADAN PFLPEWAEYF EERKK LKEA PAQYRRIRRE LWKKQGGICP VCGGEIEQDM LTEIHHILPK HKGGSDDLDN LVLIHANCHK QVHSRDGQHS RFLLKE GL UniProtKB: Maturase reverse transcriptase |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 31.6 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 38881 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)