+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate | |||||||||
 マップデータ マップデータ | Cryptococcus neoformans trehalose-6-phosphate synthase (Tps1) homotetramer bound to UDP and G6P | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Glycosyltransferase / Complex / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alpha,alpha-trehalose-phosphate synthase complex (UDP-forming) / alpha,alpha-trehalose-phosphate synthase (UDP-forming) / trehalose-phosphatase activity / alpha,alpha-trehalose-phosphate synthase (UDP-forming) activity / trehalose biosynthetic process / cellular response to heat / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Cryptococcus neoformans var. grubii H99 (菌類) Cryptococcus neoformans var. grubii H99 (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Washington EJ / Brennan RG | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2024 ジャーナル: bioRxiv / 年: 2024タイトル: Structures of trehalose-6-phosphate synthase, Tps1, from the fungal pathogen : a target for novel antifungals. 著者: Erica J Washington / Ye Zhou / Allen L Hsu / Matthew Petrovich / Jennifer L Tenor / Dena L Toffaletti / Ziqiang Guan / John R Perfect / Mario J Borgnia / Alberto Bartesaghi / Richard G Brennan 要旨: Invasive fungal diseases are a major threat to human health, resulting in more than 1.5 million annual deaths worldwide. The arsenal of antifungal therapeutics remains limited and is in dire need of ...Invasive fungal diseases are a major threat to human health, resulting in more than 1.5 million annual deaths worldwide. The arsenal of antifungal therapeutics remains limited and is in dire need of novel drugs that target additional biosynthetic pathways that are absent from humans. One such pathway involves the biosynthesis of trehalose. Trehalose is a disaccharide that is required for pathogenic fungi to survive in their human hosts. In the first step of trehalose biosynthesis, trehalose-6-phosphate synthase (Tps1) converts UDP-glucose and glucose-6-phosphate to trehalose-6-phosphate. Here, we report the structures of full-length Tps1 (CnTps1) in unliganded form and in complex with uridine diphosphate and glucose-6-phosphate. Comparison of these two structures reveals significant movement towards the catalytic pocket by the N-terminus upon ligand binding and identifies residues required for substrate-binding, as well as residues that stabilize the tetramer. Intriguingly, an intrinsically disordered domain (IDD), which is conserved amongst Cryptococcal species and closely related Basidiomycetes, extends from each subunit of the tetramer into the "solvent" but is not visible in density maps. We determined that the IDD is not required for Tps1-dependent thermotolerance and osmotic stress survival. Studies with UDP-galactose highlight the exquisite substrate specificity of CnTps1. , these studies expand our knowledge of trehalose biosynthesis in and highlight the potential of developing antifungal therapeutics that disrupt the synthesis of this disaccharide or the formation of a functional tetramer and the use of cryo-EM in the structural characterization of CnTps1-ligand/drug complexes. SIGNIFICANCE STATEMENT: Fungal infections are responsible for over a million deaths worldwide each year. Biosynthesis of a disaccharide, trehalose, is required for multiple pathogenic fungi to ...SIGNIFICANCE STATEMENT: Fungal infections are responsible for over a million deaths worldwide each year. Biosynthesis of a disaccharide, trehalose, is required for multiple pathogenic fungi to transition from the environment to the human host. Enzymes in the trehalose biosynthesis pathway are absent in humans and, therefore, are potentially significant targets for novel antifungal therapeutics. One enzyme in the trehalose biosynthesis is trehalose-6-phosphate synthase (Tps1). Here, we describe the cryo-electron microscopy structures of the CnTps1 homo-tetramer in the unliganded form and in complex with a substrate and a product. These structures and subsequent biochemical analysis reveal key details of substrate-binding residues and substrate specificity. These structures should facilitate structure-guided design of inhibitors against CnTps1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29172.map.gz emd_29172.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29172-v30.xml emd-29172-v30.xml emd-29172.xml emd-29172.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29172_fsc.xml emd_29172_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29172.png emd_29172.png | 118.9 KB | ||
| Filedesc metadata |  emd-29172.cif.gz emd-29172.cif.gz | 6 KB | ||
| その他 |  emd_29172_half_map_1.map.gz emd_29172_half_map_1.map.gz emd_29172_half_map_2.map.gz emd_29172_half_map_2.map.gz | 59 MB 59 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29172 http://ftp.pdbj.org/pub/emdb/structures/EMD-29172 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29172 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29172 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29172_validation.pdf.gz emd_29172_validation.pdf.gz | 826.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29172_full_validation.pdf.gz emd_29172_full_validation.pdf.gz | 826.3 KB | 表示 | |
| XML形式データ |  emd_29172_validation.xml.gz emd_29172_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_29172_validation.cif.gz emd_29172_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29172 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29172 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29172 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29172 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fhwMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29172.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29172.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryptococcus neoformans trehalose-6-phosphate synthase (Tps1) homotetramer bound to UDP and G6P | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Cryptococcus neoformans trehalose-6-phosphate synthase (Tps1) homotetramer bound to...
| ファイル | emd_29172_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryptococcus neoformans trehalose-6-phosphate synthase (Tps1) homotetramer bound to UDP and G6P Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryptococcus neoformans trehalose-6-phosphate synthase (Tps1) homotetramer bound to...
| ファイル | emd_29172_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryptococcus neoformans trehalose-6-phosphate synthase (Tps1) homotetramer bound to UDP and G6P Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphat...
| 全体 | 名称: Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphat...
| 超分子 | 名称: Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Cryptococcus neoformans var. grubii H99 (菌類) Cryptococcus neoformans var. grubii H99 (菌類) |
| 分子量 | 理論値: 307 KDa |
-分子 #1: Alpha,alpha-trehalose-phosphate synthase (UDP-forming)
| 分子 | 名称: Alpha,alpha-trehalose-phosphate synthase (UDP-forming) タイプ: protein_or_peptide / ID: 1 / 詳細: UDP G6P / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cryptococcus neoformans var. grubii H99 (菌類) Cryptococcus neoformans var. grubii H99 (菌類) |
| 分子量 | 理論値: 76.8155 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHSSG VDLGTENLYF QSNAMTTMSN DIPNSPTSTS FTGTFSPAAT AANTAANART SDAPSPTTSS SGPKLETSKE QRLIVVSNR LPVTISKDDN GEYHFKMSSG GLVSALSGCK KTMSFTWIGW PGKDIPMQDR ETVNRRLLDE YNCYPVYLSD E LADSHYNG ...文字列: MHHHHHHSSG VDLGTENLYF QSNAMTTMSN DIPNSPTSTS FTGTFSPAAT AANTAANART SDAPSPTTSS SGPKLETSKE QRLIVVSNR LPVTISKDDN GEYHFKMSSG GLVSALSGCK KTMSFTWIGW PGKDIPMQDR ETVNRRLLDE YNCYPVYLSD E LADSHYNG FSNSILWPLF HYHPGEMNFD AAHWLAYREA NMRFADVVSS LVQAGDMVWV QDYHLMLLPM LLRSMITGES AQ GEMVRQE LGRVKEGVDD TVVKEVLKMG PGVAQAEDEG VEMLDDVEEE GGEMDVKSSP KRPHYARGMS TFQKQELVAK EKG KEGIRI GFFLHTPFPS SEIYRILPVR REILLGVLQC DLIGFHTYDY ARHFLSSCTR ILGLETQPNG IEFDGRYCQV GTFP IGIDP NQFIEGLQKE SIVKRLRSLE ARFEGVKVII GVDRLDYIKG IPQKLQALET FLTQHPEWIG KVVLVQLAIP SRQDV EEYQ DLRACVNELV GRINGRFGTV ESVPIHYMHK SVPFEELTAM YALADACLVT STRDGMNLVA YEYISSQAER HGSMIL SEF AGAAQSFNGS LLINPWDVQS TADAINQALT LSPQQRKTNW QKLFNYVSKY TAEAWGVSFV NELNRLSGQR PSGPTGL AG RRKSGSLSRT SSKASIQRRK SSQSGIVTGL GAAAGAAVNW AQAQVQGGSQ T UniProtKB: alpha,alpha-trehalose-phosphate synthase (UDP-forming) |
-分子 #2: URIDINE-5'-DIPHOSPHATE
| 分子 | 名称: URIDINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: UDP |
|---|---|
| 分子量 | 理論値: 404.161 Da |
| Chemical component information |  ChemComp-UDP: |
-分子 #3: 6-O-phosphono-alpha-D-glucopyranose
| 分子 | 名称: 6-O-phosphono-alpha-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 4 / 式: G6P |
|---|---|
| 分子量 | 理論値: 260.136 Da |
| Chemical component information | 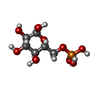 ChemComp-G6P: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.75 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)